RDKit | 基于Murcko骨架聚类化合物库
化合物多样性评估
一种方法是使用合适的指纹技术将化合物矢量化并评估他们之间的距离。这种方法经常被使用,但是对于人类很难直观地理解化合物之间的距离。
基于Murcko骨架,分子大致聚集在化合物骨架中,并且每个簇中的顶部化合物都作为候选化合物。此方法非常符合人类的直觉,因此可以预期人将通过查看候选化合物列表来自动缩小化合物的范围。
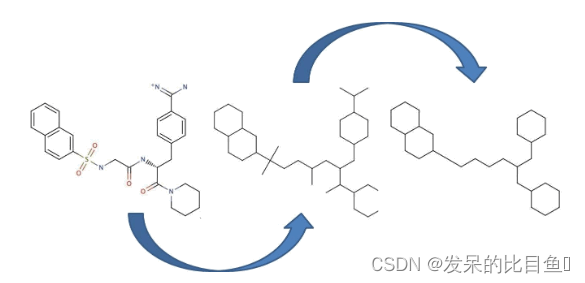
Murcko骨架生成
基于Murcko骨架聚类化合物库
导入库
import numpy as np
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Scaffolds import MurckoScaffold
from rdkit.Chem.Draw import IPythonConsole
载入数据
sdfloader = Chem.SDMolSupplier("Enamine_Premium_processed.sdf") ### load from multisdfile
mol_list = [ mol for mol in [sdfloader[i] for i in range(len(sdfloader))] if mol is not None]
smi_scaffolds = [ MurckoScaffold.MurckoScaffoldSmiles(mol=mol, includeChirality=False) for mol in mol_list]
mol_scaffolds = [Chem.MolFromSmiles(smi_scaffold) for smi_scaffold in smi_scaffolds]
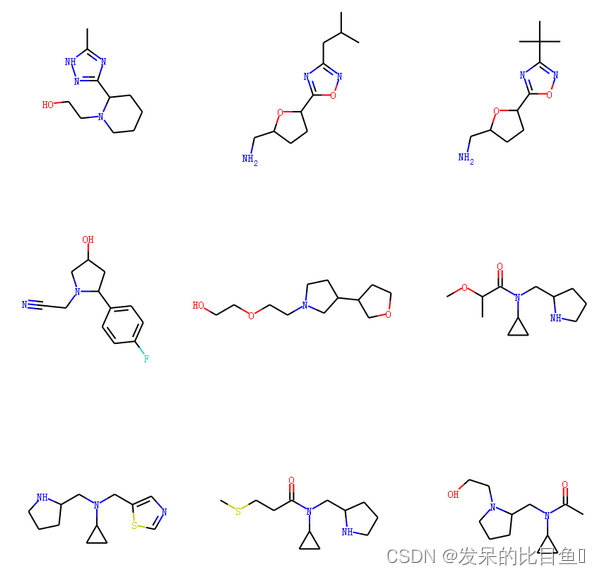
可视化原始化合物
Draw.MolsToGridImage(mol_list[:9], molsPerRow=3, subImgSize=(300,300))
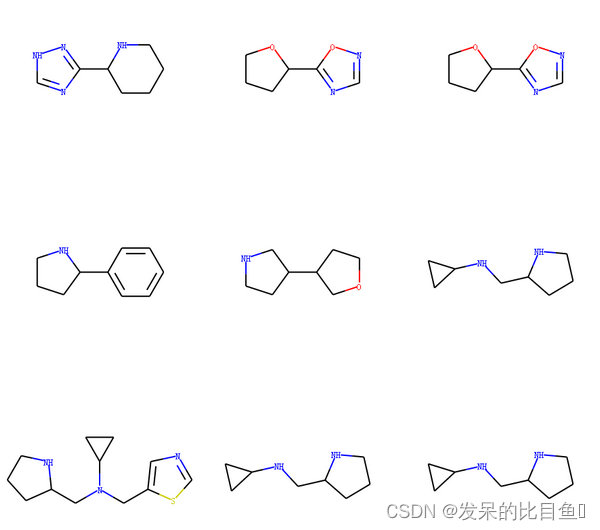
可视化骨架化合物
Draw.MolsToGridImage(mol_scaffolds[:9], molsPerRow=3, subImgSize=(300,300))
基于Murcko骨架聚类
scaffolds = {}
clusters_list =[]
idx = 1
for mol in mol_list:
scaffold_smi = MurckoScaffold.MurckoScaffoldSmiles(mol=mol, includeChirality=False)
if scaffold_smi not in scaffolds.keys():
scaffolds[scaffold_smi] = idx
idx+=1
cluster_id = scaffolds[scaffold_smi]
clusters_list.append(cluster_id)
print("Num of dataset:",len(mol_list))
Num of dataset: 128816
print("Num of Murcko scaffolds in dataset:",len(scaffolds.keys()))
聚类11个簇,查看其化合物
clusters_list = np.array(clusters_list)
idx_c15 = np.where(clusters_list==11)[0]
mol_list_c15 = [ mol_list[i] for i in idx_c15]
Draw.MolsToGridImage(mol_list_c15, molsPerRow=3, subImgSize=(300,300))
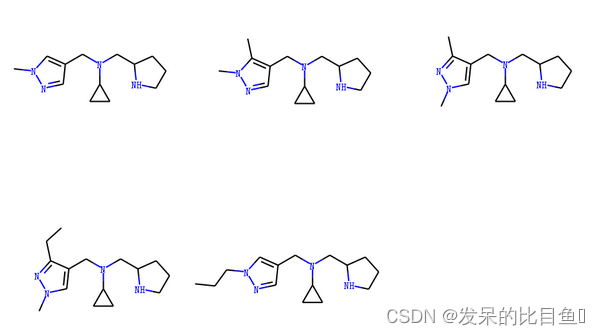






















 2135
2135











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








