一、前言
网站地址:
String:https://cn.string-db.org/TCMSP:https://old.tcmsp-e.com/tcmsp.phpHerb:http://herb.ac.cn/SwissADME:http://www.swissadme.ch/index.php
本文复现的步骤:
- 筛选药物成分,对应步骤【二】和【三】
- 获得成分相关信息,对应步骤【四】
二、TCMSP筛选药物活性成分
论文原文步骤
TCMSP筛选大黄的活性成分。以“大黄”为关键词,筛选标准包括口服生物利用度(OB)≥ 20% 和药物相似性(DL)≥ 0.18,以识别具有潜在药理活性的成分。
复现步骤
网站首页
首先进入TCMSP网站如下:

网站搜索
直接输入大黄英文名Dahuang之后点击搜索,出现如下页面,点击拉丁名称后会跳转

搜索结果初筛
可以看到这里出现了搜索结果列表,我们只关注两列:OB(生物利用度)和DL(药物相似性)

这里每列点击后其实都可以切换顺序/倒序排序的,具体可见点击列明后的显示的是上箭头还是下箭头。
但是当列过窄时可能和筛选图标重叠,我们可以手动按住列的分割线将列拉宽点。
然后点击筛选图标,按照论文原文的生物利用度(OB)≥ 20% 和药物相似性(DL)≥ 0.18进行筛选。
筛选后就从一开始的92条数据变为了29条数据(因为数据库一直在变动,所以筛选后不是29条也问题不大~)


下载数据
随后我们需要下载数据,在这里你可以选择以下任意方式之一:
手动粘贴
因为数量不多,所以可以手动粘贴到txt文件或者xlsx文件中,如下:

代码爬虫
本人写了个爬虫脚本,如下。
使用前提是下载你所使用的浏览器对应的`driver`,譬如你用的是`chrome`,那么要下`chromedriver`。
在这里我提供了`chromedriver`在本文章的资源绑定里面。
ps:不保障每人都能使用成功该脚本,受各种因素影响捏。
本人纯纯为爱发电,不提供代码的售后服务,有问题可在评论区留言,看到即回。
from selenium import webdriver
from selenium.webdriver.common.by import By
from selenium.webdriver.support.ui import WebDriverWait
from selenium.webdriver.support import expected_conditions as EC
from bs4 import BeautifulSoup
import pandas as pd
options = webdriver.ChromeOptions()
driver = webdriver.Chrome(options=options)
def get_related_targets(url):
# 打开目标网页
driver.get(url)
# 等待页面加载完成
WebDriverWait(driver, 10).until(
EC.presence_of_element_located((By.XPATH, "//a[contains(text(), 'Related Targets')]"))
)
related_targets_tab = WebDriverWait(driver, 10).until(
EC.element_to_be_clickable((By.XPATH, "//a[contains(text(), 'Related Targets')]"))
)
related_targets_tab.click()
# table = driver.find_element((By.XPATH, '//*[@id="grid2"]/div[2]/table'))
WebDriverWait(driver, 10).until(
EC.presence_of_element_located((By.XPATH, '//*[@id="grid2"]/div[2]/table'))
)
# 初始化DataFrame来存储所有页面的表格数据
all_pages_data = pd.DataFrame()
# 循环遍历所有分页
while True:
# 获取当前页面的HTML
html = driver.page_source
soup = BeautifulSoup(html, 'html.parser')
# 使用Selenium获取表格数据
table = driver.find_element(By.CSS_SELECTOR, '#grid2 > div.k-grid-content > table')
rows = table.find_elements(By.TAG_NAME, 'tr')
data = []
for row in rows:
cols = row.find_elements(By.TAG_NAME, 'td') # 或者 'th' 如果是表头
cols_text = [ele.text.strip() for ele in cols]
data.append(cols_text)
# 将当前页面的数据添加到DataFrame
# 跳过表头,直接使用数据行创建DataFrame
df = pd.DataFrame(data)
all_pages_data = pd.concat([all_pages_data, df], ignore_index=True)
# 尝试点击下一页按钮,如果找不到则跳出循环
try:
# 等待“下一页”按钮可点击
next_page_btn = WebDriverWait(driver, 10).until(
EC.element_to_be_clickable(
(By.CSS_SELECTOR, '#grid2 > div.k-pager-wrap.k-grid-pager.k-widget > a:nth-child(4)'))
)
if "k-state-disabled" not in next_page_btn.get_attribute("class"):
# 点击按钮
next_page_btn.click()
WebDriverWait(driver, 2).until(
EC.presence_of_element_located((By.CSS_SELECTOR, '#grid2 > div.k-grid-content > table'))
)
else:
print("已到达最后一页")
break
except Exception as e:
print("发生错误:", e)
break
# 关闭WebDriver
driver.quit()
# # 打印所有页面的数据
# print(all_pages_data)
# 如果需要,可以将数据保存到文件
all_pages_data.to_csv('targets.txt', index=False, header=False)
if __name__ == '__main__':
url = "https://old.tcmsp-e.com/tcmspsearch.php?qr=Coptidis%20Rhizoma&qsr=herb_en_name&token=5d5532b4c3e1a1d1aa9817e3b8d8b89a"
# 需要换成初筛后的网址
get_related_targets(url)
开通会员
该网站貌似有会员服务,会提供另一个版本的网站,可选hhh
三、Herb筛选药物活性成分
此步骤仅作演示,不涉及论文复现
有时候TCMSP查不到我们的药物或者数据量太少时(因为TCMSP上收录的动物药材较少)。这时我们可以去Herb数据库进行查找。
网站首页和搜索
网站首页如下:

可以看到结果还是比较全的,在这我们点击HERB005782,直接对应的吴茱萸。

可以看到,它整合了SymMap、TCMID、TCMSP、TCM-ID四个数据库的结果。

在这我们可以直接下载成分,靶点,甚至相关疾病等数据。

数据处理
合并整个HERB数据库
我们选择成分下载,得到命名为herb_ingredient时间戳.xlsx的文件,打开如下:

第一个是herb数据库的id,第二个是成分名。可以看到它没有列出成分的OB和DL等。
接下来我们可以去到导航栏的下载页面,将其中的ingrediant info下载下来,这就是整个herb数据库的成分信息,然后通过herb id或者其他方法合并两个表格即可(可以见下一步)
ingrediant info下载后可能是HERB_ingrediant_info.txt格式,直接将后缀改为xlsx等即可。

excel如何通过某一公共列合并两个表格匹配的行?
在excel中可以使用“vlook函数”或者“数据透视表”或者“合并查询”的功能实现。前两个是版本较旧的excel的功能,最后一个是最新版excel的功能。
如果还是觉得较为复杂,可以使用以下方法。
我们记整个HERB的成分表为表A,单独某个中药的成分表为表B,新建一个表C,我一般直接命名为HERB_中药名.xlsx。
然后将表B的herb_id这一列(不包含表头)复制到表A的最下方空白行。
然后选中第一列设置高亮重复项,再选择筛选 > 颜色筛选 > (选择橙色)
现在上方就是我们的匹配行了,将其复制到表C
如上方法缺点就是最后一步复制时比较慢。
筛选OB值
合并后我们看到表格如下:有Smile号、分子量、OB值、CAS、在其他数据库的id等信息,但是缺少DL值。

我们首先根据OB值进行筛选,新建一个表格"HERB_lack_DL.xlsx",将符合OB≥30的数据放进去。
筛选DL值
然后再根据DL值筛选。我们将刚刚"HERB_lack_DL.xlsx"中数据按照SMILES号降序排列,然后N/A值或者Not Available值就排在最前面了。
根据其有值的CAS号/TCMSP id/PubChem id/其他数据库id或者直接是名称等,尽量查询并补全其SMILES号,还可以顺便查到其DL值。
四、SwissADME二次筛选药物活性成分
论文原文步骤
使用SwissADME数据库进行了二次筛选,选择了具有高胃肠道吸收(GI absorption)并符合Lipinski、Ghose、Veber、Egan和Muegge等药物相似性标准的成分。经过这一筛选过程,最终确定了具有已知靶点的大黄七个活性成分:EUPATIN, Emodinanthrone, Physcion-9-O-beta-D-glucopyranoside_qt, 大黄酸, Toralactone, 大黄素, 和 Physcion。
复现步骤
批量获得SMILES序列号
可以见我的另一篇博客:http://t.csdnimg.cn/rADVb
筛选
拿到SMILES序列号后去SwissADME中进行转化,如下,点击Run

然后根据原文的"选择了具有高胃肠道吸收(GI absorption)",就是看图1处是否为High。
原文的“符合Lipinski、Ghose、Veber、Egan和Muegge等药物相似性标准”,就是看图2框中的地方是否有两个及以上的yes。

四、查询成分相关
TCMSP获得成分的mol2格式文件
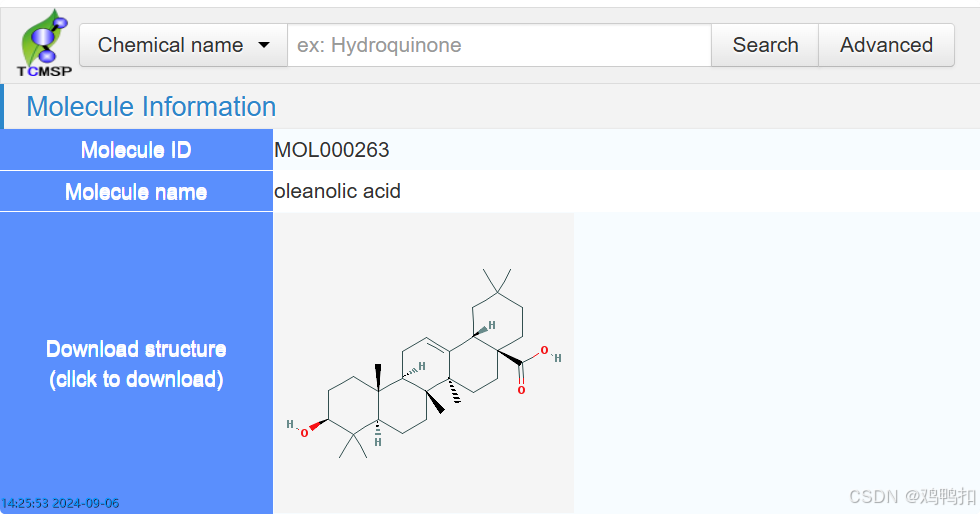
将分类从Herb name改为Chemical name,可以搜索化合物相关的信息,点击图片则下载mol格式文件。
Pubchem查询活性成分的SMILES序列号
可以通过结果预览卡片那里直接看到:

也可以来到结果详情页中看到:

如上两张图对应的是两个化合物,可以看到有两种SMILES序列号。这两者的区别是:
Canonical SMILES: 化合物的唯一SMILES字符串,由 "正则化 "算法生成。
Isomeric SMILES : 一个具有立体化学和同位素规格的SMILES字符串。
有Isomeric SMILES时就用它。
Pubchem下载活性成分的3D的SDF格式文件
Pubchem主要用来下载活性成分的mol/SDF格式文件。
Pubchem网站首页如下:

该数据库不可以通过mol ID搜索,我们需要输入药物的英文名后搜索如下:

可以看到最上面是BEST MATCH,最匹配的搜索结果。一般都是这个,但是为了保险起见,我们最好再看下它的MW即分子量,和我们想要的是不是一致的。
点进去后页面如下,接下来我们想要下载化合物的SDF格式文件。可以通过点击2D图片,或者右侧导航栏的Structures,或者右上角的Download三种途径去下载。

点击右侧导航栏的Structures后会来到滑动到如下页面,点击DOWNLOAD COORDINATES即可。



























 2858
2858

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










