目录
Proposed Network Architecture: UNet++(所提出的网络架构:Unet++)
Re-designed skip pathways(新设计的跳槽路径)
Formulation of the Unsupervised Segmentation Problem(无监督分割问题的方程)
USAR for Unsupervised Segmentation Network Learning(提出基于对抗性重构的无监督分割网络模型)
Avoid Trivial Solutions in Unsupervised Network Learning(在无人监督的网络学习中避免平凡解的解决方案)
Objective Function for Learning the Segmentation Network(分割网络学习的目标函数)
Pseudo Labels to Continually Refine the Segmentation Network(引入伪标签不断优化分割网络)
Future Work - Close the Gap with Fully-Supervised Training(未来工作-缩小与全监督的差距)
论文阅读1
UNet++: A Nested U-Net Architecture for Medical Image Segmentation (一种用于医学图像分割的嵌套式的U-Net结构)
Abstract(摘要)
在本文中,我们提出了一种新的、功能更强大的医学图像分割体系结构UNet++。我们的体系结构本质上是一个深度监督(Deep Supervision)的编解码器网络,其中编码器和解码子网络通过一系列嵌套的、密集的跳跃路径连接在一起。实验结果表明,采用深度监督(Deep Supervision)的UNet++比在UNet和wide UNet上的IOU平均增益分别为3.9和3.4。
Introduction(介绍)
最先进(最流行,最厉害)的图像分割模型是编解码器(Decoder和Encoder)体系结构的变体,如U-Net和全卷积网络(FCN)。这些结构都有一个相似之处,就是都有跳跃连接(skip connections),将来自解码器子网络的深层、语义、粗粒度特征映射与来自编码子网络的浅、低级别、细粒度特征映射相结合。事实证明,跳跃连接在恢复目标对象的细粒度细节方面是有效的;即使在复杂背景下也能生成具有精细细节的分割掩模。
为了满足医学图像更精确分割的需求,我们提出了一种新的基于嵌套密集跳过连接的分割体系结构UNET++。我们的体系结构背后的基本假设是,当来自编码器网络的高分辨率特征图在与来自解码器网络的相应的语义丰富的特征图融合之前逐渐丰富时,该模型能够更有效地捕捉前景对象的细粒度细节。实验表明,该结构是有效的,与Unet和宽Unet相比有显著的性能提升。

Proposed Network Architecture: UNet++(所提出的网络架构:Unet++)
图1(a)显示了所提出的体系结构的高级概述。如图所示,UNET++从编码器子网络或主干开始,然后是解码器子网络。UNET++与U-Net(图1a中的黑色组件)的不同之处在于,连接两个子网的重新设计的跳跃路径(以绿色和蓝色显示)以及深度监控(deep supervision)(以红色显示)的使用。
Re-designed skip pathways(新设计的跳槽路径)
重新设计的跳跃路径改变了编码器和解码器子网络的连通性。在U-Net中,编码器的特征图直接由解码器接收,而在UNET++中,编码器的特征图经历了一个密集的卷积块,其卷积层数取决于金字塔的层数。
Deep supervision(深监督网络)
我们建议在UNET++中使用深度监督[6],使模型能够在两种模式下运行:1)精确模式,其中所有分割分支的输出被平均;2)快速模式,其中最终分割图仅从其中一个分割分支中选择,该模式的选择决定了模型剪枝的程度和速度增益。
Experiments(实验)
数据集:如表1所示,我们使用四个医学成像数据集进行模型评估,涵盖不同医学成像模式的病变/器官。

基准模型(Baseline models):为了进行比较,我们使用了原始的U-Net和定制的宽U-Net架构。我们选择U-Net是因为它是图像分割的通用性能基准。我们还设计了一个宽广的U-Net,其参数数量与我们建议的体系结构相似。这是为了确保我们的体系结构带来的性能提升不是简单地因为增加了参数数量。表2详细说明了U-Net和Wide U-Net架构。


图2:U-Net、Wide U-Net和UNET++之间的定性比较,显示了息肉、肝脏和细胞核数据集的分割结果(2D-仅用于清晰的可视化)。
结果:表3比较了U-Net、Wide U-Net和UNET++在肺结节分割、结肠息肉分割、肝脏分割和细胞核分割任务中的数量参数和分割精度。

可以看出,Wide U-Net的性能一直优于U-Net,除了肝脏分割,这两种架构的性能相当。这一改进归功于Wide U-Net中的更多参数。在没有深度监督(deep supervision)的情况下,UNET++在性能上比UNET和Wide U-Net都有显著提高,IOU平均提高了2.8和3.3个点。有深度监督的UNET++比没有深度监督的UNET++平均提高0.6分。

模型剪枝(Model pruning):图3显示了应用不同级别剪枝后UNET++的分割性能。四幅图分别修剪(A)细胞核、(B)结肠息肉、(C)肝脏和(D)肺结节分割任务后UNET++的复杂性、速度和准确性。
Conclusion(结论)
UNet++的第一个优势就是精度的提升,这个应该它整合了不同层次的特征所带来的,第二个是灵活的网络结构配合深监督,让参数量巨大的深度网络在可接受的精度范围内大幅度的缩减参数量。
Unet++网络结构代码
# archs.py Unet++和Unet的网络结构
import torch
from torch import nn
__all__ = ['UNet', 'NestedUNet']
class VGGBlock(nn.Module):
def __init__(self, in_channels, middle_channels, out_channels):
super().__init__()
self.relu = nn.ReLU(inplace=True)
self.conv1 = nn.Conv2d(in_channels, middle_channels, 3, padding=1)
self.bn1 = nn.BatchNorm2d(middle_channels)
self.conv2 = nn.Conv2d(middle_channels, out_channels, 3, padding=1)
self.bn2 = nn.BatchNorm2d(out_channels)
def forward(self, x):
out = self.conv1(x)
out = self.bn1(out)
out = self.relu(out)
out = self.conv2(out)
out = self.bn2(out)
out = self.relu(out)
return out
class UNet(nn.Module):
def __init__(self, num_classes, input_channels=3, **kwargs):
super().__init__()
nb_filter = [32, 64, 128, 256, 512]
self.pool = nn.MaxPool2d(2, 2)
self.up = nn.Upsample(scale_factor=2, mode='bilinear', align_corners=True)#scale_factor:放大的倍数 插值
self.conv0_0 = VGGBlock(input_channels, nb_filter[0], nb_filter[0])
self.conv1_0 = VGGBlock(nb_filter[0], nb_filter[1], nb_filter[1])
self.conv2_0 = VGGBlock(nb_filter[1], nb_filter[2], nb_filter[2])
self.conv3_0 = VGGBlock(nb_filter[2], nb_filter[3], nb_filter[3])
self.conv4_0 = VGGBlock(nb_filter[3], nb_filter[4], nb_filter[4])
self.conv3_1 = VGGBlock(nb_filter[3]+nb_filter[4], nb_filter[3], nb_filter[3])
self.conv2_2 = VGGBlock(nb_filter[2]+nb_filter[3], nb_filter[2], nb_filter[2])
self.conv1_3 = VGGBlock(nb_filter[1]+nb_filter[2], nb_filter[1], nb_filter[1])
self.conv0_4 = VGGBlock(nb_filter[0]+nb_filter[1], nb_filter[0], nb_filter[0])
self.final = nn.Conv2d(nb_filter[0], num_classes, kernel_size=1)
def forward(self, input):
x0_0 = self.conv0_0(input)
x1_0 = self.conv1_0(self.pool(x0_0))
x2_0 = self.conv2_0(self.pool(x1_0))
x3_0 = self.conv3_0(self.pool(x2_0))
x4_0 = self.conv4_0(self.pool(x3_0))
x3_1 = self.conv3_1(torch.cat([x3_0, self.up(x4_0)], 1))
x2_2 = self.conv2_2(torch.cat([x2_0, self.up(x3_1)], 1))
x1_3 = self.conv1_3(torch.cat([x1_0, self.up(x2_2)], 1))
x0_4 = self.conv0_4(torch.cat([x0_0, self.up(x1_3)], 1))
output = self.final(x0_4)
return output
class NestedUNet(nn.Module):
def __init__(self, num_classes, input_channels=3, deep_supervision=False, **kwargs):
super().__init__()
nb_filter = [32, 64, 128, 256, 512]
self.deep_supervision = deep_supervision
self.pool = nn.MaxPool2d(2, 2)
self.up = nn.Upsample(scale_factor=2, mode='bilinear', align_corners=True)
self.conv0_0 = VGGBlock(input_channels, nb_filter[0], nb_filter[0])
self.conv1_0 = VGGBlock(nb_filter[0], nb_filter[1], nb_filter[1])
self.conv2_0 = VGGBlock(nb_filter[1], nb_filter[2], nb_filter[2])
self.conv3_0 = VGGBlock(nb_filter[2], nb_filter[3], nb_filter[3])
self.conv4_0 = VGGBlock(nb_filter[3], nb_filter[4], nb_filter[4])
self.conv0_1 = VGGBlock(nb_filter[0]+nb_filter[1], nb_filter[0], nb_filter[0])
self.conv1_1 = VGGBlock(nb_filter[1]+nb_filter[2], nb_filter[1], nb_filter[1])
self.conv2_1 = VGGBlock(nb_filter[2]+nb_filter[3], nb_filter[2], nb_filter[2])
self.conv3_1 = VGGBlock(nb_filter[3]+nb_filter[4], nb_filter[3], nb_filter[3])
self.conv0_2 = VGGBlock(nb_filter[0]*2+nb_filter[1], nb_filter[0], nb_filter[0])
self.conv1_2 = VGGBlock(nb_filter[1]*2+nb_filter[2], nb_filter[1], nb_filter[1])
self.conv2_2 = VGGBlock(nb_filter[2]*2+nb_filter[3], nb_filter[2], nb_filter[2])
self.conv0_3 = VGGBlock(nb_filter[0]*3+nb_filter[1], nb_filter[0], nb_filter[0])
self.conv1_3 = VGGBlock(nb_filter[1]*3+nb_filter[2], nb_filter[1], nb_filter[1])
self.conv0_4 = VGGBlock(nb_filter[0]*4+nb_filter[1], nb_filter[0], nb_filter[0])
if self.deep_supervision:
self.final1 = nn.Conv2d(nb_filter[0], num_classes, kernel_size=1)
self.final2 = nn.Conv2d(nb_filter[0], num_classes, kernel_size=1)
self.final3 = nn.Conv2d(nb_filter[0], num_classes, kernel_size=1)
self.final4 = nn.Conv2d(nb_filter[0], num_classes, kernel_size=1)
else:
self.final = nn.Conv2d(nb_filter[0], num_classes, kernel_size=1)
def forward(self, input):
# print('input:',input.shape)
x0_0 = self.conv0_0(input)
# print('x0_0:',x0_0.shape)
x1_0 = self.conv1_0(self.pool(x0_0))
# print('x1_0:',x1_0.shape)
x0_1 = self.conv0_1(torch.cat([x0_0, self.up(x1_0)], 1))
# print('x0_1:',x0_1.shape)
x2_0 = self.conv2_0(self.pool(x1_0))
# print('x2_0:',x2_0.shape)
x1_1 = self.conv1_1(torch.cat([x1_0, self.up(x2_0)], 1))
# print('x1_1:',x1_1.shape)
x0_2 = self.conv0_2(torch.cat([x0_0, x0_1, self.up(x1_1)], 1))
print('x0_2:',x0_2.shape)
x3_0 = self.conv3_0(self.pool(x2_0))
print('x3_0:',x3_0.shape)
x2_1 = self.conv2_1(torch.cat([x2_0, self.up(x3_0)], 1))
print('x2_1:',x2_1.shape)
x1_2 = self.conv1_2(torch.cat([x1_0, x1_1, self.up(x2_1)], 1))
print('x1_2:',x1_2.shape)
x0_3 = self.conv0_3(torch.cat([x0_0, x0_1, x0_2, self.up(x1_2)], 1))
print('x0_3:',x0_3.shape)
x4_0 = self.conv4_0(self.pool(x3_0))
print('x4_0:',x4_0.shape)
x3_1 = self.conv3_1(torch.cat([x3_0, self.up(x4_0)], 1))
print('x3_1:',x3_1.shape)
x2_2 = self.conv2_2(torch.cat([x2_0, x2_1, self.up(x3_1)], 1))
print('x2_2:',x2_2.shape)
x1_3 = self.conv1_3(torch.cat([x1_0, x1_1, x1_2, self.up(x2_2)], 1))
print('x1_3:',x1_3.shape)
x0_4 = self.conv0_4(torch.cat([x0_0, x0_1, x0_2, x0_3, self.up(x1_3)], 1))
print('x0_4:',x0_4.shape)
if self.deep_supervision:
output1 = self.final1(x0_1)
output2 = self.final2(x0_2)
output3 = self.final3(x0_3)
output4 = self.final4(x0_4)
return [output1, output2, output3, output4]
else:
output = self.final(x0_4)
return output论文阅读2
Unsupervised Network Learning for Cell (用于细胞分割的无监督网络学习)
Abstract(摘要)
细胞分割是众多生物医学图像研究的基础和关键步骤,对于现在主流的全监督细胞分割算法,虽然效率很高,但需要大量高质量的训练数据,通常标注都是需要大量的人力物力的,有时还需要领域专家标注,需要投入的成本就更大了。为了降低人工标注的负担,本文提出了一种基于对抗性重构的无监督分割网络学习(USAR)模型,能够在没有任何注释的情况下训练细胞分割网络。其核心思想是利用对抗性学习范式(GAN),通过分割网络生成的分割结果对输入图像进行对抗性重建来训练分割网络。USAR模型在两个基准数据集上展示了其在无监督方式训练分割网络方面的应用前景。
Introduction(介绍)
介绍了一下USAR的网络结构,其实就是大致的GAN网络的结构,主要就是一个生成器和一个鉴别器网络,只是这个生成器网络和平时的不太一样,它是由用于生成前景细胞掩码的分割网络和用于生成背景图像的解码器组成的生成器被设计用于重建细胞图像。
我们工作的主要是以下三部分:
1)我们将无监督细胞图像分割描述为一个轻微的欠约束问题:即从N个方程(N个输入图像)中求解N+1个未知数(N个细胞掩码图像+1个背景图像),而从N个输入图像中求解3N个未知数(N个掩码图像、N个前景图像、N个背景图像)。
2)提出了一种对抗性重构的无监督网络学习方案来训练没有任何注释的细胞分割网络USAR。
3)该模型的分割效果明显优于现有的非监督SOTA分割算法。
Methodology(方法部分)
Formulation of the Unsupervised Segmentation Problem(无监督分割问题的方程)
我们首先推导出无监督分割的数学公式:

给定要分割的输入单元图像集合![]() ,其中N是该集合中的图像数量,Mi表示前景细胞图的分割掩码(mask),Fi是前景细胞图,Bi是图片背景。整个公式想表达的意思就是:我们将输入的待分割的细胞图片Ii,可以通过前景细胞分割mask Mi分解为:前景细胞图Fi和背景Bi。
,其中N是该集合中的图像数量,Mi表示前景细胞图的分割掩码(mask),Fi是前景细胞图,Bi是图片背景。整个公式想表达的意思就是:我们将输入的待分割的细胞图片Ii,可以通过前景细胞分割mask Mi分解为:前景细胞图Fi和背景Bi。
对于细胞分割来说,我们的目的就为了获得前景细胞的分割掩码mask,即Mi,在上面等式1中,我们只知道输入的图片Ii,右手边的所有三个变量(Mi,Fi和Bi)都是未知的,所以该方程是欠约束的,为了减轻欠约束的问题(欠约束不太行,和解方程一样,欠约束会导致方程多解,但是我们的Mask只有一个),我们用原始图像Ii替换前景Fi,因为元素图片的的前景部分应该与FI相同的,所以等式1就变成了下面的等式2了:

等式2有两个未知变量(Mi和Bi),比等式1中的三个未知数小,但它仍然是欠约束的。
我们继续进行分析:
当进行特定的生物实验来分析静止图像中细胞的特性或分析图像序列中的细胞动态时,我们知道背景信息(即细胞培养液)是相对稳定的,因此,我们可以假设特定生物中的细胞图像实验有着相似的背景。在此假设下,公式2可以重写为:

即所有背景部分Bi都替换为是相同的背景B表示。
现在我们有N个方程来求解N+1个未知变量,欠约束的困境大大缓解了。虽然公式3中仍然存在一些平凡解(例如,Mi=1),但我们将在3.3节中讨论如何避免它们(在损失函数中约束做到不让Mi=1)。
USAR for Unsupervised Segmentation Network Learning(提出基于对抗性重构的无监督分割网络模型)
在对无监督问题(即公式3)进行数学描述之后,我们提出了基于对抗性重构的无监督分割网络学习(USAR)(Unsupervised Segmentation network learning by Adversarial Reconstruction)(图1)来训练无注释的分割网络来训练无注释的分割网络。

USAS模型的具体工作流程:
1.输入的image set {I1,….,In} 通过分割网络如Unet网络,来生成对应的cell Masks {M1,….,Mn},Mi和Ii逐元素相乘可以得到前景cell图
2.将随机采样的噪声矢量z输入解码器,对于所有输入图像以仅合成一个背景B
3.前景cell图 = Mi * Ii ,就是上面说的Mi和Ii逐元素相乘可以得到前景cell图
4. 非cell背景图 = (1-Mi)* B,背景的mask和合成的背景B逐元素相乘可以得到细胞背景。
5.重建图像I^i = .前景cell图 + 非cell背景图 ,将细胞区域和非细胞背景区域进行元素求和,得到重建图像I^i。
6.送入鉴别器,损失回传训练生成器
分割网络和背景解码器构成图像重建的生成器,而建立鉴别器来调节生成器,使得重建图像的分布与原始图像对齐。
Avoid Trivial Solutions in Unsupervised Network Learning(在无人监督的网络学习中避免平凡解的解决方案)

对于公式3,有两种可能的平凡segmentation:
1) a“full”segmentation:UNet将整个细胞图像分割为前景(即Mi=1),
在这种情况下,任何合成的背景图像B都可以满足重建。(即整张cell图片都被认为是mask,)
2) an“empty”segmentation: UNet将整个细胞图像分割为背景(即Mi=0),
在这种情况下,任何符合数据分布image set {I1,….,In}的合成背景图像B都可以满足无监督训练,因为默认鉴别器只能测量分布方向的差异,而对重建图像的内容没有约束。(即可能得到所谓的模式崩塌)
为了避免这两个平凡解,我们相应地提出了两个约束:
1. Sparsity for “non-full” segmentation(非全分割):为了防止UNet生成“全”分割,我们在生成的cell掩码上增加了一个稀疏约束||Mi||1(应该是L1范数),强制只有部分生成的掩码为非零(即前景),而大部分为零(即背景)(即只有前景mask为1,背景都要为0)。
2.为了避免“空”分割,即全部mask像素值为0,我们在重建图像I^i和原始输入图像Ii之间增加了一个内容损失,定义为提取的图像特征f(I^i)和f(Ii)之间的差值,这强制每个重建图像I^i应该具有与原始Ii相同的内容信息。(其中f是一个用于特征提取的预先训练的CNN,例如本文使用的是在ImageNet上预先训练的VGG16)。
Objective Function for Learning the Segmentation Network(分割网络学习的目标函数)
其中:G代表生成器:Unet+Background decoder(Generator),D代表鉴别器(Discriminator)
z是噪声向量用来产生背景B的,G(Ii,z)是重建图像I^i,λs和λp是平衡损失项的两个超参数。
整个损失函数的意思是,在训练生成器时,我们需要让生成器的重建图像在鉴别器中打高分,非空和非全损失最小。在训练鉴别器时,让鉴别器给真实图像打高分,给重建图像打低分。
Pseudo Labels to Continually Refine the Segmentation Network(引入伪标签不断优化分割网络)
为了进一步完善分割网络,我们可以利用基于伪标签的自训练策略。具体地说,在通过交替优化公式4和公式5获得分割掩码Mi之后(交替训练GAN),我们将图像的伪标签(˜Mi)定义为那些高置信度单元区(例如,大斑点)。然后,利用伪标签对分割网络进行细化,于是生成器的目标函数变为:
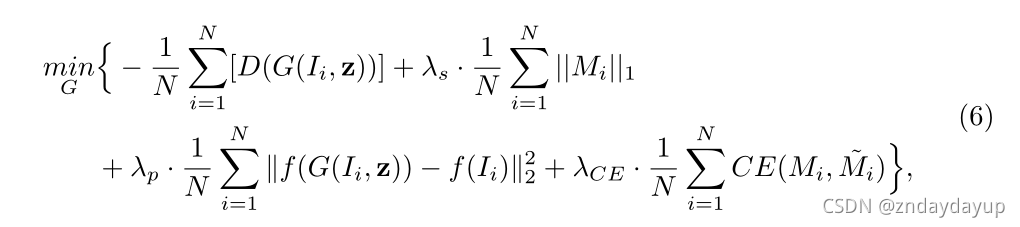
Experimental Results(实验结果)
在PhC-U373数据集和DIC-HeLa数据集上对提出的USAR模型进行了评价。PhC-U373数据集是胶质母细胞瘤-星形细胞,它包含196幅用于训练的未标记图像和34幅带标注的图像,其中10幅用于验证,24幅用于测试。
DIC-HeLa数据集,训练/验证/测试集分割为230/48/120。我们实验中的图像大小为512∗512。
采用Dice分割度量法进行定量评价, Dice的值落在[0,1]中,值越大表示分割越好。
 首先尝试了一种传统的非监督分割方法OTSU(阈值分割算法),但是分割结果并不令人满意。这是因为在这两个数据集中,一些cell的像素强度与背景非常相似,特别是在“DICHeLa”数据集中。然后,我们将所提出的USAR模型与两种最先进的非监督方法(SOTA):BPNET和RedrawNet进行了比较。如表1所示。
首先尝试了一种传统的非监督分割方法OTSU(阈值分割算法),但是分割结果并不令人满意。这是因为在这两个数据集中,一些cell的像素强度与背景非常相似,特别是在“DICHeLa”数据集中。然后,我们将所提出的USAR模型与两种最先进的非监督方法(SOTA):BPNET和RedrawNet进行了比较。如表1所示。

图2给出了这三种模型之间的一些直观比较,这也说明了所提出的USAR模式的优越性。
Future Work - Close the Gap with Fully-Supervised Training(未来工作-缩小与全监督的差距)
为了弥补这一差距,我们可以考虑半监督学习和主动学习作为未来的工作,以用少量的人力来提高准确率。
Conclusion(结论)
本文提出了一种基于对抗性重构的无监督分割网络学习(USAR)模型,能够在没有标注的情况下训练细胞分割网络,在两个公共细胞分割数据集上的验证表明,USAR模型在以无监督的方式训练高质量的分割网络方面取得了令人满意的结果。






















 5401
5401











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








