
在各种CpG区域的分布
CpG shores等概念是根据与CpG island的距离进行定义的。
CpG Shores 指的是位于CpG island上下游2kb 以内的区域;CpG Shelves指的是位于CpG shores 上下游2kb以内的区域;open sea指的是CpG islands, CpG shores, CpG shelves之外的其他区域。
可以看到,位于open sea的探针是最多的。
CpG位点可能位于基因间区Intergenic, 也可能位于基因上,而这个基因可以是编码基因,也可以是非编码基因。
可以看到,位于编码基因上的探针最多,其次是位于基因间区的探针
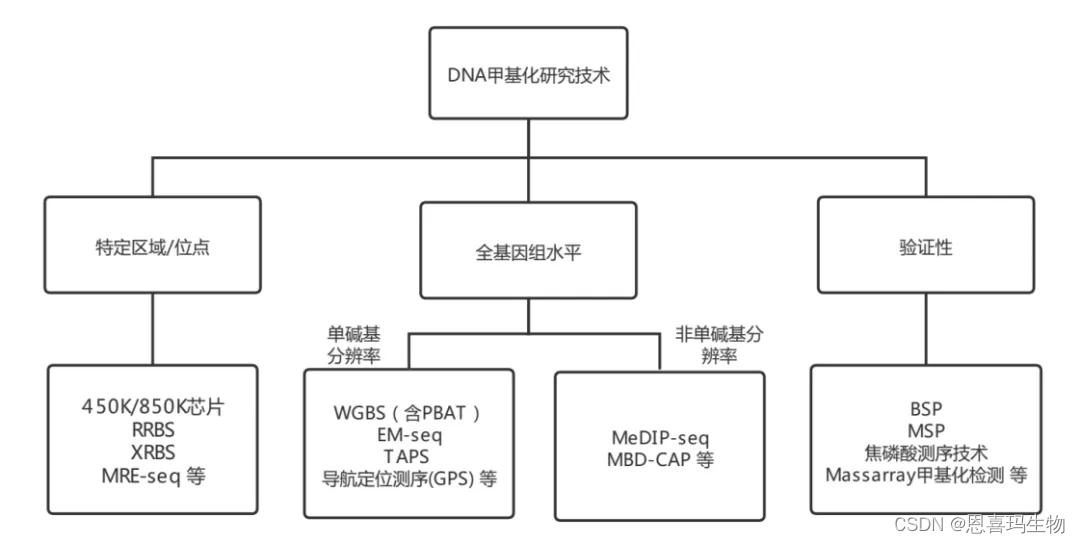
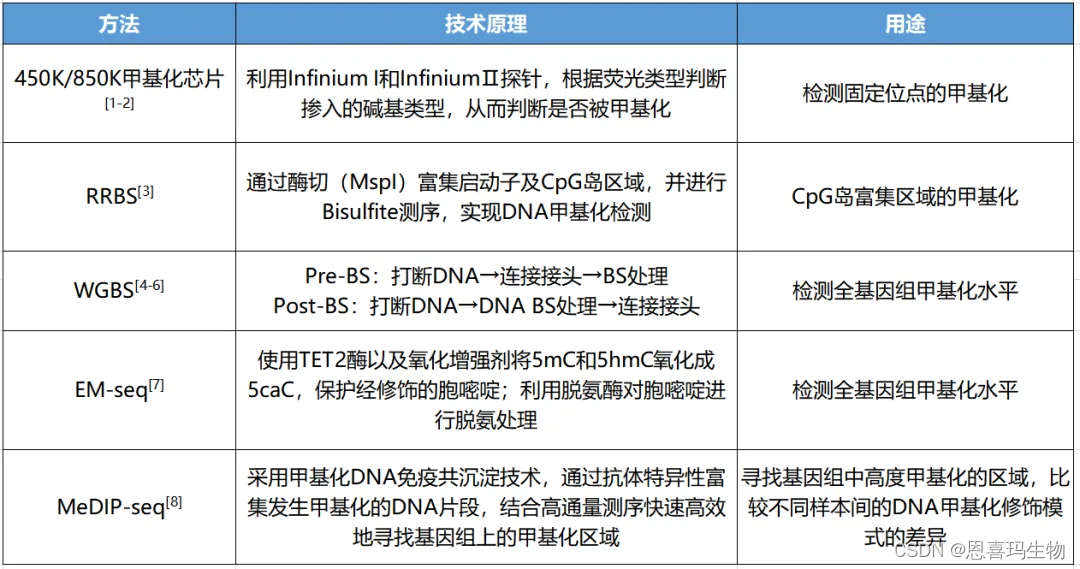
处理流程
0.下载
1.读取
2.质控:缺失值填充、offset、过滤、QC三张图
3.差异分析:标准化,champ分析流程
注意:用logFC而不用deltabeta表示变化倍数;过滤未和基因关联的探针 filter(gene != "")
差异分析
按差异区域的长度不同分类
DMP:找出一个一个的差异甲基化CDG位点
DMR:(连续的差异片段)一个连续不断都比较长的差异片段,科学家们觉得,这样的连续差异片段,对于基因的影响会更加明显,只找这样的片段,可以使得计算生物学的打击精度更为准确,也可以让最终找出来的结论数据更少,便于实险人员筛选。
DMB:(某个基因附近的全部甲基化探针)更大的差异化region区域。有的科学家觉得,DMR这样的区域还不够显著,DNA上的甲基化出现变化,可能是绵延几千位点的!而且只会在基因以外的区域,但是这些基因以外的区域发生变化,却会导致基因的表达发生变化。你可以想象成,北京周边的河北在大炼钢铁,然后北京也跟看雾霾了,大概就是这意思。
按差异区域的类型不同分类
图片
TSS200:转录起始位点上游200位置
References
https://mp.weixin.qq.com/s/-E50Jvzo8aNqVgvEB0nVGA
https://mp.weixin.qq.com/s/JHrL_DqgQY6Yh18vHySKYg
https://github.com/jmzeng1314/methy_array
https://mp.weixin.qq.com/s/VtuapPafKsZaS_WKuQx4Xg
https://mp.weixin.qq.com/s/mJ8qlSLXvvvLz98NdhL9jA
https://mp.weixin.qq.com/s/fLZFEWHt5K55FffExhD9zA
https://mp.weixin.qq.com/s/12dxY4a_UxdoXQVdIMYZMQ
http://www.360doc.com/content/21/0118/13/72917688\_957595208.shtml





















 368
368

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








