预后模型这一类型的生信文章在前几年非常火,近几年热度略有下降。笔者整理了一份模块化的预后模型代码,包括Lasso-COX回归分析,riskScore的计算和可视化,KM生存分析,nomogram、ROC曲线及决策曲线的绘制,现和大家分享。
所有脚本和测试数据可在下述链接下载:
链接:https://pan.baidu.com/s/1HXCSOarJSSSI7qBWO_ZbEQ?pwd=xg77
提取码:xg77
传送门:
预后模型模块化代码分享(2):Lasso-COX回归分析
预后模型模块化代码分享(3):riskScore可视化
预后模型模块化代码分享(4):nomogram-ROC曲线-DCA决策曲线
注:由于本项目使用tiff()函数保存图像,直接运行source()可能出现部分图像无法保存的问题,此时可打开相应R script运行代码
打开Auto_script_model.R,首先是设置目录、加载所需的包及读取数据:
setwd("E:/Rdata/CSDN/Model_1")
#Library necessary packages
if (T){
library(survival)
library(survminer)
library(dplyr)
library(grid)
library(ggpubr)
library(ggpmisc)
library(pheatmap)
library(timeROC)
library(BiocManager)
library(stringr)
library(plyr)
library(limma)
library(RColorBrewer)
library(scales)
}
#read data
RBP <- read.xlsx("Autophage gene list.xlsx")
data <- read.csv("fpkm.csv",header = T)
sur <- read_xls("clinical.xls")
deg <- read.csv("DEGs.csv",header = T)
trait_list <- c("Age","M","N","T","Treatment")
所需数据分为4部分,RBP为感兴趣的基因集,这里是一个自噬相关的基因集,源自HADb数据库。
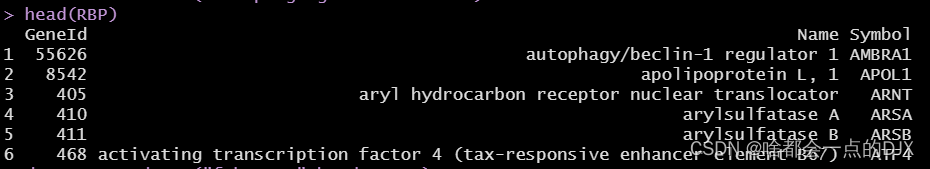
data为表达矩阵,如果是TCGA数据集,推荐使用count数据,本测试集为TCGA网站下载的乳腺浸润癌数据集
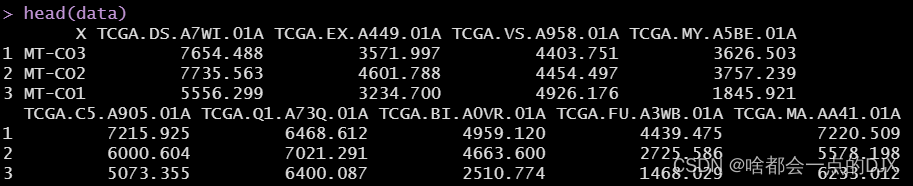
sur为样本临床信息,其中样本名为Sample,生存时间为days_to_death,生存结局为status
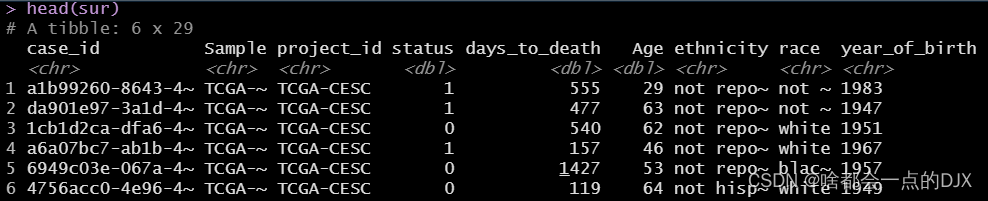
deg为差异基因表达
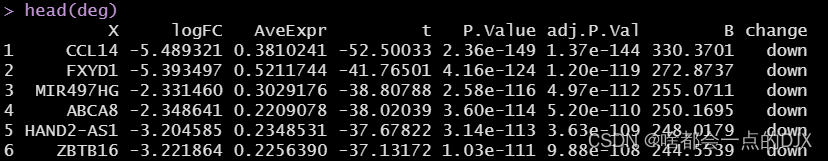
随后进行数据处理,大致流程为:
1.筛选deg,并与RBP取交集,获得gene_list
2.筛选data,仅保留gene_list中包含的基因,并将因R读取不了“-”而变成的“.”转换回去,方便与sur匹配
后续代码主要输入参数:
- data_cox:输入的矩阵数据
- sur_cox:样本的信息,应包括生存时间,生存状态等
- gene_list:感兴趣的基因列表,这里是自噬相关基因集
- time_type:生存时间的单位,这里是天
- time_seq:输入3个数字组成的向量,用于指定nomogram及ROC曲线的时间截断点,这里单位是年
deg <- deg[abs(deg$logFC) >= 1 & deg$adj.P.Val < 0.05,]
gene_RBP <- RBP$V1
index <- duplicated(as.character(gene_RBP))
RBP <- RBP[!index,]
RBP <- RBP[RBP$Sample %in% deg$X]
sur <- dplyr::arrange(sur,Sample)
row.names(data) <- data$X
data <- data[,-1]
colnames(data) <- gsub("\\.","-",substr(colnames(data),0,12))
data <- subset(data,select=sur$Sample)
data <- data[row.names(data) %in% RBP$Sample,]
data <- as.data.frame(t(data))
k <- length(data)
data$name <- row.names(data)
data <- arrange(data,name)
row.names(data) <- data$name
data <- data[,1:k]
data <- as.data.frame(t(data))
data_cox <- data
sur_cox <- sur
gene_list <- RBP$Sample
time_type <- "days"
time_seq <- c(1,2,3)
source("D:/Rcode/Auto_Lasso_cox.R")
source("D:/Rcode/Auto_riskScore_plot.R")
source("D:/Rcode/Auto_nomo_roc.R")
write.csv(cli,"E:/Rdata/CSDN/Model_1/clinical.csv")
最后检查一下数据情况,data_cox行名为基因Symbol,列名为样本名
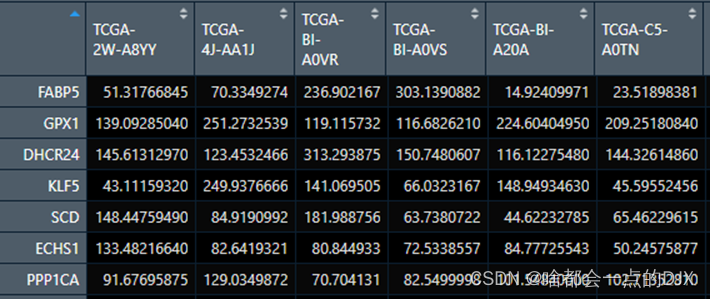
sur_cox样本名为Sample,生存时间为days_to_death,生存结局为status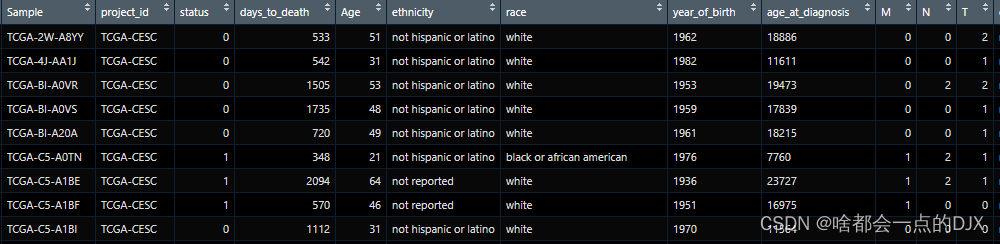























 878
878











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










