本节主要讲解如何将元数据与数据导入生物信息分析软件QIIME2,实现数据导入与检查。
本实战教程将使用来自人源化(humanized)小鼠的一组粪便样品,展示16S rRNA基因扩增子数据的“典型”QIIME 2分析。本教程旨在探讨人源化小鼠的遗传背景影响微生物群落的假设。今天,我们先从示例元数据及数据的导入开始介绍。
01元数据(Metadata)
在开始任何分析之前,熟悉元数据很重要。在本研究中,元数据文件包含7列。
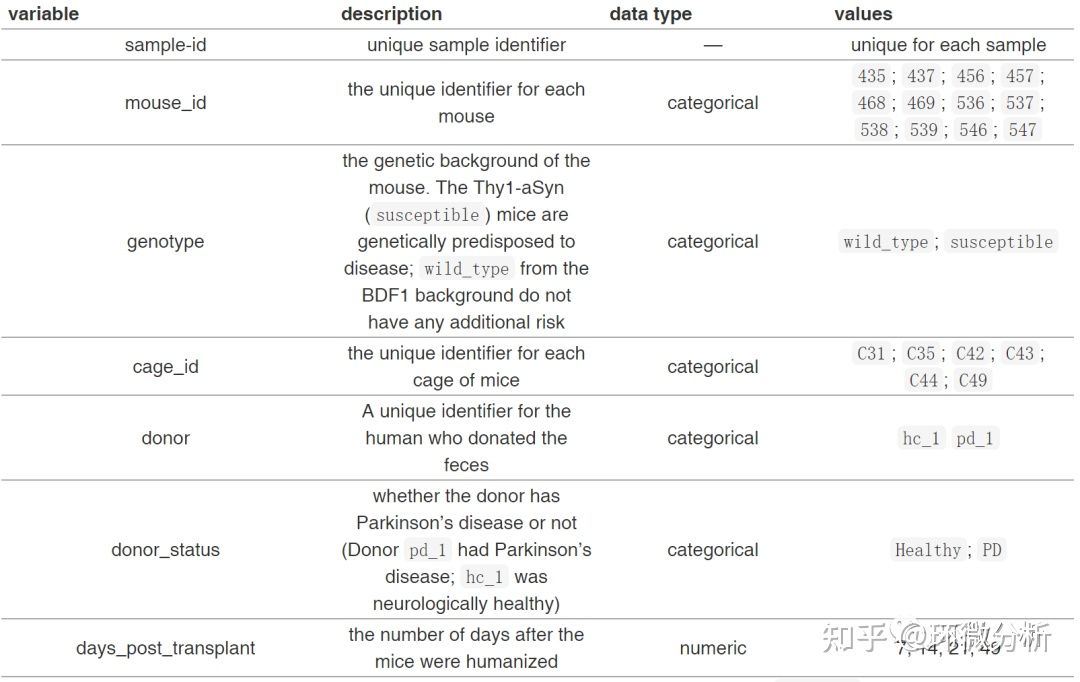
即使mouse ID看起来像一个数字,我们也会使用#q2_type指令指定它是分类型数据。整个教程将使用示例元数据。让我们运行我们的第一个QIIME 2命令,来总结和探索元数据。
qiime metadata tabulate
--m-input-file metadata.tsv
--o-visualization metadata.qzv
输出结果文件:metadata.qzv
| 文件注释:元数据可视化后生成的交互式表格可在网页中查看,也可按任意列排序。 |
02数据导入QIIME 2(Importing data into QIIME 2)
在QIIME 2中,所有数据都被构造为特定语义类型的对象。
示例样品使用EMP 515f-806r引物扩增,并在Illumina MiSeq上用2x150bp试剂盒测序。使用的引物覆盖的高变区长290bp,因此,对于150bp的读数,我们的序列将略微过短,无法在下游进行配对末端分析。因此,我们将使用单端序列。将序列导入为SampleData [SequencesWithQuality],这是拆分后的单端序列格式。如果想导入双端序列,将指定语义类型SampleData [PairedEndSequencesWithQuality]。
教程将使用样本清单格式(manifest format)导入序列,这是一种在QIIME 2中导入拆分样本数据的通用方法。普通用户常用的下机数据格式为.fastq文件,需要创建一个清单文件,然后使用qiime tools import命令手动输入。清单文件是一个文本文件(.tsv或.txt格式),它将示例标识符映射到fastq.gz或fastq的绝对文件路径,其中包含示例的序列和质量数据。清单文件还指示每个fastq.gz或fastq文件中的读取方向。fastq.gz文件位置的绝对文件路径可以包含环境变量(例如$PWD)。
清单文件内容(单端数据):

使用文件清单导入数据
time qiime tools import
--type"SampleData[SequencesWithQuality]"
--input-format SingleEndFastqManifestPhred33V2
--input-path manifest.tsv
--output-path demux_seqs.qza
命令注释:本教程全篇使用单端数据,用清单文件导入双端数据则使用以下命令。
time qiime tools import --type 'SampleData[SequencesWithQuality]' --input-path manifest.txt --output-path paired-end-demux.qza --input-format PairedEndFastqManifestPhred 3
manifest示例:

使用qiime demux summarize命令检查样本的序列和测序深度(它提供每个样本中序列数及序列质量的信息)
time qiime demux summarize
--i-data demux_seqs.qza
--o-visualization demux_seqs.qzv
输出结果文件:demux_seqs.qzv
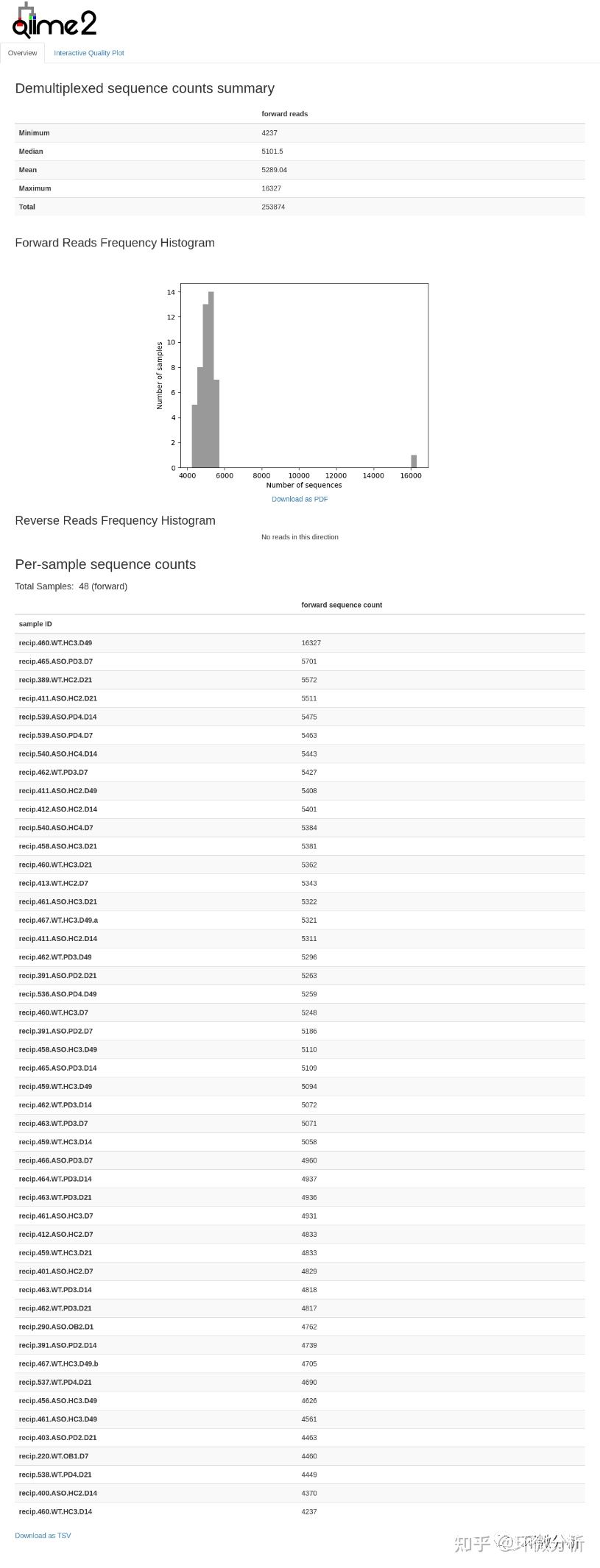
上图分为三部分。其中,扩增子一般是混样测序,上部为拆分序列计数统计;中部为样本分布情况,一般根据频率情况选择合适值进行样本过滤;下部分为每个样本的序列量,一般情况下测序深度最小的样本为数据量最少的样本。

交互图分为三部分。其中,上部为每个位置碱基的质量分布交互式箱线图,单端数据只显示forward reads,双端数据则显示forward reads和reverse reads。根据质量图可以判断序列的质量变化情况,在去噪时根据此图选择裁剪位置和长度。一般从质量开始下降部位开始裁剪。样品质量在30以上是比较好的,一般要保证在20以上,低于20要被裁减掉;鼠标悬停在质量交互图上,即可在中部的文字和表格中显示鼠标所在位置碱基质量的详细信息;下部为拆分样本的长度摘要(一般等长测序无差别)。
本文提供分析所需文件与所有输出结果文件,百度网盘下载链接:
https://pan.baidu.com/s/1AkI5nyNhq33oIw8pfVCsEg
提取码:1234
这篇推文对你有帮助吗?喜欢这篇文章吗?喜欢就不要错过呀,关注本知乎号查看更多的环境微生物生信分析相关文章。亦可以用微信扫描下方二维码关注“环微分析”微信公众号,小编在里面载入了更加完善的学习资料供广大生信分析研究者爱好者参考学习,也希望读者们发现错误后予以指出,小编愿与诸君共同进步!!!

学习环境微生物分析,关注“环微分析”公众号,持续更新,开源免费,敬请关注!
转载自原创文章:
最后,再次感谢你阅读本篇文章,真心希望对你有所帮助。感谢!





















 5984
5984











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








