(最近比较忙,断更了好久)
上一篇文章讲了宏基因组质控流程及质控软件Trimmomatic,这一期我们聊一下另一个比较火的质控软件-Fastp。
Fastp[1]一款超快速全功能的FASTQ文件自动化质控+过滤+校正+预处理软件(看着是不是很牛)。事实上这个软件也确实很牛,不仅包含丰富的功能,而且运算速度还非常快。(感兴趣的小伙伴可以点击以上Fastp查看)
Fastp功能
Fastp功能丰富,包含如下:
对Fastq文件全面质控,生成可视化报告,类似于FastQC;
过滤低质量、过段序列、N含量较多的序列;
对每一个序列的头部或尾部,计算滑动窗内的质量均值,并将均值较低的子序列进行切除(类似Trimmomatic中SLIDINGWINDOW的功能,但是快非常多)。
全局剪裁(在头/尾部,不影响去重),对于Illumina下机数据往往最后一到两个cycle需要这样处理。
去除接头污染,关键是不用输入接头序列文件,它能自己识别;
对于双端测序(PE)的数据,软件会自动查找每一对read的重叠区域,并对该重叠区域中不匹配的碱基对进行校正。
去除尾部的polyG。对于Illumina NextSeq/NovaSeq的测序数据,因为是两色法发光,polyG是常有的事,所以该特性对该两类测序平台默认打开。
对于PE数据中的overlap区间中不一致的碱基对,依据质量值进行校正。
.....
具体功能不一一举例,感兴趣的朋友可以自己去看看。(点击跳转)
Fastp 使用
fastp 作为一个生信软件,我认为它最大的优势就是安装便捷,操作简单。做生信的朋友都知道有时候软件的安装是最折磨人的过程,各种报错和依赖包版本的不适配,让人特别烦躁。而fastp 就是下载后就能使用了,不用看各种依赖包的脸色。而且fastp基于C++编译,所以运行速度也特别快。
#下载Fastp
wget http://opengene.org/fastp/fastp
#授予fastp运行权限
chmod a+x ./fastp
#运行测试
./fastp
#将fastp添加到环境变量中,就可以随时随地调用而不需要添加路径。有需要的做这一步
#/home/biosoft/fastp指fastp的安装路径,将这个改成你自己的路径就好
echo 'export PATH="/home/biosoft/fastp:$PATH" ' >>~/.bashrc
source ~/.bashrc
#单端序列质控,-i 输入待处理的fastq文件,-o 质控后的fastq文件
fastp -i in.fq -o out.fq
#双端序列,-i 输入前端序列,-I 输入后端序列;-o 输出前端序列,-O 输出后端序列,-w 调用线程数
nohup fastp -i test1.fq.gz -I test2.fq.gz -o test_clean_1.fq.gz -O test_clean_2.fq.gz -w 10 &
#很显然,fastp支持fastq和压缩的fastq文件即gz
#多个样本使用for循环就好了,具体用法可以参考上篇Trimmomatic的文章
结果
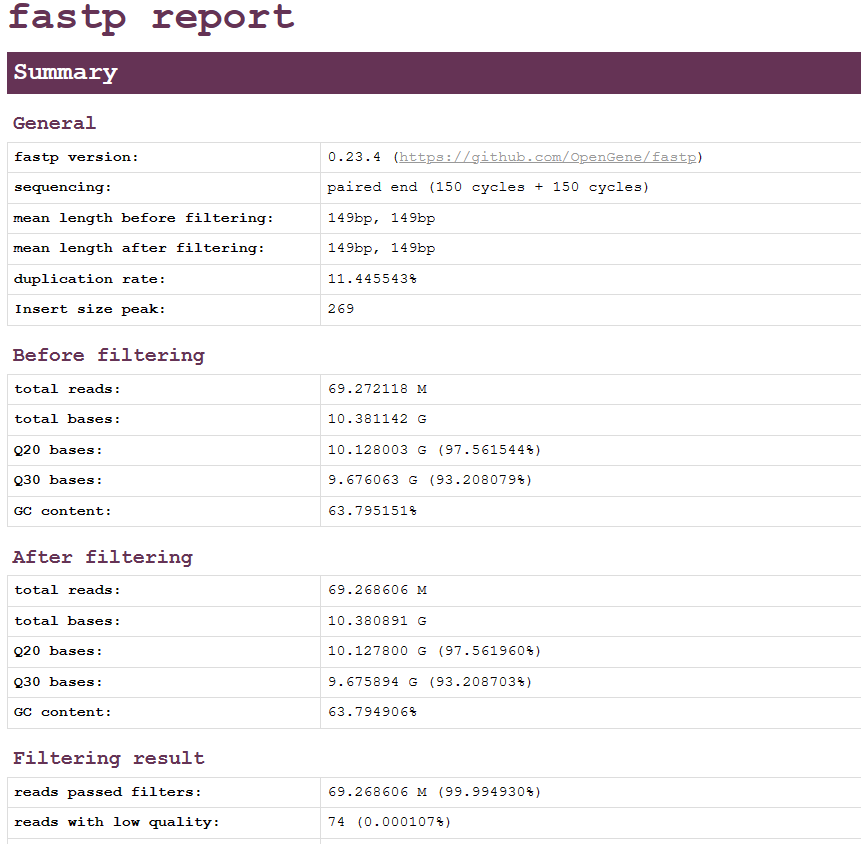
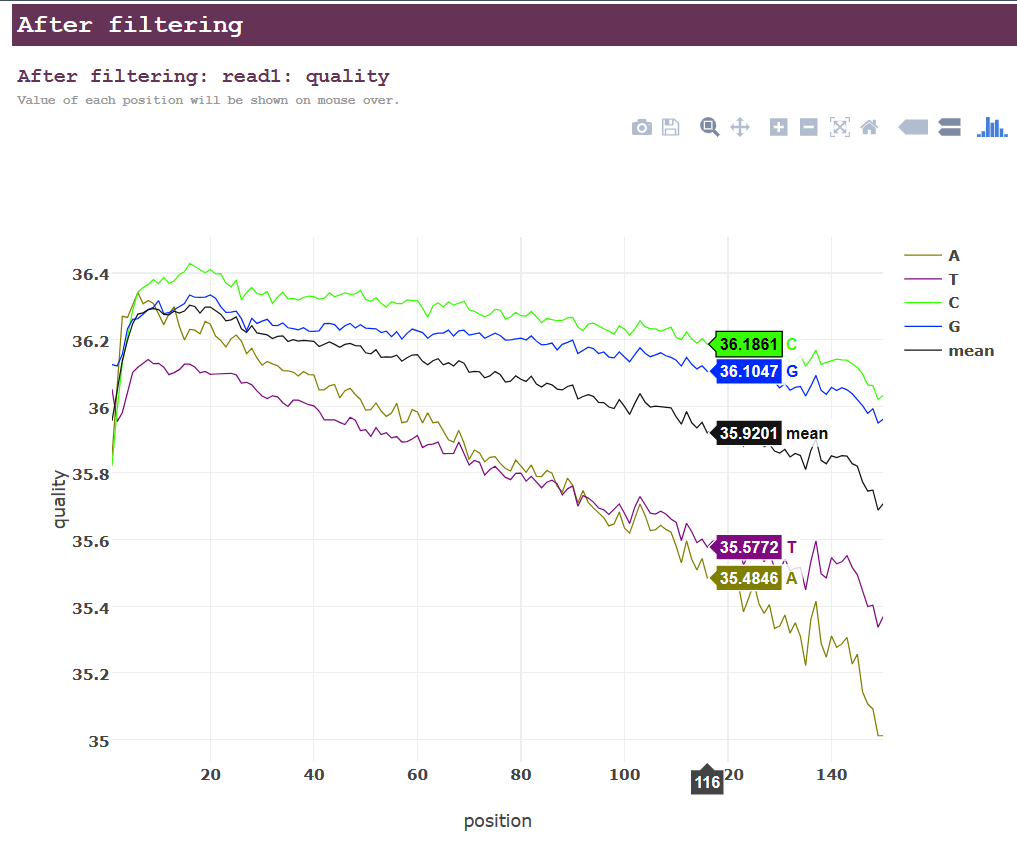
fastp 参数很多,功能也很丰富,篇幅原因我们主要关注其质控的功能。fastp运行结束除了生成质控后的fastq文件,还包括.html和.json两个格式的可视化文件,里面包含了丰富的信息。下面展示了一部分的内容,想要看完整的结果文件可以从百度网盘中下载查看:
链接:https://pan.baidu.com/s/1CcWm0TnZiVCgdRNRY_2fGw?pwd=1111 提取码:1111


Trimmomatic和Fastp的对比
选用一个前后端2.5G的fastq文件,在同一系统下,同时均调用10个线程同时测试这两款质控软件。同时对比程序运行时间、质控后质量以及接头序列的去除等。质控结果由fastqc检验,multiqc统计。
#fastp运行
nohup time fastp -i test1.fq.gz -I test2.fq.gz -o test_clean_1.fq.gz -O test_clean_2.fq.gz -w 10 &
#Trimmomatic
nohup time trimmomatic PE -phred33 test1.fq.gz test2.fq.gz output_forward_paired.fq.gz output_forward_unpaired.fq.gz output_reverse_paired.fq.gz output_reverse_unpaired.fq.gz ILLUMINACLIP:/mnt/data1/home/huodong2/miniconda3/share/trimmomatic/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36 -threads 10 &
###对比一下所消耗的时间
#fastp
1010.94user 32.40system 2:43.73elapsed 637%CPU (0avgtext+0avgdata 2482392maxresident)k
9200inputs+9851472outputs (2major+13787358minor)pagefaults 0swaps
#trimmomatic
3050.30user 61.78system 22:31.13elapsed 230%CPU (0avgtext+0avgdata 458168maxresident)k
9965424inputs+9151688outputs (20major+9107423minor)pagefaults 0swaps
对比两个程序的运行结果,我们可以看到耗费时间上存在较大的差异,Fastp运行时间为2:43.73elapsed,即163.73秒;Trimmomatic运行时间为22:31.13elapsed,即1351.13秒。很明显Fastp在运行效率这一块是领先一大截的。
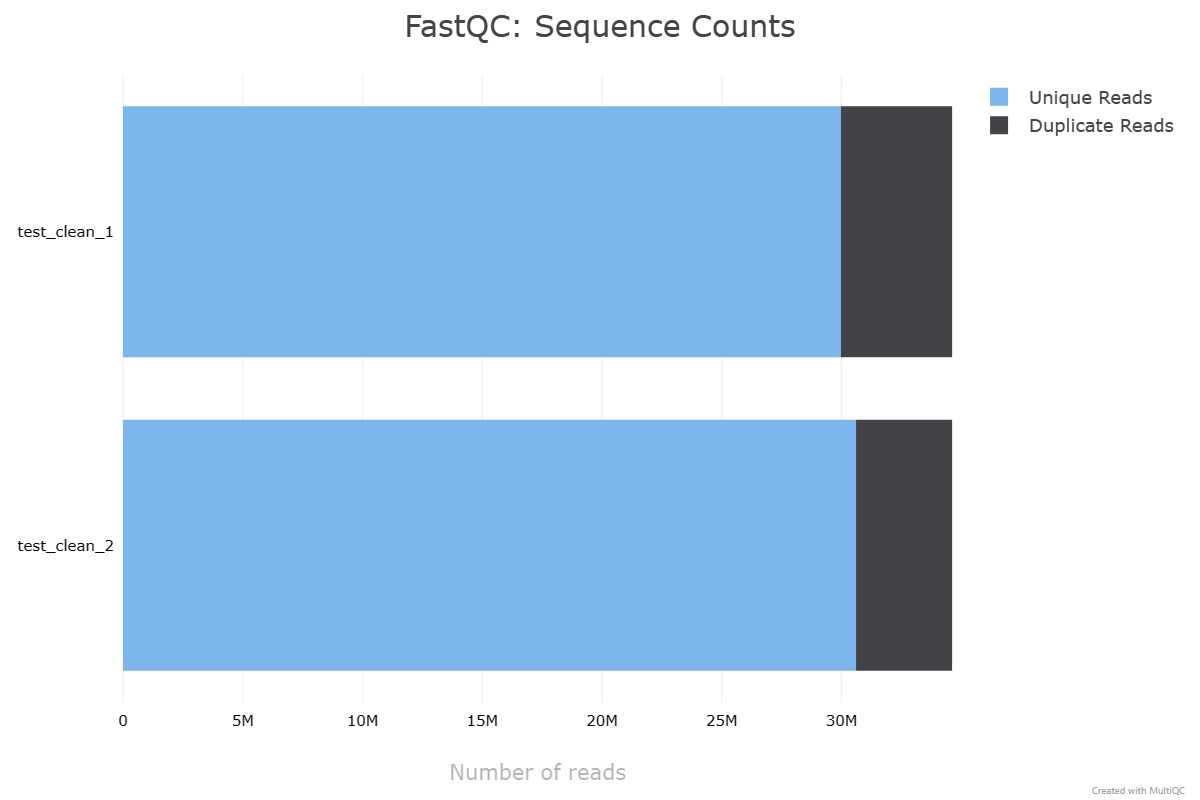
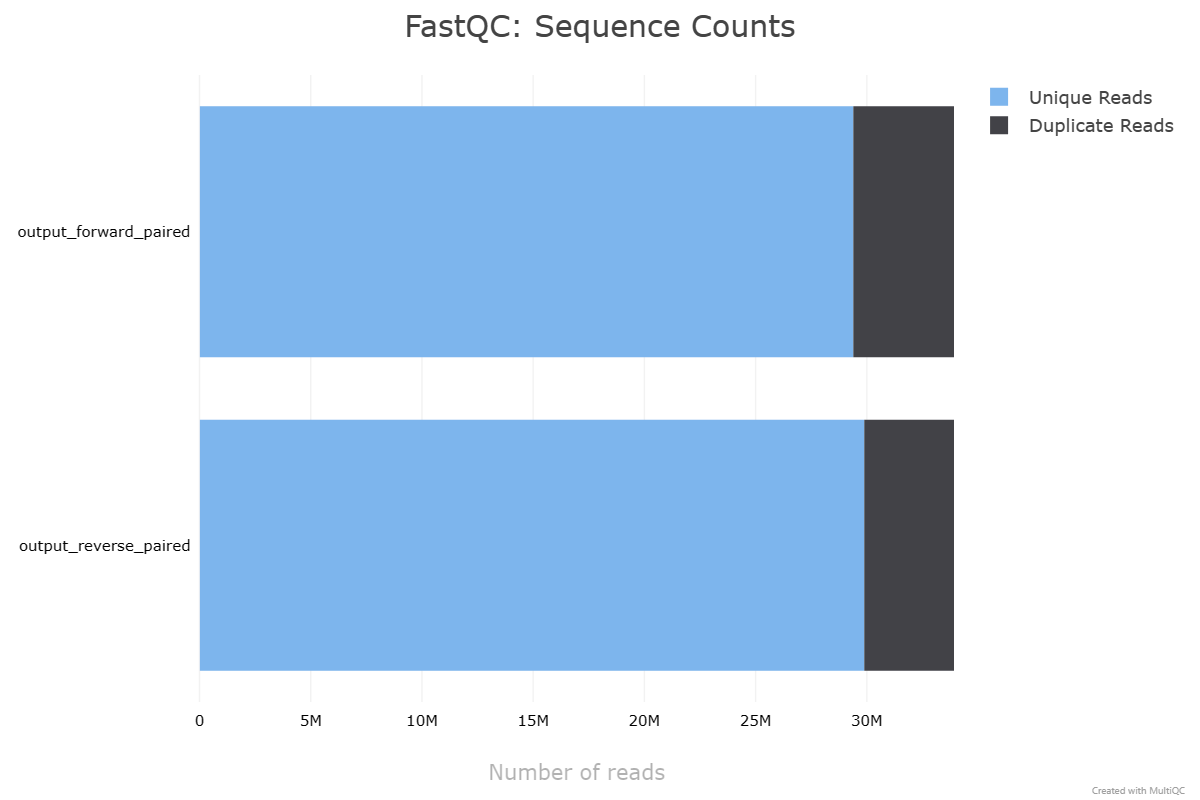
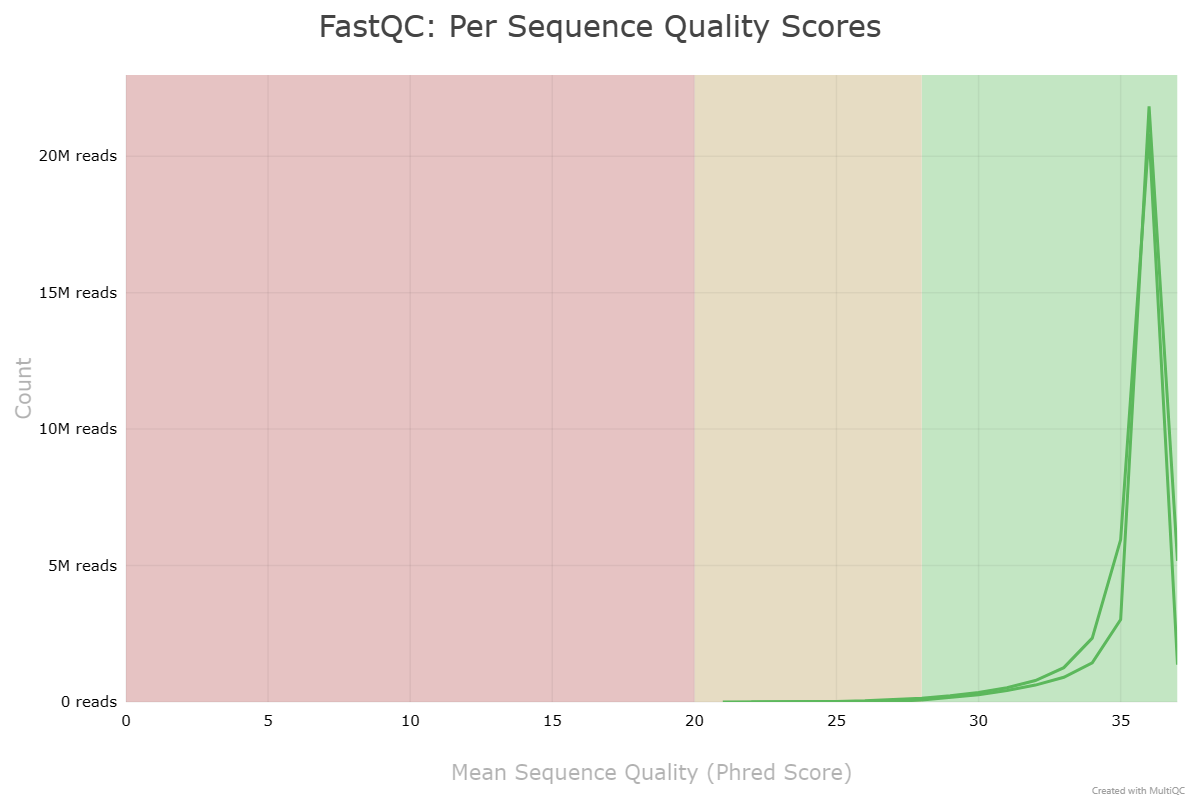
对两个软件质控的文件分别用fastqc统计,比较差异。结果展示如下。无论从接头去除情况、重复序列、序列质量等对比,从下面的结果来看二者都是大同小异,并未出现很大的差距。简单列个表格,看看指控后的序列重复情况以及序列平均质量得分。我们可以直观看到Fastp保留更多的单一序列,单其序列重复比率也会更高一些;而在序列平均质量上,Trimmomatic质控后双端序列均高于Fastp处理的数据。
总的来说,Fastp运行速度远远高于Trimmomatic,而Trimmomatic质控的序列质量略好于Fastp,单二者之间差距不大,况且Fastp在序列上保留也有不错的表现。因此,如果是常规的质控处理,不需要过多的设置的话,我个人比较推荐Fastp;但是对于更加精确的质控,可以选择Trimmomatic来处理。
| Tool | Sequence | Unique | Duplicate | Mean Quality Scores |
|---|---|---|---|---|
| Fastp | Forward | 29976729 | 4657574 | 36.085 |
| Fastp | Reverse | 30608057 | 4026246 | 35.637 |
| Trimmomatic | Forward | 29392779 | 4525057 | 36.254 |
| Trimmomatic | Reverse | 29884824 | 4033052 | 35.878 |
Fastp
Trimmomatic






[1]Shifu Chen, Yanqing Zhou, Yaru Chen, Jia Gu; fastp: an ultra-fast all-in-one FASTQ preprocessor, Bioinformatics, Volume 34, Issue 17, 1 September 2018, Pages i884–i890, https://doi.org/10.1093/bioinformatics/bty560
结语
这一期的内容其实早就想好了怎么写,奈何太忙了没什么时间来写,所以拖更了很久,给大家道个歉,接下来我尽量为大家提供更高质量的文章
最后还是希望大家关注一下我们,同时可以的话也可以推荐给你身边的朋友。























 6539
6539











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








