
微生物一直处于不断变化的状态,估计不同源微生物群落(source,“源”)对特定微生物群落(sink,“汇”)的贡献或混合比例被称为微生物溯源(MST)问题。了解样本中这些生物体来自何处以及如何形成群落,探清微生物的源头对于理解微生物群落构建机制、优先效应以及生物安全是很必要的。
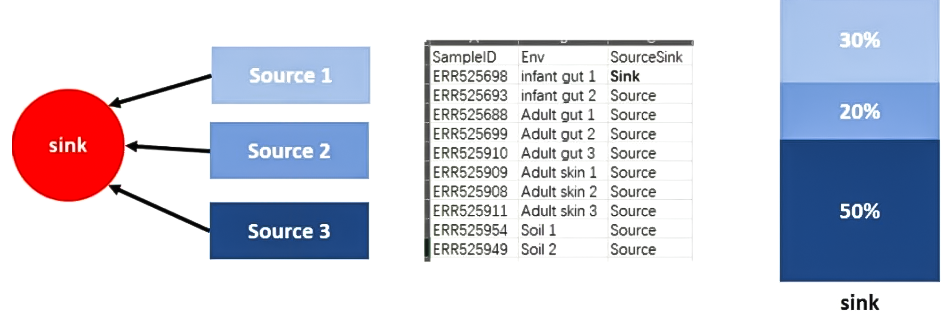
图1 微生物的“汇”和“源”分析
快速准确的微生物来源分析一直是微生物的难点,之前常用随机森林和基于贝叶斯的SourceTracker有较广泛应用,但运行速度和准确度一直不尽人意。2019年,由加州大学洛杉矶分开发的新的方法FEAST Source Tracker[1],可以实现快速、更准确的微生物来源追踪。该软件基于R语言开发,保证了方法跨平台的可用性。
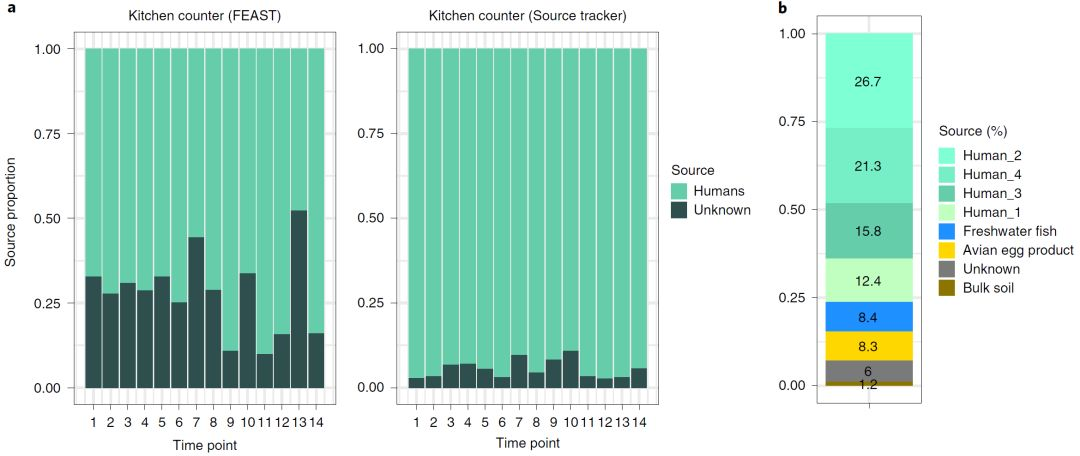
图2 使用Feast和SourceTracker的厨房柜台样品中未知来源的比例分析
A. 来源估计,考虑到12个已知的人类来源(四个居民的手、脚和鼻子)。B. FEAST估计第一时间点一个家庭厨房柜台微生物来源,使用来自地球微生物组项目的额外来源数据。
凌恩开发了


针对肠道、土壤、水体、食品等样本,可以同时快速估计多个样本潜在的多个源环境的贡献,从而帮助解开复杂微生物群落/功能基因的起源。
| 溯源样品 | 肠道、土壤、水体、食品等 |
| 溯源对象 (sink) | 细菌/古菌/病毒等 |
| ARG/MGE/MRG/VFs/KEGG/元素循环等功能基因 | |
| 源环境 (source) | 溯源样品周围的环境(如肠道、土壤、空气、水体等) |
凌恩应用FEAST软件对多种测试数据进行了溯源分析,获得了良好结果,部分结果如下:
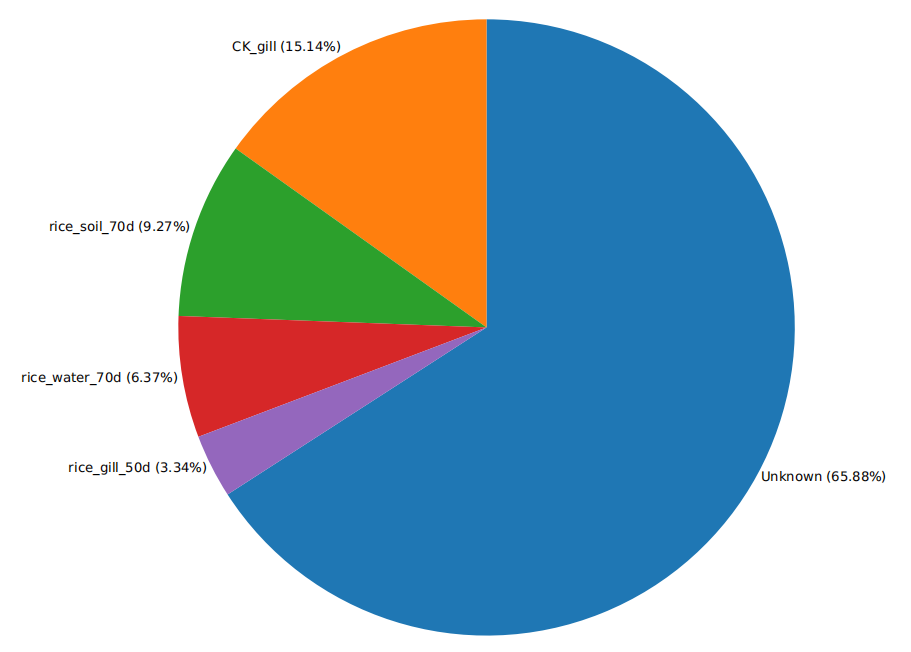
图3 植物根际土壤中微生物来源占比
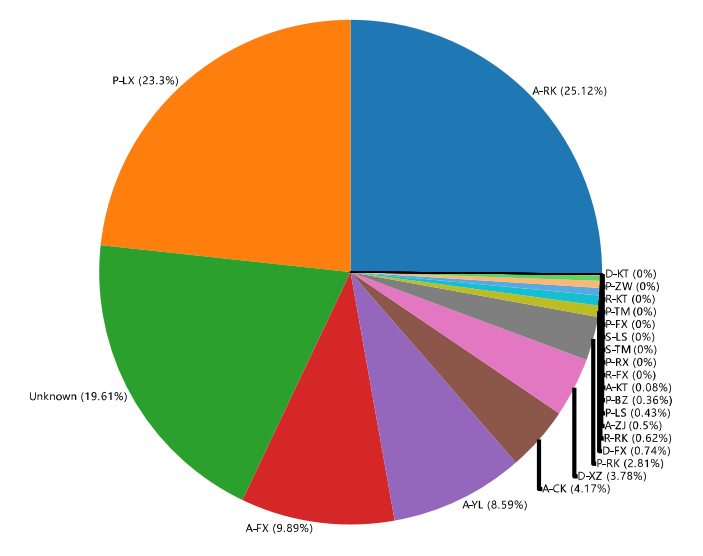
图4 肉类表面微生物抗性基因来源占比
研究示例
2024年最新发表于《Sci Total Environ》的冰川宏基因组,分析了从北极冰川和青藏高原冰川到下游水生态系统的 ARG和病毒传播,来识别ARGs、病毒及其宿主的多样性和分布,追踪ARGs和病毒的来源,以及确定推动ARG和病毒传播的环境影响因子。

图5 冰川抗性宏基因组和宏病毒组[2]
研究基于宏基因组数据鉴定出921个抗生素抗性基因(ARGs),属于22个抗生素类别。在北极冰川和青藏高原冰川中,ARGs的丰度和多样性均高于其下游生境。所有生境中有31个病毒家族,其中32.52%可以被分类到已知科,主要是Phycodnaviridae和Kyanoviridae。
通过FEAST识别源-汇系统中ARGs和病毒的潜在传播途径,发现有72个ARGs(17.39%)和3个病毒科(13.64%)在北极冰川和北冰洋生境之间共享。在青藏高原冰川、青海湖和长江流域之间有14个ARGs(1.82%)和4个病毒科(19.05%)共享。在北极冰川和北冰洋共享的ARGs中,有86.50%在北极冰川中的丰度高于北冰洋。在高原冰川、青海湖和长江流域之间共享的ARGs中,有57.10%在高原冰川中的丰度高于青海湖。在源-汇系统中,不同生境之间ARG和病毒丰度的降低可能表明了ARGs和病毒从丰度高的生境传播到丰度较低的生境。
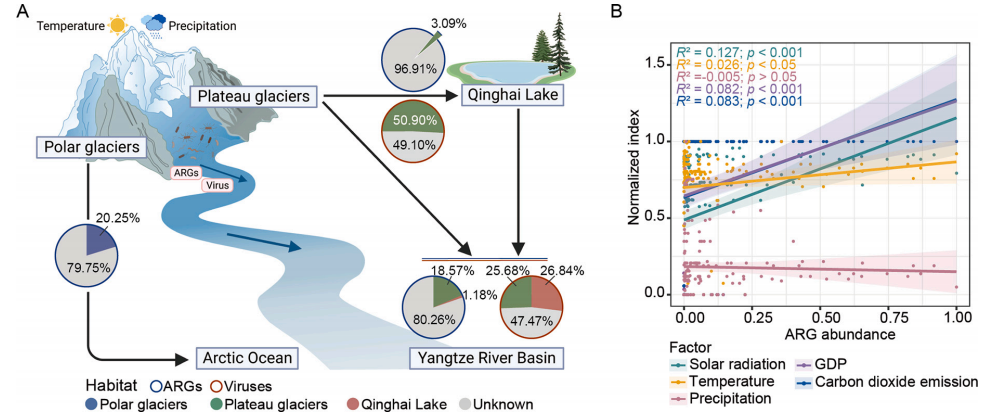
图6 溯源分析和环境因子对ARG丰度的影响[2]
再来看一篇2022年发表于《Nature Communicatons》的文献,分析了玉米植物的木质部中具有可固氮的高度保守的核心细菌微生物群落,该类固氮细菌能为玉米提供氮营养,类似人类的 “肠道细菌群” 。

图7 玉米植物的木质部中具有可固氮的高度保守的核心细菌微生物群落[3]
本文发现茎木质部选择性地招募了以Gammapproteobacteria为主的高度保守的微生物。结果表明,携带固氮酶基因(nifH)的细菌类群在茎木质部的比例大于根和叶等其他器官。在木质部汁液中发现的25个核心细菌分类群中,确认几种分离的菌株是活性固氮或协助生物固氮。
作者使用快速期望最大化微生物源跟踪(FEAST)来跟踪植物细菌微生物组的潜在来源。宿主对从表面到内部的栖息地中微生物群落的组成模式表现出强烈的选择作用。例如,内生菌(RE)中60.61%的微生物组来自根际土壤(RS)。木质部汁液(VE)充当地上和地下部分之间的媒介,其24.21%的细菌微生物组来自RE。
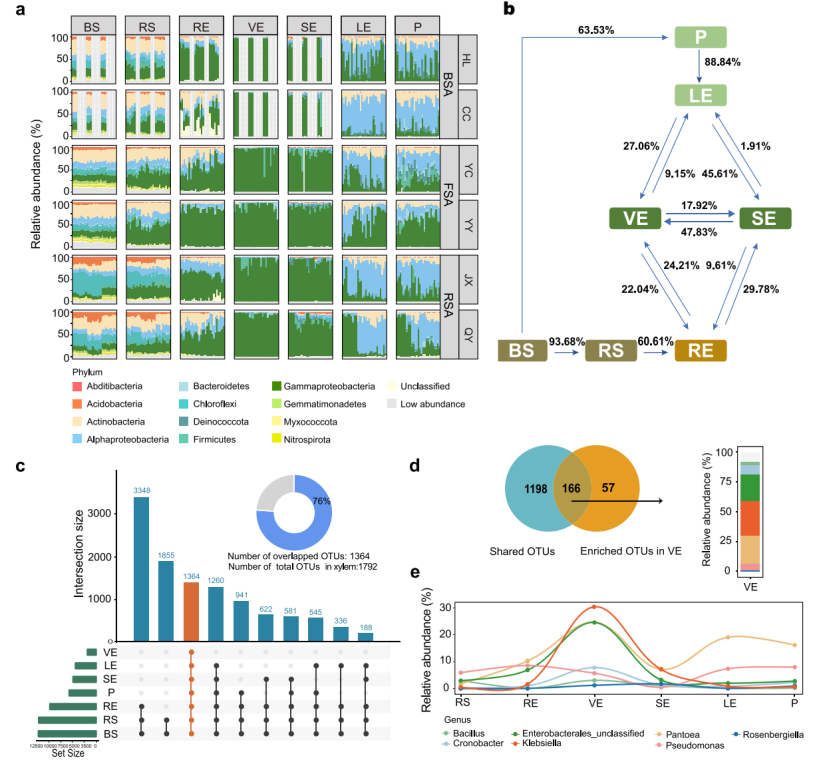
图8 土壤-植物holobiont的玉米细菌群落组成[3]
参考文献
[1]FEAST: fast expectation-maximization for microbial source tracking. Nature Methods, 2019.
[2]Metagenomic analysis of antibiotic-resistance genes and viruses released from glaciers into downstream habitats. Science of the Total Environment, 2024.
[3]A highly conserved core bacterial microbiota with nitrogen-fixation capacity inhabits the xylem sap in maize plants. Nature communications, 2022.






















 4308
4308











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










