前面的近20篇博文已经牵扯到很多机器学习算法咯,已经吊足了胃口,决定从后面开始正式系统的学习机器学习理论,并尝试进入实战阶段,涵盖:
加州理工(caltech)的 Yaser Abu-Mostafa教授的机器学习,偏重于传统统计理论
斯坦福大学(Staford U)的Andrew Ng教授的机器学习,偏重于实用,直观理解
多伦多大学(Tronto U)的Geoffery Hinton教授的高级神经网络,偏重于神经网络和深度学习
斯坦福大学(Staford U)的Daphne Koller教授的概率图模型,偏重于推理和结构化学习
上面的作为理论指导,以《机器学习实战》(machine learning in action)作为主线,机器学习实战更适合码农,因此这个实用性不言而喻。
机器学习分两大类,有监督学习(supervised learning)和无监督学习(unsupervised learning)。有监督学习又可分两类:分类(classification.)和回归(regression),分类的任务就是把一个样本划为某个已知类别,每个样本的类别信息在训练时需要给定,比如人脸识别、行为识别、目标检测等都属于分类。回归的任务则是预测一个数值,比如给定房屋市场的数据(面积,位置等样本信息)来预测房价走势。而无监督学习也可以成两类:聚类(clustering)和密度估计(density estimation),聚类则是把一堆数据聚成弱干组,没有类别信息;密度估计则是估计一堆数据的统计参数信息来描述数据,比如深度学习的RBM。
根据机器学习实战讲解顺序,先学习K近邻法(K Nearest Neighbors-KNN)
K近邻法是有监督学习方法,原理很简单,假设我们有一堆分好类的样本数据,分好类表示每个样本都一个对应的已知类标签,当来一个测试样本要我们判断它的类别是,就分别计算到每个样本的距离,然后选取离测试样本最近的前K个样本的标签累计投票,得票数最多的那个标签就为测试样本的标签。
例子(电影分类):
(图一)
(图一)中横坐标表示一部电影中的打斗统计个数,纵坐标表示接吻次数。我们要对(图一)中的问号这部电影进行分类,其他几部电影的统计数据和类别如(图二)所示:
(图二)
从(图二)中可以看出有三部电影的类别是Romance,有三部电影的类别是Action,那如何判断问号表示的这部电影的类别?根据KNN原理,我们需要在(图一)所示的坐标系中计算问号到所有其他电影之间的距离。计算出的欧式距离如(图三)所示:

(图三)
由于我们的标签只有两类,那假设我们选K=6/2=3,由于前三个距离最近的电影都是Romance,那么问号表示的电影被判定为Romance。
代码实战(Python版本):
先来看看KNN的实现:
- from numpy import *
- import operator
- from os import listdir
- def classify0(inX, dataSet, labels, k):
- dataSetSize = dataSet.shape[0] #获取一条样本大小
- diffMat = tile(inX, (dataSetSize,1)) - dataSet #计算距离
- sqDiffMat = diffMat**2 #计算距离
- sqDistances = sqDiffMat.sum(axis=1) #计算距离
- distances = sqDistances**0.5 #计算距离
- sortedDistIndicies = distances.argsort() #距离排序
- classCount={}
- for i in range(k):
- voteIlabel = labels[sortedDistIndicies[i]] #前K个距离最近的投票统计
- classCount[voteIlabel] = classCount.get(voteIlabel,0) + 1 #前K个距离最近的投票统计
- sortedClassCount = sorted(classCount.iteritems(), key=operator.itemgetter(1), reverse=True) #对投票统计进行排序
- return sortedClassCount[0][0] #返回最高投票的类别
下面取一些样本测试KNN:
- def file2matrix(filename):
- fr = open(filename)
- numberOfLines = len(fr.readlines()) #get the number of lines in the file
- returnMat = zeros((numberOfLines,3)) #prepare matrix to return
- classLabelVector = [] #prepare labels return
- fr = open(filename)
- index = 0
- for line in fr.readlines():
- line = line.strip()
- listFromLine = line.split('\t')
- returnMat[index,:] = listFromLine[0:3]
- classLabelVector.append(int(listFromLine[-1]))
- index += 1
- return returnMat,classLabelVector
- def autoNorm(dataSet):
- minVals = dataSet.min(0)
- maxVals = dataSet.max(0)
- ranges = maxVals - minVals
- normDataSet = zeros(shape(dataSet))
- m = dataSet.shape[0]
- normDataSet = dataSet - tile(minVals, (m,1))
- normDataSet = normDataSet/tile(ranges, (m,1)) #element wise divide
- return normDataSet, ranges, minVals
- def datingClassTest():
- hoRatio = 0.50 #hold out 50%
- datingDataMat,datingLabels = file2matrix('datingTestSet2.txt') #load data setfrom file
- normMat, ranges, minVals = autoNorm(datingDataMat)
- m = normMat.shape[0]
- numTestVecs = int(m*hoRatio)
- errorCount = 0.0
- for i in range(numTestVecs):
- classifierResult = classify0(normMat[i,:],normMat[numTestVecs:m,:],datingLabels[numTestVecs:m],3)
- print "the classifier came back with: %d, the real answer is: %d" % (classifierResult, datingLabels[i])
- if (classifierResult != datingLabels[i]): errorCount += 1.0
- print "the total error rate is: %f" % (errorCount/float(numTestVecs))
- print errorCount
上面的代码中第一个函数从文本文件中读取样本数据,第二个函数把样本归一化,归一化的好处就是降低样本不同特征之间数值量级对距离计算的显著性影响
datingClassTest则是对KNN测试,留了一半数据进行测试,文本文件中的每条数据都有标签,这样可以计算错误率,运行的错误率为:the total error rate is: 0.064000
总结:
优点:高精度,对离群点不敏感,对数据不需要假设模型
缺点:判定时计算量太大,需要大量的内存
工作方式:数值或者类别
下面挑选一步样本数据发出来:
参考文献:machine learning in action
转载请注明来源:http://blog.csdn.net/cuoqu/article/details/9255377
机器学习理论与实战(二)决策树
决策树也是有监督机器学习方法。 电影《无耻混蛋》里有一幕游戏,在德军小酒馆里有几个人在玩20问题游戏,游戏规则是一个设迷者在纸牌中抽出一个目标(可以是人,也可以是物),而猜谜者可以提问题,设迷者只能回答是或者不是,在几个问题(最多二十个问题)之后,猜谜者通过逐步缩小范围就准确的找到了答案。这就类似于决策树的工作原理。(图一)是一个判断邮件类别的工作方式,可以看出判别方法很简单,基本都是阈值判断,关键是如何构建决策树,也就是如何训练一个决策树。
(图一)
构建决策树的伪代码如下:
Check if every item in the dataset is in the same class:
If so return the class label
Else
find the best feature to split the data
split the dataset
create a branch node
for each split
call create Branch and add the result to the branch node
return branch node
原则只有一个,尽量使得每个节点的样本标签尽可能少,注意上面伪代码中一句说:find the best feature to split the data,那么如何find thebest feature?一般有个准则就是尽量使得分支之后节点的类别纯一些,也就是分的准确一些。如(图二)中所示,从海洋中捞取的5个动物,我们要判断他们是否是鱼,先用哪个特征?
(图二)
为了提高识别精度,我们是先用“能否在陆地存活”还是“是否有蹼”来判断?我们必须要有一个衡量准则,常用的有信息论、基尼纯度等,这里使用前者。我们的目标就是选择使得分割后数据集的标签信息增益最大的那个特征,信息增益就是原始数据集标签基熵减去分割后的数据集标签熵,换句话说,信息增益大就是熵变小,使得数据集更有序。熵的计算如(公式一)所示:

(公式一)
有了指导原则,那就进入代码实战阶段,先来看看熵的计算代码:
- def calcShannonEnt(dataSet):
- numEntries = len(dataSet)
- labelCounts = {}
- for featVec in dataSet: #the the number of unique elements and their occurance
- currentLabel = featVec[-1]
- if currentLabel not in labelCounts.keys(): labelCounts[currentLabel] = 0
- labelCounts[currentLabel] += 1 #收集所有类别的数目,创建字典
- shannonEnt = 0.0
- for key in labelCounts:
- prob = float(labelCounts[key])/numEntries
- shannonEnt -= prob * log(prob,2) #log base 2 计算熵
- return shannonEnt
有了熵的计算代码,接下来看依照信息增益变大的原则选择特征的代码:
- def splitDataSet(dataSet, axis, value):
- retDataSet = []
- for featVec in dataSet:
- if featVec[axis] == value:
- reducedFeatVec = featVec[:axis] #chop out axis used for splitting
- reducedFeatVec.extend(featVec[axis+1:])
- retDataSet.append(reducedFeatVec)
- return retDataSet
- def chooseBestFeatureToSplit(dataSet):
- numFeatures = len(dataSet[0]) - 1 #the last column is used for the labels
- baseEntropy = calcShannonEnt(dataSet)
- bestInfoGain = 0.0; bestFeature = -1
- for i in range(numFeatures): #iterate over all the features
- featList = [example[i] for example in dataSet]#create a list of all the examples of this feature
- uniqueVals = set(featList) #get a set of unique values
- newEntropy = 0.0
- for value in uniqueVals:
- subDataSet = splitDataSet(dataSet, i, value)
- prob = len(subDataSet)/float(len(dataSet))
- newEntropy += prob * calcShannonEnt(subDataSet)
- infoGain = baseEntropy - newEntropy #calculate the info gain; ie reduction in entropy
- if (infoGain > bestInfoGain): #compare this to the best gain so far #选择信息增益最大的代码在此
- bestInfoGain = infoGain #if better than current best, set to best
- bestFeature = i
- return bestFeature #returns an integer
从最后一个if可以看出,选择使得信息增益最大的特征作为分割特征,现在有了特征分割准则,继续进入一下个环节,如何构建决策树,其实就是依照最上面的伪代码写下去,采用递归的思想依次分割下去,直到执行完成就构建了决策树。代码如下:
- def majorityCnt(classList):
- classCount={}
- for vote in classList:
- if vote not in classCount.keys(): classCount[vote] = 0
- classCount[vote] += 1
- sortedClassCount = sorted(classCount.iteritems(), key=operator.itemgetter(1), reverse=True)
- return sortedClassCount[0][0]
- def createTree(dataSet,labels):
- classList = [example[-1] for example in dataSet]
- if classList.count(classList[0]) == len(classList):
- return classList[0]#stop splitting when all of the classes are equal
- if len(dataSet[0]) == 1: #stop splitting when there are no more features in dataSet
- return majorityCnt(classList)
- bestFeat = chooseBestFeatureToSplit(dataSet)
- bestFeatLabel = labels[bestFeat]
- myTree = {bestFeatLabel:{}}
- del(labels[bestFeat])
- featValues = [example[bestFeat] for example in dataSet]
- uniqueVals = set(featValues)
- for value in uniqueVals:
- subLabels = labels[:] #copy all of labels, so trees don't mess up existing labels
- myTree[bestFeatLabel][value] = createTree(splitDataSet(dataSet, bestFeat, value),subLabels)
- return myTree
用图二的样本构建的决策树如(图三)所示:
(图三)
有了决策树,就可以用它做分类咯,分类代码如下:
- def classify(inputTree,featLabels,testVec):
- firstStr = inputTree.keys()[0]
- secondDict = inputTree[firstStr]
- featIndex = featLabels.index(firstStr)
- key = testVec[featIndex]
- valueOfFeat = secondDict[key]
- if isinstance(valueOfFeat, dict):
- classLabel = classify(valueOfFeat, featLabels, testVec)
- else: classLabel = valueOfFeat
- return classLabel
最后给出序列化决策树(把决策树模型保存在硬盘上)的代码:
- def storeTree(inputTree,filename):
- import pickle
- fw = open(filename,'w')
- pickle.dump(inputTree,fw)
- fw.close()
- def grabTree(filename):
- import pickle
- fr = open(filename)
- return pickle.load(fr)
优点:检测速度快
缺点:容易过拟合,可以采用修剪的方式来尽量避免
参考文献:machine learning in action
贝叶斯决策一直很有争议,今年是贝叶斯250周年,历经沉浮,今天它的应用又开始逐渐活跃,有兴趣的可以看看斯坦福Brad Efron大师对其的反思,两篇文章:“Bayes'Theorem in the 21st Century”和“A250-YEAR ARGUMENT:BELIEF, BEHAVIOR, AND THE BOOTSTRAP”。俺就不参合这事了,下面来看看朴素贝叶斯分类器。
有时我们想知道给定一个样本时,它属于每个类别的概率是多少,即P(Ci|X),Ci表示类别,X表示测试样本,有了概率后我们可以选择最大的概率的类别。要求这个概率要用经典贝叶斯公式,如(公式一)所示:
(公式一)
(公式一)中的右边每项一般都是可以计算出的,例如(图一)中两个桶中分别装了黑色(Black)和灰色(Grey)的球。

(图一)
假设Bucket A和BucketB是类别,C1和C2,当给定一个球时,我们想判断它最可能从哪个桶里出来的,换句话说是什么类别?这就可以根据(公式一)来算,(公式一)的右边部分的每项都可以计算出来,比如P(gray|bucketA)=2/4,P(gray|bucketB)=1/3,更严格的计算方法是:
P(gray|bucketB) = P(gray andbucketB)/P(bucketB),
而P(gray and bucketB) = 1/7,P(bucketB)= 3/7
那么P(gray|bucketB)=P(gray and bucketB)/ P(bucketB)=(1/7)/(3/7)=1/3
这就是朴素贝叶斯的原理,根据后验概率来判断,选择P(Ci|X)最大的作为X的类别Ci,另外朴素贝叶斯只所以被称为朴素的原因是,它假设了特征之间都是独立的,如(图二)所示:

(图二)
尽管这个假设很不严密,但是在实际应用中它仍然很有效果,比如文本分类,下面就来看下文本分类实战,判断聊天信息是否是辱骂(abusive)信息(也就是类别为两类,是否辱骂信息),在此之前,先强调下,朴素贝叶斯的特征向量可以是多维的,上面的公式是一维的,二维的如(公式二)所示,都是相同的计算方法:

(公式二)
对文本分类,首先的任务就是把文本转成数字向量,也就是提取特征。特征可以说某个关键字在文章中出现的次数(bag of words),比如垃圾邮件中经常出现“公司”,“酬宾”等字样,特征多样,可以根据所需自己建立特征。本例子中采用标记字(token)的方法,标记字可以是任何字符的组合,比如URL,单词,IP地址等,当然判断是否是辱骂信息大多数都是类似于单词的形式。下面来根据代码说下:
首先我们获取一些训练集:
- from numpy import *
- def loadDataSet():
- postingList=[['my', 'dog', 'has', 'flea', 'problems', 'help', 'please'],
- ['maybe', 'not', 'take', 'him', 'to', 'dog', 'park', 'stupid'],
- ['my', 'dalmation', 'is', 'so', 'cute', 'I', 'love', 'him'],
- ['stop', 'posting', 'stupid', 'worthless', 'garbage'],
- ['mr', 'licks', 'ate', 'my', 'steak', 'how', 'to', 'stop', 'him'],
- ['quit', 'buying', 'worthless', 'dog', 'food', 'stupid']]
- classVec = [0,1,0,1,0,1] #1 is abusive, 0 not
- return postingList,classVec
训练集是从聊天室里摘取的6句话,每句话都有一个标签0或者1,表示是否是辱骂信息(abusive or not abusive)。当然可以把每个消息看成是一个文档,只不过文档单词比这个多,但是一样的道理。接下来处理训练集,看看训练集有多少个不同的(唯一的)单词组成。代码如下:
- def createVocabList(dataSet):
- vocabSet = set([]) #create empty set
- for document in dataSet:
- vocabSet = vocabSet | set(document) #union of the two sets
- return list(vocabSet)
该函数返回一个由唯一单词组成的词汇表。接下来就是特征处理的关键步骤,同样先贴代码:
- def setOfWords2Vec(vocabList, inputSet):
- returnVec = [0]*len(vocabList)
- for word in inputSet:
- if word in vocabList:
- returnVec[vocabList.index(word)] = 1
- else: print "the word: %s is not in my Vocabulary!" % word
- return returnVec
这个函数功能:输入词汇表和消息,通过逐个索引词汇表,然后看消息中的是否有对应的字在词汇表中,如果有就标记1,没有就标记0,这样就把每条消息都转成了和词汇表一样长度的有0和1组成的特征向量,如(图三)所示:

(图三)
有了特征向量,我们就可以训练朴素贝叶斯分类器了,其实就是计算(公式三)右边部分的三个概率,(公式三)如下:

(公式三)
其中w是特征向量。
代码如下:
- def trainNB0(trainMatrix,trainCategory):
- numTrainDocs = len(trainMatrix)
- numWords = len(trainMatrix[0])
- pAbusive = sum(trainCategory)/float(numTrainDocs)
- p0Num = ones(numWords); p1Num = ones(numWords) #change to ones()
- p0Denom = 2.0; p1Denom = 2.0 #change to 2.0
- for i in range(numTrainDocs):
- if trainCategory[i] == 1:
- p1Num += trainMatrix[i]
- p1Denom += sum(trainMatrix[i])
- else:
- p0Num += trainMatrix[i]
- p0Denom += sum(trainMatrix[i])
- p1Vect = log(p1Num/p1Denom) #change to log()
- p0Vect = log(p0Num/p0Denom) #change to log()
- return p0Vect,p1Vect,pAbusive
上面的代码中输入的是特征向量组成的矩阵,和一个由标签组成的向量,其中pAbusive是类别概率P(ci),因为只有两类,计算一类后,另外一类可以直接用1-p得出。接下来初始化计算p(wi|c1)和p(wi|c0)的分子和分母,这里惟一让人好奇的就是为什么分母p0Denom和p1Denom都初始化为2?这是因为在实际应用中,我们计算出了(公式三)右半部分的概率后,也就是p(wi|ci)后,注意wi表示消息中的一个字,接下来就是判断整条消息属于某个类别的概率,就要计算p(w0|1)p(w1|1)p(w2|1)的形式,这样如果某个wi为0,这样整个概率都为0,或者都很小连乘后会更小,甚至round off 0。这样就会影响判断,因此把他们转到对数空间中来做运算,对数在机器学习里经常用到,在保持单调的情况下避免因数值运算带来的歧义问题,而且对数可以把乘法转到加法运算,加速了运算。因此上面的代码中把所有的出现次数初始化为1,然后把分母初始为2,接着都是累加,在对数空间中从0还是1开始累加,最后比较大小不会受影响的。
最后贴出分类代码:
- def classifyNB(vec2Classify, p0Vec, p1Vec, pClass1):
- p1 = sum(vec2Classify * p1Vec) + log(pClass1) #element-wise mult
- p0 = sum(vec2Classify * p0Vec) + log(1.0 - pClass1)
- if p1 > p0:
- return 1
- else:
- return 0
分类代码也是在对数空间中计算的后验概率,然后通过比较大小来判断消息属于那一类。
总结:
优点:对小量数据很有效,可以处理多类
缺点:很依赖于数据的准备
朴素贝叶斯在概率图模型里被划为判别模型(Discriminative model)
参考文献:
[1] Machine learning in action.Peter Harrington
[2]Probabilistic graphical model.Daphne Koller
机器学习理论与实战(四)逻辑回归从这节算是开始进入“正规”的机器学习了吧,之所以“正规”因为它开始要建立价值函数(cost function),接着优化价值函数求出权重,然后测试验证。这整套的流程是机器学习必经环节。今天要学习的话题是逻辑回归,逻辑回归也是一种有监督学习方法(supervised machine learning)。逻辑回归一般用来做预测,也可以用来做分类,预测是某个类别^.^!线性回归想比大家都不陌生了,y=kx+b,给定一堆数据点,拟合出k和b的值就行了,下次给定X时,就可以计算出y,这就是回归。而逻辑回归跟这个有点区别,它是一种非线性函数,拟合功能颇为强大,而且它是连续函数,可以对其求导,这点很重要,如果一个函数不可求导,那它在机器学习用起来很麻烦,早期的海维赛德(Heaviside)阶梯函数就因此被sigmoid函数取代,因为可导意味着我们可以很快找到其极值点,这就是优化方法的重要思想之一:利用求导,得到梯度,然后用梯度下降法更新参数。
下面来看看逻辑回归的sigmoid函数,如(图一)所示:
(图一)
(图一)中上图是sigmoid函数在定义域[-5,5] 上的形状,而下图是在定义域[-60,60]上的形状,由这两个图可以看出,它比较适合做二类的回归,因为严重两级分化。Sigmoid函数的如(公式一)所示:
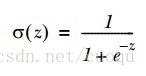
(公式一)
现在有了二类回归函数模型,就可以把特征映射到这个模型上了,而且sigmoid函数的自变量只有一个Z,假设我们的特征为X=[x0,x1,x2…xn]。令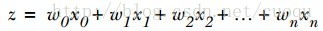
,当给定大批的训练样本特征X时,我们只要找到合适的W=[w0,w1,w2…wn]来正确的把每个样本特征X映射到sigmoid函数的两级上,也就是说正确的完成了类别回归就行了,那么以后来个测试样本,只要和权重相乘后,带入sigmoid函数计算出的值就是预测值啦,很简单是吧。那怎么求权重W呢?
要计算W,就要进入优化求解阶段咯,用的方法是梯度下降法或者随机梯度下降法。说到梯度下降,梯度下降一般对什么求梯度呢?梯度是一个函数上升最快的方向,沿着梯度方向我们可以很快找到极值点。我们找什么极值?仔细想想,当然是找训练模型的误差极值,当模型预测值和训练样本给出的正确值之间的误差和最小时,模型参数就是我们要求的。当然误差最小有可能导致过拟合,这个以后再说。我们先建立模型训练误差价值函数(cost function),如(公式二)所示:

(公式二)
(公式二)中Y表示训练样本真实值,当J(theta)最小时的所得的theta就是我们要求的模型权重,可以看出J(theta)是个凸函数,得到的最小值也是全局最小。对其求导后得出梯度,如(公式三)所示:

(公式三)
由于我们是找极小值,而梯度方向是极大值方向,因此我们取负号,沿着负梯度方向更新参数,如(公式四)所示:

(公式四)
按照(公式四)的参数更新方法,当权重不再变化时,我们就宣称找到了极值点,此时的权重也是我们要求的,整个参数更新示意图如(图二)所示:

(图二)
原理到此为止逻辑回归基本就说完了,下面进入代码实战阶段:
- from numpy import *
- def loadDataSet():
- dataMat = []; labelMat = []
- fr = open('testSet.txt')
- for line in fr.readlines():
- lineArr = line.strip().split()
- dataMat.append([1.0, float(lineArr[0]), float(lineArr[1])])
- labelMat.append(int(lineArr[2]))
- return dataMat,labelMat
- def sigmoid(inX):
- return 1.0/(1+exp(-inX))
上面两个函数分别是加载训练集和定义sigmoid函数,都比较简单。下面发出梯度下降的代码:
- def gradAscent(dataMatIn, classLabels):
- dataMatrix = mat(dataMatIn) #convert to NumPy matrix
- labelMat = mat(classLabels).transpose() #convert to NumPy matrix
- m,n = shape(dataMatrix)
- alpha = 0.001
- maxCycles = 500
- weights = ones((n,1))
- for k in range(maxCycles): #heavy on matrix operations
- h = sigmoid(dataMatrix*weights) #matrix mult
- error = (labelMat - h) #vector subtraction
- weights = weights + alpha * dataMatrix.transpose()* error #matrix mult
- return weights
梯度下降输入训练集和对应标签,接着就是迭代跟新参数,计算梯度,然后更新参数,注意倒数第二句就是按照(公式三)和(公式四)来更新参数。
为了直观的看到我们得到的权重是否正确的,我们把权重和样本打印出来,下面是相关打印代码:
- def plotBestFit(weights):
- import matplotlib.pyplot as plt
- dataMat,labelMat=loadDataSet()
- dataArr = array(dataMat)
- n = shape(dataArr)[0]
- xcord1 = []; ycord1 = []
- xcord2 = []; ycord2 = []
- for i in range(n):
- if int(labelMat[i])== 1:
- xcord1.append(dataArr[i,1]); ycord1.append(dataArr[i,2])
- else:
- xcord2.append(dataArr[i,1]); ycord2.append(dataArr[i,2])
- fig = plt.figure()
- ax = fig.add_subplot(111)
- ax.scatter(xcord1, ycord1, s=30, c='red', marker='s')
- ax.scatter(xcord2, ycord2, s=30, c='green')
- x = arange(-3.0, 3.0, 0.1)
- y = (-weights[0]-weights[1]*x)/weights[2]
- ax.plot(x, y)
- plt.xlabel('X1'); plt.ylabel('X2');
- plt.show()
打印的效果图如(图三)所示:

(图三)
可以看出效果蛮不错的,小错误是难免的,如果训练集没有错误反而危险,说到这基本就说完了,但是考虑到这个方法对少量样本(几百的)还行,在实际中当遇到10亿数量级时,而且特征维数上千时,这种方法很恐怖,光计算梯度就要消耗大量时间,因此要使用随机梯度下降方法。随机梯度下降算法和梯度下降算法原理一样,只是计算梯度不再使用所有样本,而是使用一个或者一小批来计算梯度,这样可以减少计算代价,虽然权重更新的路径很曲折,但最终也会收敛的,如(图四)所示
(图四)
下面也发出随机梯度下降的代码:
- def stocGradAscent1(dataMatrix, classLabels, numIter=150):
- m,n = shape(dataMatrix)
- weights = ones(n) #initialize to all ones
- for j in range(numIter):
- dataIndex = range(m)
- for i in range(m):
- alpha = 4/(1.0+j+i)+0.0001 #apha decreases with iteration, does not
- randIndex = int(random.uniform(0,len(dataIndex)))#go to 0 because of the constant
- h = sigmoid(sum(dataMatrix[randIndex]*weights))
- error = classLabels[randIndex] - h
- weights = weights + alpha * error * dataMatrix[randIndex]
- del(dataIndex[randIndex])
- return weights
最后也给出一个分类的代码,只要把阈值设为0.5,大于0.5划为一类,小于0.5划为另一类就行了,代码如下:
- def classifyVector(inX, weights):
- prob = sigmoid(sum(inX*weights))
- if prob > 0.5: return 1.0
- else: return 0.0
总结:
优点:计算量不高,容易实现,对现实数据也很容易描述
缺点:很容易欠拟合,精度可能也会不高
参考文献:
[1] machine learning in action. Peter Harrington
[2] machine learning.Andrew Ng
做机器学习的一定对支持向量机(support vector machine-SVM)颇为熟悉,因为在深度学习出现之前,SVM一直霸占着机器学习老大哥的位子。他的理论很优美,各种变种改进版本也很多,比如latent-SVM, structural-SVM等。这节先来看看SVM的理论吧,在(图一)中A图表示有两类的数据集,图B,C,D都提供了一个线性分类器来对数据进行分类?但是哪个效果好一些?
(图一)
可能对这个数据集来说,三个的分类器都一样足够好了吧,但是其实不然,这个只是训练集,现实测试的样本分布可能会比较散一些,各种可能都有,为了应对这种情况,我们要做的就是尽可能的使得线性分类器离两个数据集都尽可能的远,因为这样就会减少现实测试样本越过分类器的风险,提高检测精度。这种使得数据集到分类器之间的间距(margin)最大化的思想就是支持向量机的核心思想,而离分类器距离最近的样本成为支持向量。既然知道了我们的目标就是为了寻找最大边距,怎么寻找支持向量?如何实现?下面以(图二)来说明如何完成这些工作。
(图二)
假设(图二)中的直线表示一个超面,为了方面观看显示成一维直线,特征都是超面维度加一维度的,图中也可以看出,特征是二维,而分类器是一维的。如果特征是三维的,分类器就是一个平面。假设超面的解析式为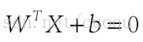 ,那么点A到超面的距离为
,那么点A到超面的距离为
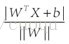 ,下面给出这个距离证明:
,下面给出这个距离证明:
(图三)
在(图三)中,青色菱形表示超面,Xn为数据集中一点,W是超面权重,而且W是垂直于超面的。证明垂直很简单,假设X’和X’’都是超面上的一点,
,因此W垂直于超面。知道了W垂直于超面,那么Xn到超面的距离其实就是Xn和超面上任意一点x的连线在W上的投影,如(图四)所示:

(图四)
而(Xn-X)在W上的投影

(公式一)
注意最后使用了配项法并且用了超面解析式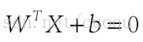 才得出了距离计算。有了距离就可以来推导我们刚开始的想法:使得分类器距所有样本距离最远,即最大化边距,但是最大化边距的前提是我们要找到支持向量,也就是离分类器最近的样本点,此时我们就要完成两个优化任务,找到离分类器最近的点(支持向量),然后最大化边距。如(公式二)所示:
才得出了距离计算。有了距离就可以来推导我们刚开始的想法:使得分类器距所有样本距离最远,即最大化边距,但是最大化边距的前提是我们要找到支持向量,也就是离分类器最近的样本点,此时我们就要完成两个优化任务,找到离分类器最近的点(支持向量),然后最大化边距。如(公式二)所示:

(公式二)
大括号里面表示找到距离分类超面最近的支持向量,大括号外面则是使得超面离支持向量的距离最远,要优化这个函数相当困难,目前没有太有效的优化方法。但是我们可以把问题转换一下,如果我们把 大括号里面的优化问题固定住 ,然后来优化外面的就很容易了,可以用现在的优化方法来求解,因此我们做一个假设,假设大括号里的分子 等于1,那么我们只剩下优化W咯,整个优化公式就可以写成(公式三)的形式:
等于1,那么我们只剩下优化W咯,整个优化公式就可以写成(公式三)的形式:
(公式三)
这下就简单了,有等式约束的优化,约束式子为 ,这个约束等式背后还有个小窍门,假设我们把样本
Xn
的标签设为
1
或者
-1
,当
Xn
在超面上面(或者右边)时,带入超面解析式得到大于
0
的值,乘上标签
1
仍然为本身,可以表示离超面的距离;当
Xn
在超面下面(或者左边)时,带入超面解析式得到小于
0
的值,乘上标签
-1
也是正值,仍然可以表示距离,因此我们把通常两类的标签
0
和
1
转换成
-1
和
1
就可以把标签信息完美的融进等式约束中,(公式三)最后一行也体现出来咯。下面继续说优化 求解(公式四)的方法,在最优化中,通常我们需要求解的最优化问题有如下几类:
,这个约束等式背后还有个小窍门,假设我们把样本
Xn
的标签设为
1
或者
-1
,当
Xn
在超面上面(或者右边)时,带入超面解析式得到大于
0
的值,乘上标签
1
仍然为本身,可以表示离超面的距离;当
Xn
在超面下面(或者左边)时,带入超面解析式得到小于
0
的值,乘上标签
-1
也是正值,仍然可以表示距离,因此我们把通常两类的标签
0
和
1
转换成
-1
和
1
就可以把标签信息完美的融进等式约束中,(公式三)最后一行也体现出来咯。下面继续说优化 求解(公式四)的方法,在最优化中,通常我们需要求解的最优化问题有如下几类:
(i)无约束优化问题,可以写为:
min f(x);
(ii)有等式约束的优化问题,可以写为:
min f(x),
s.t. h_i(x) = 0; i =1, ..., n
(iii)有不等式约束的优化问题,可以写为:
min f(x),
s.t. g_i(x) <= 0; i =1, ..., n
h_j(x) = 0; j =1,..., m
对于第(i)类的优化问题,常常使用的方法就是Fermat定理,即使用求取f(x)的导数,然后令其为零,可以求得候选最优值,再在这些候选值中验证;如果是凸函数,可以保证是最优解。
对于第(ii)类的优化问题,常常使用的方法就是拉格朗日乘子法(LagrangeMultiplier),即把等式约束h_i(x)用一个系数与f(x)写为一个式子,称为拉格朗日函数,而系数称为拉格朗日乘子。通过拉格朗日函数对各个变量求导,令其为零,可以求得候选值集合,然后验证求得最优值。
对于第(iii)类的优化问题,常常使用的方法就是KKT条件。同样地,我们把所有的等式、不等式约束与f(x)写为一个式子,也叫拉格朗日函数,系数也称拉格朗日乘子,通过一些条件,可以求出最优值的必要条件,这个条件称为KKT条件。
而(公式三)很明显符合第二类优化方法,因此可以使用拉格朗日乘子法来对其求解,在求解之前,我们先对(公式四)做个简单的变换。最大化||W||的导数可以最小化||W||或者W’W,如(公式四)所示:

(公式四)
套进拉格朗日乘子法公式得到如(公式五)所示的样子:

(公式五)
在(公式五)中通过拉格朗日乘子法函数分别对W和b求导,为了得到极值点,令导数为0,得到
,然后把他们代入拉格朗日乘子法公式里得到(公式六)的形式:

(公式六)
(公式六)后两行是目前我们要求解的优化函数,现在只需要做个二次规划即可求出alpha,二次规划优化求解如(公式七)所示:

(公式七)
通过(公式七)求出alpha后,就可以用(公式六)中的第一行求出W。到此为止,SVM的公式推导基本完成了,可以看出数学理论很严密,很优美,尽管有些同行们认为看起枯燥,但是最好沉下心来从头看完,也不难,难的是优化。二次规划求解计算量很大,在实际应用中常用SMO(Sequential minimal optimization)算法,SMO算法打算放在下节结合代码来说。
参考文献:
[1]machine learning in action. Peter Harrington
[2] Learning From Data. Yaser S.Abu-Mostafa
上节基本完成了SVM的理论推倒,寻找最大化间隔的目标最终转换成求解拉格朗日乘子变量alpha的求解问题,求出了alpha即可求解出SVM的权重W,有了权重也就有了最大间隔距离,但是其实上节我们有个假设:就是训练集是线性可分的,这样求出的alpha在[0,infinite]。但是如果数据不是线性可分的呢?此时我们就要允许部分的样本可以越过分类器,这样优化的目标函数就可以不变,只要引入松弛变量


(图五)
这样有了错分类的代价,我们把上节(公式四)的目标函数上添加上这一项错分类代价,得到如(公式八)的形式:

(公式八)
重复上节的拉格朗日乘子法步骤,得到(公式九):

(公式九)
多了一个Un乘子,当然我们的工作就是继续求解此目标函数,继续重复上节的步骤,求导得到(公式十):
(公式十)
又因为alpha大于0,而且Un大于0,所以0<alpha<C,为了解释的清晰一些,我们把(公式九)的KKT条件也发出来(上节中的第三类优化问题),注意Un是大于等于0:

推导到现在,优化函数的形式基本没变,只是多了一项错分类的价值,但是多了一个条件,0<alpha<C,C是一个常数,它的作用就是在允许有错误分类的情况下,控制最大化间距,它太大了会导致过拟合,太小了会导致欠拟合。接下来的步骤貌似大家都应该知道了,多了一个C常量的限制条件,然后继续用SMO算法优化求解二次规划,但是我想继续把核函数也一次说了,如果样本线性不可分,引入核函数后,把样本映射到高维空间就可以线性可分,如(图六)所示的线性不可分的样本:

(图六)
在(图六)中,现有的样本是很明显线性不可分,但是加入我们利用现有的样本X之间作些不同的运算,如(图六)右边所示的样子,而让f作为新的样本(或者说新的特征)是不是更好些?现在把X已经投射到高维度上去了,但是f我们不知道,此时核函数就该上场了,以高斯核函数为例,在(图七)中选几个样本点作为基准点,来利用核函数计算f,如(图七)所示:

(图七)
这样就有了f,而核函数此时相当于对样本的X和基准点一个度量,做权重衰减,形成依赖于x的新的特征f,把f放在上面说的SVM中继续求解alpha,然后得出权重就行了,原理很简单吧,为了显得有点学术味道,把核函数也做个样子加入目标函数中去吧,如(公式十一)所示:

(公式十一)
其中K(Xn,Xm)是核函数,和上面目标函数比没有多大的变化,用SMO优化求解就行了,代码如下:
- def smoPK(dataMatIn, classLabels, C, toler, maxIter): #full Platt SMO
- oS = optStruct(mat(dataMatIn),mat(classLabels).transpose(),C,toler)
- iter = 0
- entireSet = True; alphaPairsChanged = 0
- while (iter < maxIter) and ((alphaPairsChanged > 0) or (entireSet)):
- alphaPairsChanged = 0
- if entireSet: #go over all
- for i in range(oS.m):
- alphaPairsChanged += innerL(i,oS)
- print "fullSet, iter: %d i:%d, pairs changed %d" % (iter,i,alphaPairsChanged)
- iter += 1
- else:#go over non-bound (railed) alphas
- nonBoundIs = nonzero((oS.alphas.A > 0) * (oS.alphas.A < C))[0]
- for i in nonBoundIs:
- alphaPairsChanged += innerL(i,oS)
- print "non-bound, iter: %d i:%d, pairs changed %d" % (iter,i,alphaPairsChanged)
- iter += 1
- if entireSet: entireSet = False #toggle entire set loop
- elif (alphaPairsChanged == 0): entireSet = True
- print "iteration number: %d" % iter
- return oS.b,oS.alphas
下面演示一个小例子,手写识别。
(1)收集数据:提供文本文件
(2)准备数据:基于二值图像构造向量
(3)分析数据:对图像向量进行目测
(4)训练算法:采用两种不同的核函数,并对径向基函数采用不同的设置来运行SMO算法。
(5)测试算法:编写一个函数来测试不同的核函数,并计算错误率
(6)使用算法:一个图像识别的完整应用还需要一些图像处理的只是,此demo略。
完整代码如下:
- from numpy import *
- from time import sleep
- def loadDataSet(fileName):
- dataMat = []; labelMat = []
- fr = open(fileName)
- for line in fr.readlines():
- lineArr = line.strip().split('\t')
- dataMat.append([float(lineArr[0]), float(lineArr[1])])
- labelMat.append(float(lineArr[2]))
- return dataMat,labelMat
- def selectJrand(i,m):
- j=i #we want to select any J not equal to i
- while (j==i):
- j = int(random.uniform(0,m))
- return j
- def clipAlpha(aj,H,L):
- if aj > H:
- aj = H
- if L > aj:
- aj = L
- return aj
- def smoSimple(dataMatIn, classLabels, C, toler, maxIter):
- dataMatrix = mat(dataMatIn); labelMat = mat(classLabels).transpose()
- b = 0; m,n = shape(dataMatrix)
- alphas = mat(zeros((m,1)))
- iter = 0
- while (iter < maxIter):
- alphaPairsChanged = 0
- for i in range(m):
- fXi = float(multiply(alphas,labelMat).T*(dataMatrix*dataMatrix[i,:].T)) + b
- Ei = fXi - float(labelMat[i])#if checks if an example violates KKT conditions
- if ((labelMat[i]*Ei < -toler) and (alphas[i] < C)) or ((labelMat[i]*Ei > toler) and (alphas[i] > 0)):
- j = selectJrand(i,m)
- fXj = float(multiply(alphas,labelMat).T*(dataMatrix*dataMatrix[j,:].T)) + b
- Ej = fXj - float(labelMat[j])
- alphaIold = alphas[i].copy(); alphaJold = alphas[j].copy();
- if (labelMat[i] != labelMat[j]):
- L = max(0, alphas[j] - alphas[i])
- H = min(C, C + alphas[j] - alphas[i])
- else:
- L = max(0, alphas[j] + alphas[i] - C)
- H = min(C, alphas[j] + alphas[i])
- if L==H: print "L==H"; continue
- eta = 2.0 * dataMatrix[i,:]*dataMatrix[j,:].T - dataMatrix[i,:]*dataMatrix[i,:].T - dataMatrix[j,:]*dataMatrix[j,:].T
- if eta >= 0: print "eta>=0"; continue
- alphas[j] -= labelMat[j]*(Ei - Ej)/eta
- alphas[j] = clipAlpha(alphas[j],H,L)
- if (abs(alphas[j] - alphaJold) < 0.00001): print "j not moving enough"; continue
- alphas[i] += labelMat[j]*labelMat[i]*(alphaJold - alphas[j])#update i by the same amount as j
- #the update is in the oppostie direction
- b1 = b - Ei- labelMat[i]*(alphas[i]-alphaIold)*dataMatrix[i,:]*dataMatrix[i,:].T - labelMat[j]*(alphas[j]-alphaJold)*dataMatrix[i,:]*dataMatrix[j,:].T
- b2 = b - Ej- labelMat[i]*(alphas[i]-alphaIold)*dataMatrix[i,:]*dataMatrix[j,:].T - labelMat[j]*(alphas[j]-alphaJold)*dataMatrix[j,:]*dataMatrix[j,:].T
- if (0 < alphas[i]) and (C > alphas[i]): b = b1
- elif (0 < alphas[j]) and (C > alphas[j]): b = b2
- else: b = (b1 + b2)/2.0
- alphaPairsChanged += 1
- print "iter: %d i:%d, pairs changed %d" % (iter,i,alphaPairsChanged)
- if (alphaPairsChanged == 0): iter += 1
- else: iter = 0
- print "iteration number: %d" % iter
- return b,alphas
- def kernelTrans(X, A, kTup): #calc the kernel or transform data to a higher dimensional space
- m,n = shape(X)
- K = mat(zeros((m,1)))
- if kTup[0]=='lin': K = X * A.T #linear kernel
- elif kTup[0]=='rbf':
- for j in range(m):
- deltaRow = X[j,:] - A
- K[j] = deltaRow*deltaRow.T
- K = exp(K/(-1*kTup[1]**2)) #divide in NumPy is element-wise not matrix like Matlab
- else: raise NameError('Houston We Have a Problem -- \
- That Kernel is not recognized')
- return K
- class optStruct:
- def __init__(self,dataMatIn, classLabels, C, toler, kTup): # Initialize the structure with the parameters
- self.X = dataMatIn
- self.labelMat = classLabels
- self.C = C
- self.tol = toler
- self.m = shape(dataMatIn)[0]
- self.alphas = mat(zeros((self.m,1)))
- self.b = 0
- self.eCache = mat(zeros((self.m,2))) #first column is valid flag
- self.K = mat(zeros((self.m,self.m)))
- for i in range(self.m):
- self.K[:,i] = kernelTrans(self.X, self.X[i,:], kTup)
- def calcEk(oS, k):
- fXk = float(multiply(oS.alphas,oS.labelMat).T*oS.K[:,k] + oS.b)
- Ek = fXk - float(oS.labelMat[k])
- return Ek
- def selectJ(i, oS, Ei): #this is the second choice -heurstic, and calcs Ej
- maxK = -1; maxDeltaE = 0; Ej = 0
- oS.eCache[i] = [1,Ei] #set valid #choose the alpha that gives the maximum delta E
- validEcacheList = nonzero(oS.eCache[:,0].A)[0]
- if (len(validEcacheList)) > 1:
- for k in validEcacheList: #loop through valid Ecache values and find the one that maximizes delta E
- if k == i: continue #don't calc for i, waste of time
- Ek = calcEk(oS, k)
- deltaE = abs(Ei - Ek)
- if (deltaE > maxDeltaE):
- maxK = k; maxDeltaE = deltaE; Ej = Ek
- return maxK, Ej
- else: #in this case (first time around) we don't have any valid eCache values
- j = selectJrand(i, oS.m)
- Ej = calcEk(oS, j)
- return j, Ej
- def updateEk(oS, k):#after any alpha has changed update the new value in the cache
- Ek = calcEk(oS, k)
- oS.eCache[k] = [1,Ek]
- def innerL(i, oS):
- Ei = calcEk(oS, i)
- if ((oS.labelMat[i]*Ei < -oS.tol) and (oS.alphas[i] < oS.C)) or ((oS.labelMat[i]*Ei > oS.tol) and (oS.alphas[i] > 0)):
- j,Ej = selectJ(i, oS, Ei) #this has been changed from selectJrand
- alphaIold = oS.alphas[i].copy(); alphaJold = oS.alphas[j].copy();
- if (oS.labelMat[i] != oS.labelMat[j]):
- L = max(0, oS.alphas[j] - oS.alphas[i])
- H = min(oS.C, oS.C + oS.alphas[j] - oS.alphas[i])
- else:
- L = max(0, oS.alphas[j] + oS.alphas[i] - oS.C)
- H = min(oS.C, oS.alphas[j] + oS.alphas[i])
- if L==H: print "L==H"; return 0
- eta = 2.0 * oS.K[i,j] - oS.K[i,i] - oS.K[j,j] #changed for kernel
- if eta >= 0: print "eta>=0"; return 0
- oS.alphas[j] -= oS.labelMat[j]*(Ei - Ej)/eta
- oS.alphas[j] = clipAlpha(oS.alphas[j],H,L)
- updateEk(oS, j) #added this for the Ecache
- if (abs(oS.alphas[j] - alphaJold) < 0.00001): print "j not moving enough"; return 0
- oS.alphas[i] += oS.labelMat[j]*oS.labelMat[i]*(alphaJold - oS.alphas[j])#update i by the same amount as j
- updateEk(oS, i) #added this for the Ecache #the update is in the oppostie direction
- b1 = oS.b - Ei- oS.labelMat[i]*(oS.alphas[i]-alphaIold)*oS.K[i,i] - oS.labelMat[j]*(oS.alphas[j]-alphaJold)*oS.K[i,j]
- b2 = oS.b - Ej- oS.labelMat[i]*(oS.alphas[i]-alphaIold)*oS.K[i,j]- oS.labelMat[j]*(oS.alphas[j]-alphaJold)*oS.K[j,j]
- if (0 < oS.alphas[i]) and (oS.C > oS.alphas[i]): oS.b = b1
- elif (0 < oS.alphas[j]) and (oS.C > oS.alphas[j]): oS.b = b2
- else: oS.b = (b1 + b2)/2.0
- return 1
- else: return 0
- def smoP(dataMatIn, classLabels, C, toler, maxIter,kTup=('lin', 0)): #full Platt SMO
- oS = optStruct(mat(dataMatIn),mat(classLabels).transpose(),C,toler, kTup)
- iter = 0
- entireSet = True; alphaPairsChanged = 0
- while (iter < maxIter) and ((alphaPairsChanged > 0) or (entireSet)):
- alphaPairsChanged = 0
- if entireSet: #go over all
- for i in range(oS.m):
- alphaPairsChanged += innerL(i,oS)
- print "fullSet, iter: %d i:%d, pairs changed %d" % (iter,i,alphaPairsChanged)
- iter += 1
- else:#go over non-bound (railed) alphas
- nonBoundIs = nonzero((oS.alphas.A > 0) * (oS.alphas.A < C))[0]
- for i in nonBoundIs:
- alphaPairsChanged += innerL(i,oS)
- print "non-bound, iter: %d i:%d, pairs changed %d" % (iter,i,alphaPairsChanged)
- iter += 1
- if entireSet: entireSet = False #toggle entire set loop
- elif (alphaPairsChanged == 0): entireSet = True
- print "iteration number: %d" % iter
- return oS.b,oS.alphas
- def calcWs(alphas,dataArr,classLabels):
- X = mat(dataArr); labelMat = mat(classLabels).transpose()
- m,n = shape(X)
- w = zeros((n,1))
- for i in range(m):
- w += multiply(alphas[i]*labelMat[i],X[i,:].T)
- return w
- def testRbf(k1=1.3):
- dataArr,labelArr = loadDataSet('testSetRBF.txt')
- b,alphas = smoP(dataArr, labelArr, 200, 0.0001, 10000, ('rbf', k1)) #C=200 important
- datMat=mat(dataArr); labelMat = mat(labelArr).transpose()
- svInd=nonzero(alphas.A>0)[0]
- sVs=datMat[svInd] #get matrix of only support vectors
- labelSV = labelMat[svInd];
- print "there are %d Support Vectors" % shape(sVs)[0]
- m,n = shape(datMat)
- errorCount = 0
- for i in range(m):
- kernelEval = kernelTrans(sVs,datMat[i,:],('rbf', k1))
- predict=kernelEval.T * multiply(labelSV,alphas[svInd]) + b
- if sign(predict)!=sign(labelArr[i]): errorCount += 1
- print "the training error rate is: %f" % (float(errorCount)/m)
- dataArr,labelArr = loadDataSet('testSetRBF2.txt')
- errorCount = 0
- datMat=mat(dataArr); labelMat = mat(labelArr).transpose()
- m,n = shape(datMat)
- for i in range(m):
- kernelEval = kernelTrans(sVs,datMat[i,:],('rbf', k1))
- predict=kernelEval.T * multiply(labelSV,alphas[svInd]) + b
- if sign(predict)!=sign(labelArr[i]): errorCount += 1
- print "the test error rate is: %f" % (float(errorCount)/m)
- def img2vector(filename):
- returnVect = zeros((1,1024))
- fr = open(filename)
- for i in range(32):
- lineStr = fr.readline()
- for j in range(32):
- returnVect[0,32*i+j] = int(lineStr[j])
- return returnVect
- def loadImages(dirName):
- from os import listdir
- hwLabels = []
- trainingFileList = listdir(dirName) #load the training set
- m = len(trainingFileList)
- trainingMat = zeros((m,1024))
- for i in range(m):
- fileNameStr = trainingFileList[i]
- fileStr = fileNameStr.split('.')[0] #take off .txt
- classNumStr = int(fileStr.split('_')[0])
- if classNumStr == 9: hwLabels.append(-1)
- else: hwLabels.append(1)
- trainingMat[i,:] = img2vector('%s/%s' % (dirName, fileNameStr))
- return trainingMat, hwLabels
- def testDigits(kTup=('rbf', 10)):
- dataArr,labelArr = loadImages('trainingDigits')
- b,alphas = smoP(dataArr, labelArr, 200, 0.0001, 10000, kTup)
- datMat=mat(dataArr); labelMat = mat(labelArr).transpose()
- svInd=nonzero(alphas.A>0)[0]
- sVs=datMat[svInd]
- labelSV = labelMat[svInd];
- print "there are %d Support Vectors" % shape(sVs)[0]
- m,n = shape(datMat)
- errorCount = 0
- for i in range(m):
- kernelEval = kernelTrans(sVs,datMat[i,:],kTup)
- predict=kernelEval.T * multiply(labelSV,alphas[svInd]) + b
- if sign(predict)!=sign(labelArr[i]): errorCount += 1
- print "the training error rate is: %f" % (float(errorCount)/m)
- dataArr,labelArr = loadImages('testDigits')
- errorCount = 0
- datMat=mat(dataArr); labelMat = mat(labelArr).transpose()
- m,n = shape(datMat)
- for i in range(m):
- kernelEval = kernelTrans(sVs,datMat[i,:],kTup)
- predict=kernelEval.T * multiply(labelSV,alphas[svInd]) + b
- if sign(predict)!=sign(labelArr[i]): errorCount += 1
- print "the test error rate is: %f" % (float(errorCount)/m)
- '''''#######********************************
- Non-Kernel VErsions below
- '''#######********************************
- class optStructK:
- def __init__(self,dataMatIn, classLabels, C, toler): # Initialize the structure with the parameters
- self.X = dataMatIn
- self.labelMat = classLabels
- self.C = C
- self.tol = toler
- self.m = shape(dataMatIn)[0]
- self.alphas = mat(zeros((self.m,1)))
- self.b = 0
- self.eCache = mat(zeros((self.m,2))) #first column is valid flag
- def calcEkK(oS, k):
- fXk = float(multiply(oS.alphas,oS.labelMat).T*(oS.X*oS.X[k,:].T)) + oS.b
- Ek = fXk - float(oS.labelMat[k])
- return Ek
- def selectJK(i, oS, Ei): #this is the second choice -heurstic, and calcs Ej
- maxK = -1; maxDeltaE = 0; Ej = 0
- oS.eCache[i] = [1,Ei] #set valid #choose the alpha that gives the maximum delta E
- validEcacheList = nonzero(oS.eCache[:,0].A)[0]
- if (len(validEcacheList)) > 1:
- for k in validEcacheList: #loop through valid Ecache values and find the one that maximizes delta E
- if k == i: continue #don't calc for i, waste of time
- Ek = calcEk(oS, k)
- deltaE = abs(Ei - Ek)
- if (deltaE > maxDeltaE):
- maxK = k; maxDeltaE = deltaE; Ej = Ek
- return maxK, Ej
- else: #in this case (first time around) we don't have any valid eCache values
- j = selectJrand(i, oS.m)
- Ej = calcEk(oS, j)
- return j, Ej
- def updateEkK(oS, k):#after any alpha has changed update the new value in the cache
- Ek = calcEk(oS, k)
- oS.eCache[k] = [1,Ek]
- def innerLK(i, oS):
- Ei = calcEk(oS, i)
- if ((oS.labelMat[i]*Ei < -oS.tol) and (oS.alphas[i] < oS.C)) or ((oS.labelMat[i]*Ei > oS.tol) and (oS.alphas[i] > 0)):
- j,Ej = selectJ(i, oS, Ei) #this has been changed from selectJrand
- alphaIold = oS.alphas[i].copy(); alphaJold = oS.alphas[j].copy();
- if (oS.labelMat[i] != oS.labelMat[j]):
- L = max(0, oS.alphas[j] - oS.alphas[i])
- H = min(oS.C, oS.C + oS.alphas[j] - oS.alphas[i])
- else:
- L = max(0, oS.alphas[j] + oS.alphas[i] - oS.C)
- H = min(oS.C, oS.alphas[j] + oS.alphas[i])
- if L==H: print "L==H"; return 0
- eta = 2.0 * oS.X[i,:]*oS.X[j,:].T - oS.X[i,:]*oS.X[i,:].T - oS.X[j,:]*oS.X[j,:].T
- if eta >= 0: print "eta>=0"; return 0
- oS.alphas[j] -= oS.labelMat[j]*(Ei - Ej)/eta
- oS.alphas[j] = clipAlpha(oS.alphas[j],H,L)
- updateEk(oS, j) #added this for the Ecache
- if (abs(oS.alphas[j] - alphaJold) < 0.00001): print "j not moving enough"; return 0
- oS.alphas[i] += oS.labelMat[j]*oS.labelMat[i]*(alphaJold - oS.alphas[j])#update i by the same amount as j
- updateEk(oS, i) #added this for the Ecache #the update is in the oppostie direction
- b1 = oS.b - Ei- oS.labelMat[i]*(oS.alphas[i]-alphaIold)*oS.X[i,:]*oS.X[i,:].T - oS.labelMat[j]*(oS.alphas[j]-alphaJold)*oS.X[i,:]*oS.X[j,:].T
- b2 = oS.b - Ej- oS.labelMat[i]*(oS.alphas[i]-alphaIold)*oS.X[i,:]*oS.X[j,:].T - oS.labelMat[j]*(oS.alphas[j]-alphaJold)*oS.X[j,:]*oS.X[j,:].T
- if (0 < oS.alphas[i]) and (oS.C > oS.alphas[i]): oS.b = b1
- elif (0 < oS.alphas[j]) and (oS.C > oS.alphas[j]): oS.b = b2
- else: oS.b = (b1 + b2)/2.0
- return 1
- else: return 0
- def smoPK(dataMatIn, classLabels, C, toler, maxIter): #full Platt SMO
- oS = optStruct(mat(dataMatIn),mat(classLabels).transpose(),C,toler)
- iter = 0
- entireSet = True; alphaPairsChanged = 0
- while (iter < maxIter) and ((alphaPairsChanged > 0) or (entireSet)):
- alphaPairsChanged = 0
- if entireSet: #go over all
- for i in range(oS.m):
- alphaPairsChanged += innerL(i,oS)
- print "fullSet, iter: %d i:%d, pairs changed %d" % (iter,i,alphaPairsChanged)
- iter += 1
- else:#go over non-bound (railed) alphas
- nonBoundIs = nonzero((oS.alphas.A > 0) * (oS.alphas.A < C))[0]
- for i in nonBoundIs:
- alphaPairsChanged += innerL(i,oS)
- print "non-bound, iter: %d i:%d, pairs changed %d" % (iter,i,alphaPairsChanged)
- iter += 1
- if entireSet: entireSet = False #toggle entire set loop
- elif (alphaPairsChanged == 0): entireSet = True
- print "iteration number: %d" % iter
- return oS.b,oS.alphas
运行结果如(图八)所示:

(图八)
上面代码有兴趣的可以读读,用的话,建议使用libsvm。
参考文献:
[1]machine learning in action. PeterHarrington
[2] pattern recognition and machinelearning. Christopher M. Bishop
[3]machine learning.Andrew Ng
Adaboost也是一种原理简单,但很实用的有监督机器学习算法,它是daptive boosting的简称。说到boosting算法,就不得提一提bagging算法,他们两个都是把一些弱分类器组合起来来进行分类的方法,统称为集成方法(ensemble method),类似于投资,“不把鸡蛋放在一个篮子”,虽然每个弱分类器分类的不那么准确,但是如果把多个弱分类器组合起来可以得到相当不错的结果,另外要说的是集成方法还可以组合不同的分类器,而Adaboost和boosting算法的每个弱分类器的类型都一样的。他们两个不同的地方是:boosting的每个弱分类器组合起来的权重不一样,本节的Adaboost就是一个例子,而bagging的每个弱分类器的组合权重是相等,代表的例子就是random forest。Random forest的每个弱分类器是决策树,输出的类别有多个决策树分类的类别的众数决定。今天的主题是Adaboost,下面来看看Adaboost的工作原理:
既然Adaboost的每个弱分类器的类型都一样,那么怎么组织安排每个分类器呢?如(图一)所示:
(图一)
(图一)是Adaboost的原理示意图,左边矩形表示数据集,中间表示根据特征阈值来做分类,这样每一个弱分类器都类似于一个单节点的决策树,其实就是阈值判断而已,右边的三角形对每个弱分类器赋予一个权重,最后根据每个弱分类器的加权组合来判断总体类别。要注意一下数据集从上到下三个矩形内的直方图不一样,这表示每个样本的权重也发生了变化,样本权重的一开始初始化成相等的权重,然后根据弱分类器的错误率来调整每个弱分类器的全总alpha,如(图一)中的三角形所示,alpha 的计算如(公式一)所示:

(公式一)
从(公式一)中也能感觉出来,弱分类器权重alpha和弱分类器分类错误率epsilon成反比,如果不能看出反比关系,分子分母同时除以epsilon就可以了,而ln是单调函数。这很make sense,当然分类器的错误率越高,越不能器重它,它的权重就应该低。同样的道理,样本也要区分对待,样本的权重要用弱分类器权重来计算,其实也是间接靠分类错误率,如(公式二)所示:
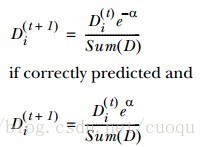
(公式二)
其中D表示样本权重向量,有多少个样本就有多少个权重,下标i表示样本索引,而上标t表示上一次分类器训练迭代次数。这样一直更新迭代,一直到最大迭代次数或者整个分类器错误率为0或者不变时停止迭代,就完成了Adaboost的训练。但是这样就可以把样本分开了吗?下面从一组图解答这个问题,如(图二)所示:

(图二)
由(图二)所示,每个弱分类器Hi可以要求不高的准确率,哪怕错误率是50%也可以接受,但是最后通过线性加权组合就可以得到一个很好的分类器,这点也可以通过错误率分析验证,有兴趣的可以看看:http://math.mit.edu/~rothvoss/18.304.3PM/Presentations/1-Eric-Boosting304FinalRpdf.pdf,想了解为什么alpha的计算如(公式一)的样子,可以看看:http://math.mit.edu/~rothvoss/18.304.3PM/Presentations/1-Eric-Boosting304FinalRpdf.pdf。
这样Adaboost的原理基本分析完毕,下面进入代码实战阶段:
首先来准备个简单数据集:
- from numpy import *
- def loadSimpData():
- datMat = matrix([[ 1. , 2.1],
- [ 2. , 1.1],
- [ 1.3, 1. ],
- [ 1. , 1. ],
- [ 2. , 1. ]])
- classLabels = [1.0, 1.0, -1.0, -1.0, 1.0]
- return datMat,classLabels
上面有5个样本,接下来就是初始化每个样本的权重,刚开始相等的:
- D = mat(ones((5,1))/5)
有了样本和初始化权重,接下来的任务就是构建一个弱分类器,其实就是一个单节点决策树,找到决策树每个特征维度上对应的最佳阈值以及表示是大于阈值还是小于阈值为正样本的标识符。代码如下:
- def buildStump(dataArr,classLabels,D):
- dataMatrix = mat(dataArr); labelMat = mat(classLabels).T
- m,n = shape(dataMatrix)
- numSteps = 10.0; bestStump = {}; bestClasEst = mat(zeros((m,1)))
- minError = inf #init error sum, to +infinity
- for i in range(n):#loop over all dimensions
- rangeMin = dataMatrix[:,i].min(); rangeMax = dataMatrix[:,i].max();
- stepSize = (rangeMax-rangeMin)/numSteps
- for j in range(-1,int(numSteps)+1):#loop over all range in current dimension
- for inequal in ['lt', 'gt']: #go over less than and greater than
- threshVal = (rangeMin + float(j) * stepSize)
- predictedVals = stumpClassify(dataMatrix,i,threshVal,inequal)#call stump classify with i, j, lessThan
- errArr = mat(ones((m,1)))
- errArr[predictedVals == labelMat] = 0
- weightedError = D.T*errArr #calc total error multiplied by D
- #print "split: dim %d, thresh %.2f, thresh ineqal: %s, the weighted error is %.3f" % (i, threshVal, inequal, weightedError)
- if weightedError < minError:
- minError = weightedError
- bestClasEst = predictedVals.copy()
- bestStump['dim'] = i
- bestStump['thresh'] = threshVal
- bestStump['ineq'] = inequal
- return bestStump,minError,bestClasEst
注意代码中有三个for循环,这三个for循环其实就是为了完成决策树的每个特征维度上对应的最佳阈值以及表示是大于阈值还是小于阈值为正样本的标识符,这三个要素。其中it,gt分别表示大于和小于,阈值的选择是靠增加步长来需找,最终三者的确定是靠决策树分类错误率最小者决定,每个决策树的分类代码如下,很简单,就是靠阈值判断:
- def stumpClassify(dataMatrix,dimen,threshVal,threshIneq):#just classify the data
- retArray = ones((shape(dataMatrix)[0],1))
- if threshIneq == 'lt':
- retArray[dataMatrix[:,dimen] <= threshVal] = -1.0
- else:
- retArray[dataMatrix[:,dimen] > threshVal] = -1.0
- return retArray
有了弱分类器的构造代码,下面来看Adaboost的训练代码:
- def adaBoostTrainDS(dataArr,classLabels,numIt=40):
- weakClassArr = []
- m = shape(dataArr)[0]
- D = mat(ones((m,1))/m) #init D to all equal
- aggClassEst = mat(zeros((m,1)))
- for i in range(numIt):
- bestStump,error,classEst = buildStump(dataArr,classLabels,D)#build Stump
- #print "D:",D.T
- alpha = float(0.5*log((1.0-error)/max(error,1e-16)))#calc alpha, throw in max(error,eps) to account for error=0
- bestStump['alpha'] = alpha
- weakClassArr.append(bestStump) #store Stump Params in Array
- #print "classEst: ",classEst.T
- expon = multiply(-1*alpha*mat(classLabels).T,classEst) #exponent for D calc, getting messy
- D = multiply(D,exp(expon)) #Calc New D for next iteration
- D = D/D.sum()
- #calc training error of all classifiers, if this is 0 quit for loop early (use break)
- aggClassEst += alpha*classEst
- #print "aggClassEst: ",aggClassEst.T
- aggErrors = multiply(sign(aggClassEst) != mat(classLabels).T,ones((m,1)))
- errorRate = aggErrors.sum()/m
- print "total error: ",errorRate
- if errorRate == 0.0: break
- return weakClassArr,aggClassEst
上面的代码中训练过程主要任务就是完成(公式二)中的样本权重D和弱分类器权重alpha的更新,另外还要注意一下,代码中迭代了40次,每次都调用了buildStump,这就意味着创建了40个弱分类器。当模型收敛后,有了样本权重和弱弱弱分类器权重,最后就是对测试样本进行分类,分类代码如下:
- def adaClassify(datToClass,classifierArr):
- dataMatrix = mat(datToClass)#do stuff similar to last aggClassEst in adaBoostTrainDS
- m = shape(dataMatrix)[0]
- aggClassEst = mat(zeros((m,1)))
- for i in range(len(classifierArr)):
- classEst = stumpClassify(dataMatrix,classifierArr[i]['dim'],\
- classifierArr[i]['thresh'],\
- classifierArr[i]['ineq'])#call stump classify
- aggClassEst += classifierArr[i]['alpha']*classEst
- print aggClassEst
- return sign(aggClassEst)
考虑到有些做学术的为了比较不同机器学习算法的好坏,常常需要画ROC曲线,这里也给出画ROC的代码:
- def plotROC(predStrengths, classLabels):
- import matplotlib.pyplot as plt
- cur = (1.0,1.0) #cursor
- ySum = 0.0 #variable to calculate AUC
- numPosClas = sum(array(classLabels)==1.0)
- yStep = 1/float(numPosClas); xStep = 1/float(len(classLabels)-numPosClas)
- sortedIndicies = predStrengths.argsort()#get sorted index, it's reverse
- fig = plt.figure()
- fig.clf()
- ax = plt.subplot(111)
- #loop through all the values, drawing a line segment at each point
- for index in sortedIndicies.tolist()[0]:
- if classLabels[index] == 1.0:
- delX = 0; delY = yStep;
- else:
- delX = xStep; delY = 0;
- ySum += cur[1]
- #draw line from cur to (cur[0]-delX,cur[1]-delY)
- ax.plot([cur[0],cur[0]-delX],[cur[1],cur[1]-delY], c='b')
- cur = (cur[0]-delX,cur[1]-delY)
- ax.plot([0,1],[0,1],'b--')
- plt.xlabel('False positive rate'); plt.ylabel('True positive rate')
- plt.title('ROC curve for AdaBoost horse colic detection system')
- ax.axis([0,1,0,1])
- plt.show()
- print "the Area Under the Curve is: ",ySum*xStep
到此位置,Adaboost的代码也介绍完了,最终程序的运行结果如(图三)所示:

(图三)
而Adaboost的模型ROC运行曲线如(图四)所示:
(图四)
最近MIT的几个人证明了Adaboost可以用一阶梯度的角度来解释,详见链接
参考文献:
[1] machinelearning in action.
[2] http://www.robots.ox.ac.uk/~az/lectures/cv/adaboost_matas.pdf
机器学习理论与实战(八)回归
按照《机器学习实战》的主线,结束有监督学习中关于分类的机器学习方法,进入回归部分。所谓回归就是数据进行曲线拟合,回归一般用来做预测,涵盖线性回归(经典最小二乘法)、局部加权线性回归、岭回归和逐步线性回归。先来看下线性回归,即经典最小二乘法,说到最小二乘法就不得说下线性代数,因为一般说线性回归只通过计算一个公式就可以得到答案,如(公式一)所示:
(公式一)
其中X是表示样本特征组成的矩阵,Y表示对应的值,比如房价,股票走势等,(公式一)是直接通过对(公式二)求导得到的,因为(公式二)是凸函数,导数等于零的点就是最小点。

(公式二)
不过并不是所有的码农能从(公式二)求导得到(公式一)的解,因此这里给出另外一个直观的解,直观理解建立起来后,后续几个回归就简单类推咯。从初中的投影点说起,如(图一)所示:

(图一)
在(图一)中直线a上离点b最近的点是点b在其上的投影,即垂直于a的交点p。p是b在a上的投影点。试想一下,如果我们把WX看成多维的a,即空间中的一个超面来代替二维空间中的直线,而y看成b,那现在要使得(公式二)最小是不是就是寻找(图一)中的e,即垂直于WX的垂线,因为只有垂直时e才最小。下面来看看如何通过寻找垂线并最终得到W。要寻找垂线,先从(图二)中的夹角theta 说起吧,因为当cos(theta)=0时,他们也就垂直了。下面来分析下直线或者向量之间的夹角,如(图二)所示:
(图二)
在(图二)中, 表示三角形
表示三角形
 的斜边,那么:
的斜边,那么:

角beta也可以得到同样的计算公式,接着利用三角形和差公式得到(公式三):

(公式三)
(公式三)表示的是两直线或者两向量之间的夹角公式,很多同学都学过。再仔细看下,发现分子其实是向量a,b之间的内积(点积),因此公式三变为简洁的(公式四)的样子:

(公式四)
接下来继续分析(图一)中的投影,为了方便观看,增加了一些提示如(图三)所示:

(图三)
在(图三)中,假设向量b在向量a中的投影为p(注意,这里都上升为向量空间,不再使用直线,因为(公式四)是通用的)。投影p和a 在同一方向上(也可以反方向),因此我们可以用一个系数乘上a来表示p,比如(图三)中的 ,有了投影向量p,那么我们就可以表示向量e,因为根据向量法则,
,有了投影向量p,那么我们就可以表示向量e,因为根据向量法则,
 ,有因为a和e垂直,因此
,有因为a和e垂直,因此
 ,展开求得系数x,如(公式五)所示:
,展开求得系数x,如(公式五)所示:
(公式五)
(公式五)是不是很像(公式一)?只不过公式一的分母写成了另外的形式,不过别急,现在的系数只是一个标量数字,因为a,b都是一个向量,我们要扩展一下,把a从向量扩展到子空间,因为(公式一)中的X是样本矩阵,矩阵有列空间和行空间,如(图四)所示:

(图四)
(图四)中的A表示样本矩阵X,假设它有两个列a1和a2,我们要找一些线性组合系数来找一个和(图三)一样的接受b 投影的向量,而这个向量通过矩阵列和系数的线性组合表示。求解的这个系数的思路和上面完全一样,就是寻找投影所在的向量和垂线e的垂直关系,得到系数,如(公式六)所示:

(公式六)
这下(公式六)和(公式一)完全一样了,基于最小二乘法的线性回归也就推导完成了,而局部加权回归其实只是相当于对不同样本之间的关系给出了一个权重,所以叫局部加权,如(公式七)所示:

(公式七)
而权重的计算可通过高斯核(高斯公式)来完成,核的作用就是做权重衰减,很多地方都要用到,表示样本的重要程度,一般离目标进的重要程度大些,高斯核可以很好的描述这种关系。如(公式八)所示,其中K是个超参数,根据情况灵活设置:

(公式八)
(图五)是当K分别为1.0, 0.01,0.003时的局部加权线性回归的样子,可以看出当K=1.0时,和线性回归没区别:
(图五)
而岭回归的样子如(公式九)所示:

(公式九)
岭回归主要是解决的问题就是当XX’无法求逆时,比如当特征很多,样本很少,矩阵X不是满秩矩阵,此时求逆会出错,但是通过加上一个对角为常量lambda的矩阵,就可以很巧妙的避免这个计算问题,因此会多一个参数lambda,lambda的最优选择由交叉验证(cross-validation)来决定,加上一个对角不为0的矩阵很形象的在对角上抬高了,因此称为岭。不同的lambda会使得系数缩减,如(图六)所示:

(图六)
说到系数缩减大家可能会觉得有奇怪,感觉有点类似于正则,但是这里只是相当于在(公式六)中增大分母,进而缩小系数,另外还有一些系数缩减的方法,比如直接增加一些约束,如(公式十)和(公式十一)所示:

(公式十)

(公式十一)
当线性回归增加了(公式十)的约束变得和桥回归差不多,系数缩减了,而如果增加了(公式十一)的约束时就是稀疏回归咯,(我自己造的名词,sorry),系数有一些0。
有了约束后,求解起来就不像上面那样直接计算个矩阵运算就行了,回顾第五节说中支持向量机原理,需要使用二次规划求解,不过仍然有一些像SMO算法一样的简化求解算法,比如前向逐步回归方法:
前向逐步回归的伪代码如(图七)所示,也不难,仔细阅读代码就可以理解:

(图七)
下面直接给出上面四种回归的代码:
- from numpy import *
- def loadDataSet(fileName): #general function to parse tab -delimited floats
- numFeat = len(open(fileName).readline().split('\t')) - 1 #get number of fields
- dataMat = []; labelMat = []
- fr = open(fileName)
- for line in fr.readlines():
- lineArr =[]
- curLine = line.strip().split('\t')
- for i in range(numFeat):
- lineArr.append(float(curLine[i]))
- dataMat.append(lineArr)
- labelMat.append(float(curLine[-1]))
- return dataMat,labelMat
- def standRegres(xArr,yArr):
- xMat = mat(xArr); yMat = mat(yArr).T
- xTx = xMat.T*xMat
- if linalg.det(xTx) == 0.0:
- print "This matrix is singular, cannot do inverse"
- return
- ws = xTx.I * (xMat.T*yMat)
- return ws
- def lwlr(testPoint,xArr,yArr,k=1.0):
- xMat = mat(xArr); yMat = mat(yArr).T
- m = shape(xMat)[0]
- weights = mat(eye((m)))
- for j in range(m): #next 2 lines create weights matrix
- diffMat = testPoint - xMat[j,:] #
- weights[j,j] = exp(diffMat*diffMat.T/(-2.0*k**2))
- xTx = xMat.T * (weights * xMat)
- if linalg.det(xTx) == 0.0:
- print "This matrix is singular, cannot do inverse"
- return
- ws = xTx.I * (xMat.T * (weights * yMat))
- return testPoint * ws
- def lwlrTest(testArr,xArr,yArr,k=1.0): #loops over all the data points and applies lwlr to each one
- m = shape(testArr)[0]
- yHat = zeros(m)
- for i in range(m):
- yHat[i] = lwlr(testArr[i],xArr,yArr,k)
- return yHat
- def lwlrTestPlot(xArr,yArr,k=1.0): #same thing as lwlrTest except it sorts X first
- yHat = zeros(shape(yArr)) #easier for plotting
- xCopy = mat(xArr)
- xCopy.sort(0)
- for i in range(shape(xArr)[0]):
- yHat[i] = lwlr(xCopy[i],xArr,yArr,k)
- return yHat,xCopy
- def rssError(yArr,yHatArr): #yArr and yHatArr both need to be arrays
- return ((yArr-yHatArr)**2).sum()
- def ridgeRegres(xMat,yMat,lam=0.2):
- xTx = xMat.T*xMat
- denom = xTx + eye(shape(xMat)[1])*lam
- if linalg.det(denom) == 0.0:
- print "This matrix is singular, cannot do inverse"
- return
- ws = denom.I * (xMat.T*yMat)
- return ws
- def ridgeTest(xArr,yArr):
- xMat = mat(xArr); yMat=mat(yArr).T
- yMean = mean(yMat,0)
- yMat = yMat - yMean #to eliminate X0 take mean off of Y
- #regularize X's
- xMeans = mean(xMat,0) #calc mean then subtract it off
- xVar = var(xMat,0) #calc variance of Xi then divide by it
- xMat = (xMat - xMeans)/xVar
- numTestPts = 30
- wMat = zeros((numTestPts,shape(xMat)[1]))
- for i in range(numTestPts):
- ws = ridgeRegres(xMat,yMat,exp(i-10))
- wMat[i,:]=ws.T
- return wMat
- def regularize(xMat):#regularize by columns
- inMat = xMat.copy()
- inMeans = mean(inMat,0) #calc mean then subtract it off
- inVar = var(inMat,0) #calc variance of Xi then divide by it
- inMat = (inMat - inMeans)/inVar
- return inMat
- def stageWise(xArr,yArr,eps=0.01,numIt=100):
- xMat = mat(xArr); yMat=mat(yArr).T
- yMean = mean(yMat,0)
- yMat = yMat - yMean #can also regularize ys but will get smaller coef
- xMat = regularize(xMat)
- m,n=shape(xMat)
- #returnMat = zeros((numIt,n)) #testing code remove
- ws = zeros((n,1)); wsTest = ws.copy(); wsMax = ws.copy()
- for i in range(numIt):
- print ws.T
- lowestError = inf;
- for j in range(n):
- for sign in [-1,1]:
- wsTest = ws.copy()
- wsTest[j] += eps*sign
- yTest = xMat*wsTest
- rssE = rssError(yMat.A,yTest.A)
- if rssE < lowestError:
- lowestError = rssE
- wsMax = wsTest
- ws = wsMax.copy()
- #returnMat[i,:]=ws.T
- #return returnMat
- #def scrapePage(inFile,outFile,yr,numPce,origPrc):
- # from BeautifulSoup import BeautifulSoup
- # fr = open(inFile); fw=open(outFile,'a') #a is append mode writing
- # soup = BeautifulSoup(fr.read())
- # i=1
- # currentRow = soup.findAll('table', r="%d" % i)
- # while(len(currentRow)!=0):
- # title = currentRow[0].findAll('a')[1].text
- # lwrTitle = title.lower()
- # if (lwrTitle.find('new') > -1) or (lwrTitle.find('nisb') > -1):
- # newFlag = 1.0
- # else:
- # newFlag = 0.0
- # soldUnicde = currentRow[0].findAll('td')[3].findAll('span')
- # if len(soldUnicde)==0:
- # print "item #%d did not sell" % i
- # else:
- # soldPrice = currentRow[0].findAll('td')[4]
- # priceStr = soldPrice.text
- # priceStr = priceStr.replace('$','') #strips out $
- # priceStr = priceStr.replace(',','') #strips out ,
- # if len(soldPrice)>1:
- # priceStr = priceStr.replace('Free shipping', '') #strips out Free Shipping
- # print "%s\t%d\t%s" % (priceStr,newFlag,title)
- # fw.write("%d\t%d\t%d\t%f\t%s\n" % (yr,numPce,newFlag,origPrc,priceStr))
- # i += 1
- # currentRow = soup.findAll('table', r="%d" % i)
- # fw.close()
- from time import sleep
- import json
- import urllib2
- def searchForSet(retX, retY, setNum, yr, numPce, origPrc):
- sleep(10)
- myAPIstr = 'AIzaSyD2cR2KFyx12hXu6PFU-wrWot3NXvko8vY'
- searchURL = 'https://www.googleapis.com/shopping/search/v1/public/products?key=%s&country=US&q=lego+%d&alt=json' % (myAPIstr, setNum)
- pg = urllib2.urlopen(searchURL)
- retDict = json.loads(pg.read())
- for i in range(len(retDict['items'])):
- try:
- currItem = retDict['items'][i]
- if currItem['product']['condition'] == 'new':
- newFlag = 1
- else: newFlag = 0
- listOfInv = currItem['product']['inventories']
- for item in listOfInv:
- sellingPrice = item['price']
- if sellingPrice > origPrc * 0.5:
- print "%d\t%d\t%d\t%f\t%f" % (yr,numPce,newFlag,origPrc, sellingPrice)
- retX.append([yr, numPce, newFlag, origPrc])
- retY.append(sellingPrice)
- except: print 'problem with item %d' % i
- def setDataCollect(retX, retY):
- searchForSet(retX, retY, 8288, 2006, 800, 49.99)
- searchForSet(retX, retY, 10030, 2002, 3096, 269.99)
- searchForSet(retX, retY, 10179, 2007, 5195, 499.99)
- searchForSet(retX, retY, 10181, 2007, 3428, 199.99)
- searchForSet(retX, retY, 10189, 2008, 5922, 299.99)
- searchForSet(retX, retY, 10196, 2009, 3263, 249.99)
- def crossValidation(xArr,yArr,numVal=10):
- m = len(yArr)
- indexList = range(m)
- errorMat = zeros((numVal,30))#create error mat 30columns numVal rows
- for i in range(numVal):
- trainX=[]; trainY=[]
- testX = []; testY = []
- random.shuffle(indexList)
- for j in range(m):#create training set based on first 90% of values in indexList
- if j < m*0.9:
- trainX.append(xArr[indexList[j]])
- trainY.append(yArr[indexList[j]])
- else:
- testX.append(xArr[indexList[j]])
- testY.append(yArr[indexList[j]])
- wMat = ridgeTest(trainX,trainY) #get 30 weight vectors from ridge
- for k in range(30):#loop over all of the ridge estimates
- matTestX = mat(testX); matTrainX=mat(trainX)
- meanTrain = mean(matTrainX,0)
- varTrain = var(matTrainX,0)
- matTestX = (matTestX-meanTrain)/varTrain #regularize test with training params
- yEst = matTestX * mat(wMat[k,:]).T + mean(trainY)#test ridge results and store
- errorMat[i,k]=rssError(yEst.T.A,array(testY))
- #print errorMat[i,k]
- meanErrors = mean(errorMat,0)#calc avg performance of the different ridge weight vectors
- minMean = float(min(meanErrors))
- bestWeights = wMat[nonzero(meanErrors==minMean)]
- #can unregularize to get model
- #when we regularized we wrote Xreg = (x-meanX)/var(x)
- #we can now write in terms of x not Xreg: x*w/var(x) - meanX/var(x) +meanY
- xMat = mat(xArr); yMat=mat(yArr).T
- meanX = mean(xMat,0); varX = var(xMat,0)
- unReg = bestWeights/varX
- print "the best model from Ridge Regression is:\n",unReg
- print "with constant term: ",-1*sum(multiply(meanX,unReg)) + mean(yMat)
以上各种回归方法没有考虑实际数据的噪声,如果噪声很多,直接用上述的回归不是太好,因此需要加上正则,然后迭代更新权重
参考文献:
[1] machine learning in action.Peter Harrington
[2]Linear Algebra and Its Applications_4ed.Gilbert_Strang
前一节的回归是一种全局回归模型,它设定了一个模型,不管是线性还是非线性的模型,然后拟合数据得到参数,现实中会有些数据很复杂,肉眼几乎看不出符合那种模型,因此构建全局的模型就有点不合适。这节介绍的树回归就是为了解决这类问题,它通过构建决策节点把数据数据切分成区域,然后局部区域进行回归拟合。先来看看分类回归树吧(CART:Classification And Regression Trees),这个模型优点就是上面所说,可以对复杂和非线性的数据进行建模,缺点是得到的结果不容易理解。顾名思义它可以做分类也可以做回归,至于分类前面在说决策树时已经说过了,这里略过。直接通过分析回归树的代码来理解吧:
- from numpy import *
- def loadDataSet(fileName): #general function to parse tab -delimited floats
- dataMat = [] #assume last column is target value
- fr = open(fileName)
- for line in fr.readlines():
- curLine = line.strip().split('\t')
- fltLine = map(float,curLine) #map all elements to float()
- dataMat.append(fltLine)
- return dataMat
- def binSplitDataSet(dataSet, feature, value):
- mat0 = dataSet[nonzero(dataSet[:,feature] > value)[0],:][0]
- mat1 = dataSet[nonzero(dataSet[:,feature] <= value)[0],:][0]
- return mat0,mat1
上面两个函数,第一个函数加载样本数据,第二个函数用来指定在某个特征和维度上切分数据,示例如(图一)所示:

(图一)
注意一下,CART是一种通过二元切分来构建树的,前面的决策树的构建是通过香农熵最小作为度量,树的节点是个离散的阈值;这里不再使用香农熵,因为我们要做回归,因此这里使用计算分割数据的方差作为度量,而树的节点也对应使用使得方差最小的某个连续数值(其实是特征值)。试想一下,如果方差越小,说明误差那个节点最能表述那块数据。下面来看看树的构建代码:
- def createTree(dataSet, leafType=regLeaf, errType=regErr, ops=(1,4)):#assume dataSet is NumPy Mat so we can array filtering
- feat, val = chooseBestSplit(dataSet, leafType, errType, ops)#choose the best split
- if feat == None: return val #if the splitting hit a stop condition return val(叶子节点值)
- retTree = {}
- retTree['spInd'] = feat
- retTree['spVal'] = val
- lSet, rSet = binSplitDataSet(dataSet, feat, val)
- retTree['left'] = createTree(lSet, leafType, errType, ops)
- retTree['right'] = createTree(rSet, leafType, errType, ops)
- return retTree
这段代码中主要工作任务就是选择最佳分割特征,然后分割,是叶子节点就返回,不是叶子节点就递归的生成树结构。其中调用了最佳分割特征的函数:chooseBestSplit,前面决策树的构建中,这个函数里用熵来度量,这里采用误差(方差)来度量,同样先看代码:
- def chooseBestSplit(dataSet, leafType=regLeaf, errType=regErr, ops=(1,4)):
- tolS = ops[0]; tolN = ops[1]
- #if all the target variables are the same value: quit and return value
- if len(set(dataSet[:,-1].T.tolist()[0])) == 1: #exit cond 1
- return None, leafType(dataSet)
- m,n = shape(dataSet)
- #the choice of the best feature is driven by Reduction in RSS error from mean
- S = errType(dataSet)
- bestS = inf; bestIndex = 0; bestValue = 0
- for featIndex in range(n-1):
- for splitVal in set(dataSet[:,featIndex]):
- mat0, mat1 = binSplitDataSet(dataSet, featIndex, splitVal)
- if (shape(mat0)[0] < tolN) or (shape(mat1)[0] < tolN): continue
- newS = errType(mat0) + errType(mat1)
- if newS < bestS:
- bestIndex = featIndex
- bestValue = splitVal
- bestS = newS
- #if the decrease (S-bestS) is less than a threshold don't do the split
- if (S - bestS) < tolS:
- return None, leafType(dataSet) #exit cond 2
- mat0, mat1 = binSplitDataSet(dataSet, bestIndex, bestValue)
- if (shape(mat0)[0] < tolN) or (shape(mat1)[0] < tolN): #exit cond 3
- return None, leafType(dataSet)
- return bestIndex,bestValue#returns the best feature to split on
- #and the value used for that split
这段代码的主干是:
遍历每个特征:
遍历每个特征值:
把数据集切分成两份
计算此时的切分误差
如果切分误差小于当前最小误差,更新最小误差值,当前切分为最佳切分
返回最佳切分的特征值和阈值
尤其注意最后的返回值,因为它是构建树每个节点成分的东西。另外代码中errType=regErr 调用了regErr函数来计算方差,下面给出:
- def regErr(dataSet):
- return var(dataSet[:,-1]) * shape(dataSet)[0]
如果误差变化不大时(代码中(S - bestS)),则生成叶子节点,叶子节点函数是:
- def regLeaf(dataSet):#returns the value used for each leaf
- return mean(dataSet[:,-1])
这样回归树构建的代码就初步分析完毕了,运行结果如(图二)所示:

(图二)
数据ex00.txt在文章最后给出,它的分布如(图三)所示:

(图三)
根据(图三),我们可以大概看出(图二)的代码的运行结果具有一定的合理性,选用X(用0表示)特征作为分割特征,然后左右节点各选了一个中心值来描述树回归。节点比较少,但很能说明问题,下面给出一个比较复杂数据跑出的结果,如(图四)所示:
(图四)
对应的数据如(图五)所示:
(图五)
对于树的叶子节点和节点值的合理性,大家逐个对照(图五)来验证吧。下面简单的说下树的修剪,如果特征维度比较高,很容易发生节点过多,造成过拟合,过拟合(overfit)会出现high variance, 而欠拟合(under fit)会出现high bias,这点是题外话,因为机器学习理论一般要讲这些,当出现过拟合时,一般使用正则方法,由于回归树没有建立目标函数,因此这里解决过拟合的方法就是修剪树,简单的说就是使用少量的、关键的特征来判别,下面来看看如何修剪树:很简单,就是递归的遍历一个子树,从叶子节点开始,计算同一父节点的两个子节点合并后的误差,再计算不合并的误差,如果合并会降低误差,就把叶子节点合并。说到误差,其实前面的chooseBestSplit函数里有一句代码:
- #if the decrease (S-bestS) is less than a threshold don't do the split
- if (S - bestS) < tolS:
tolS 是个阈值,当误差变化不太大时,就不再分裂下去,其实也是修剪树的方法,只不过它是事前修剪,而计算合并误差的则是事后修剪。下面是其代码:
- def getMean(tree):
- if isTree(tree['right']): tree['right'] = getMean(tree['right'])
- if isTree(tree['left']): tree['left'] = getMean(tree['left'])
- return (tree['left']+tree['right'])/2.0
- def prune(tree, testData):
- if shape(testData)[0] == 0: return getMean(tree) #if we have no test data collapse the tree
- if (isTree(tree['right']) or isTree(tree['left'])):#if the branches are not trees try to prune them
- lSet, rSet = binSplitDataSet(testData, tree['spInd'], tree['spVal'])
- if isTree(tree['left']): tree['left'] = prune(tree['left'], lSet)
- if isTree(tree['right']): tree['right'] = prune(tree['right'], rSet)
- #if they are now both leafs, see if we can merge them
- if not isTree(tree['left']) and not isTree(tree['right']):
- lSet, rSet = binSplitDataSet(testData, tree['spInd'], tree['spVal'])
- errorNoMerge = sum(power(lSet[:,-1] - tree['left'],2)) +\
- sum(power(rSet[:,-1] - tree['right'],2))
- treeMean = (tree['left']+tree['right'])/2.0
- errorMerge = sum(power(testData[:,-1] - treeMean,2))
- if errorMerge < errorNoMerge:
- print "merging"
- return treeMean
- else: return tree
- else: return tree
说完了树回归,再简单的提下模型树,因为树回归每个节点是一些特征和特征值,选取的原则是根据特征方差最小。如果把叶子节点换成分段线性函数,那么就变成了模型树,如(图六)所示:

(图六)
(图六)中明显是两个直线组成,以X坐标(0.0-0.3)和(0.3-1.0)分成的两个线段。如果我们用两个叶子节点保存两个线性回归模型,就完成了这部分数据的拟合。实现也比较简单,代码如下:
- def linearSolve(dataSet): #helper function used in two places
- m,n = shape(dataSet)
- X = mat(ones((m,n))); Y = mat(ones((m,1)))#create a copy of data with 1 in 0th postion
- X[:,1:n] = dataSet[:,0:n-1]; Y = dataSet[:,-1]#and strip out Y
- xTx = X.T*X
- if linalg.det(xTx) == 0.0:
- raise NameError('This matrix is singular, cannot do inverse,\n\
- try increasing the second value of ops')
- ws = xTx.I * (X.T * Y)
- return ws,X,Y
- def modelLeaf(dataSet):#create linear model and return coeficients
- ws,X,Y = linearSolve(dataSet)
- return ws
- def modelErr(dataSet):
- ws,X,Y = linearSolve(dataSet)
- yHat = X * ws
- return sum(power(Y - yHat,2))
代码和树回归相似,只不过modelLeaf在返回叶子节点时,要完成一个线性回归,由linearSolve来完成。最后一个函数modelErr则和回归树的regErr函数起着同样的作用。
谢天谢地,这篇文章一个公式都没有出现,但同时也希望没有数学的语言,表述会清楚。
数据ex00.txt:
0.036098 0.155096
0.993349 1.077553
0.530897 0.893462
0.712386 0.564858
0.343554 -0.371700
0.098016 -0.332760
0.691115 0.834391
0.091358 0.099935
0.727098 1.000567
0.951949 0.945255
0.768596 0.760219
0.541314 0.893748
0.146366 0.034283
0.673195 0.915077
0.183510 0.184843
0.339563 0.206783
0.517921 1.493586
0.703755 1.101678
0.008307 0.069976
0.243909 -0.029467
0.306964 -0.177321
0.036492 0.408155
0.295511 0.002882
0.837522 1.229373
0.202054 -0.087744
0.919384 1.029889
0.377201 -0.243550
0.814825 1.095206
0.611270 0.982036
0.072243 -0.420983
0.410230 0.331722
0.869077 1.114825
0.620599 1.334421
0.101149 0.068834
0.820802 1.325907
0.520044 0.961983
0.488130 -0.097791
0.819823 0.835264
0.975022 0.673579
0.953112 1.064690
0.475976 -0.163707
0.273147 -0.455219
0.804586 0.924033
0.074795 -0.349692
0.625336 0.623696
0.656218 0.958506
0.834078 1.010580
0.781930 1.074488
0.009849 0.056594
0.302217 -0.148650
0.678287 0.907727
0.180506 0.103676
0.193641 -0.327589
0.343479 0.175264
0.145809 0.136979
0.996757 1.035533
0.590210 1.336661
0.238070 -0.358459
0.561362 1.070529
0.377597 0.088505
0.099142 0.025280
0.539558 1.053846
0.790240 0.533214
0.242204 0.209359
0.152324 0.132858
0.252649 -0.055613
0.895930 1.077275
0.133300 -0.223143
0.559763 1.253151
0.643665 1.024241
0.877241 0.797005
0.613765 1.621091
0.645762 1.026886
0.651376 1.315384
0.697718 1.212434
0.742527 1.087056
0.901056 1.055900
0.362314 -0.556464
0.948268 0.631862
0.000234 0.060903
0.750078 0.906291
0.325412 -0.219245
0.726828 1.017112
0.348013 0.048939
0.458121 -0.061456
0.280738 -0.228880
0.567704 0.969058
0.750918 0.748104
0.575805 0.899090
0.507940 1.107265
0.071769 -0.110946
0.553520 1.391273
0.401152 -0.121640
0.406649 -0.366317
0.652121 1.004346
0.347837 -0.153405
0.081931 -0.269756
0.821648 1.280895
0.048014 0.064496
0.130962 0.184241
0.773422 1.125943
0.789625 0.552614
0.096994 0.227167
0.625791 1.244731
0.589575 1.185812
0.323181 0.180811
0.822443 1.086648
0.360323 -0.204830
0.950153 1.022906
0.527505 0.879560
0.860049 0.717490
0.007044 0.094150
0.438367 0.034014
0.574573 1.066130
0.536689 0.867284
0.782167 0.886049
0.989888 0.744207
0.761474 1.058262
0.985425 1.227946
0.132543 -0.329372
0.346986 -0.150389
0.768784 0.899705
0.848921 1.170959
0.449280 0.069098
0.066172 0.052439
0.813719 0.706601
0.661923 0.767040
0.529491 1.022206
0.846455 0.720030
0.448656 0.026974
0.795072 0.965721
0.118156 -0.077409
0.084248 -0.019547
0.845815 0.952617
0.576946 1.234129
0.772083 1.299018
0.696648 0.845423
0.595012 1.213435
0.648675 1.287407
0.897094 1.240209
0.552990 1.036158
0.332982 0.210084
0.065615 -0.306970
0.278661 0.253628
0.773168 1.140917
0.203693 -0.064036
0.355688 -0.119399
0.988852 1.069062
0.518735 1.037179
0.514563 1.156648
0.976414 0.862911
0.919074 1.123413
0.697777 0.827805
0.928097 0.883225
0.900272 0.996871
0.344102 -0.061539
0.148049 0.204298
0.130052 -0.026167
0.302001 0.317135
0.337100 0.026332
0.314924 -0.001952
0.269681 -0.165971
0.196005 -0.048847
0.129061 0.305107
0.936783 1.026258
0.305540 -0.115991
0.683921 1.414382
0.622398 0.766330
0.902532 0.861601
0.712503 0.933490
0.590062 0.705531
0.723120 1.307248
0.188218 0.113685
0.643601 0.782552
0.520207 1.209557
0.233115 -0.348147
0.465625 -0.152940
0.884512 1.117833
0.663200 0.701634
0.268857 0.073447
0.729234 0.931956
0.429664 -0.188659
0.737189 1.200781
0.378595 -0.296094
0.930173 1.035645
0.774301 0.836763
0.273940 -0.085713
0.824442 1.082153
0.626011 0.840544
0.679390 1.307217
0.578252 0.921885
0.785541 1.165296
0.597409 0.974770
0.014083 -0.132525
0.663870 1.187129
0.552381 1.369630
0.683886 0.999985
0.210334 -0.006899
0.604529 1.212685
0.250744 0.046297
接下来就要说下无监督机器学习方法,所谓无监督机器学习前面也说过,就是没有标签的情况,对样本数据进行聚类分析、关联性分析等。主要包括K均值聚类(K-means clustering)和关联分析,这两大类都可以说的很简单也可以说的很复杂,学术的东西本身就一直在更新着。比如K均值聚类可以扩展一下形成层次聚类(Hierarchical Clustering),也可以进入概率分布的空间进行聚类,就像前段时间很火的LDA聚类,虽然最近深度玻尔兹曼机(DBM)打败了它,但它也是自然语言处理领域(NLP:Natural Language Processing)的一个有力工具,有过辉煌的一段故事。而关联性分析又是另外一个比较有力的工具,它又称购物篮分析,我们可以大概可以体会到它的用途,挖掘目标之间的关联性,经典的故事就是啤酒和尿布的关联。另外多说一下,google最近的两大核心技术:深度学习和知识图,深度学习就不说了,而知识图就是挖掘关系的。找到了关系就找到了金矿,关系也可以用复杂网络(complex network)来建模。这些话题就打住吧,今天就来说下K均值聚类和二分K均值聚类。
K均值聚类比较简单,再说原理之前,先来看个样本图,如(图一)所示:
(图一)
假如(图一)中是我们的样本数据,每个样本都没有类别,我们想对他们简单的划下类,在(图一)中明显的有四“堆”数据,我们用什么方法能把他们分成四类呢?K均值聚类就是解决这种问题的(好腻 = =!),K均值聚类的原理如下:
随机的分配K个质心(上图中K为4)
如果样本中任意一个数据点归属的簇号(堆类别)发生改变
遍历每一个样本点
遍历每一个质心
计算数据点到质心的距离
把数据点分配到距其最近的簇
对每一个簇,重新计算簇中所有样本点的均值作为质心
K均值简单的一句话总结就是:更新质心,更新每个样本的所属的类别。按照上述更新规则,当没有样本的簇号发生改变了,迭代也就终止咯。下面就来看看代码吧:
- from numpy import *
- def loadDataSet(fileName): #general function to parse tab -delimited floats
- dataMat = [] #assume last column is target value
- fr = open(fileName)
- for line in fr.readlines():
- curLine = line.strip().split('\t')
- fltLine = map(float,curLine) #map all elements to float()
- dataMat.append(fltLine)
- return dataMat
- def distEclud(vecA, vecB):
- return sqrt(sum(power(vecA - vecB, 2))) #la.norm(vecA-vecB)
- def randCent(dataSet, k):
- n = shape(dataSet)[1]
- centroids = mat(zeros((k,n)))#create centroid mat
- for j in range(n):#create random cluster centers, within bounds of each dimension
- minJ = min(dataSet[:,j])
- rangeJ = float(max(dataSet[:,j]) - minJ)
- centroids[:,j] = mat(minJ + rangeJ * random.rand(k,1))
- return centroids
- def kMeans(dataSet, k, distMeas=distEclud, createCent=randCent):
- m = shape(dataSet)[0]
- clusterAssment = mat(zeros((m,2)))#create mat to assign data points
- #to a centroid, also holds SE of each point
- centroids = createCent(dataSet, k)
- clusterChanged = True
- while clusterChanged:
- clusterChanged = False
- for i in range(m):#for each data point assign it to the closest centroid
- minDist = inf; minIndex = -1
- for j in range(k):
- distJI = distMeas(centroids[j,:],dataSet[i,:])
- if distJI < minDist:
- minDist = distJI; minIndex = j
- if clusterAssment[i,0] != minIndex: clusterChanged = True
- clusterAssment[i,:] = minIndex,minDist**2
- print centroids
- for cent in range(k):#recalculate centroids
- ptsInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]]#get all the point in this cluster
- centroids[cent,:] = mean(ptsInClust, axis=0) #assign centroid to mean
- return centroids, clusterAssment
代码也很简单,其中函数loadDataSet用来加载数据集,函数distEclud用来计算两个样本的距离,函数randCent为样本随机的分配K个质心(centroid),另外注意一下样本的质心维度和样本维度是一样的,这个应该没有异议,高维空间中的坐标,最后函数kMeans则是k均值聚类算法的核心步骤,和原理都是一一对应的。下面是运行结果:

(图二)
从(图二)可以看出质心的更新,不过按照k-means的聚类规则很容易陷入局部最小,陷入局部最小说简单的就是随机初始的质心分布的不是太好,最后迭代终止后,两个质心有可能在同一堆数据上,而另外一个质心成了另外两堆离的近样本的唯一质心(如下面图三所示)。说的复杂一些就是马尔科夫岁机场中配置的代价函数不是好的目标函数(虽然这里没写出这个函数)。为了解决这个问题,有人提出了二分K均值聚类算法,该算法也比较简单,首先把所有样本作为一个簇,然后二分该簇,接着选择其中一个簇进行继续进行二分。选择哪一个簇二分的原则就是能否使得误差平方和(SSE: Sum of Squared Error)进可能的小。也就是说该算法有了个好的目标函数,SSE的计算其实就是距离和。下面来看看二分K均值聚类算法的代码:
- def biKmeans(dataSet, k, distMeas=distEclud):
- m = shape(dataSet)[0]
- clusterAssment = mat(zeros((m,2)))
- centroid0 = mean(dataSet, axis=0).tolist()[0]
- centList =[centroid0] #create a list with one centroid
- for j in range(m):#calc initial Error
- clusterAssment[j,1] = distMeas(mat(centroid0), dataSet[j,:])**2
- while (len(centList) < k):
- lowestSSE = inf
- for i in range(len(centList)):
- ptsInCurrCluster = dataSet[nonzero(clusterAssment[:,0].A==i)[0],:]#get the data points currently in cluster i
- centroidMat, splitClustAss = kMeans(ptsInCurrCluster, 2, distMeas)
- sseSplit = sum(splitClustAss[:,1])#compare the SSE to the currrent minimum
- sseNotSplit = sum(clusterAssment[nonzero(clusterAssment[:,0].A!=i)[0],1])
- print "sseSplit, and notSplit: ",sseSplit,sseNotSplit
- if (sseSplit + sseNotSplit) < lowestSSE:
- bestCentToSplit = i
- bestNewCents = centroidMat
- bestClustAss = splitClustAss.copy()
- lowestSSE = sseSplit + sseNotSplit
- bestClustAss[nonzero(bestClustAss[:,0].A == 1)[0],0] = len(centList) #change 1 to 3,4, or whatever
- bestClustAss[nonzero(bestClustAss[:,0].A == 0)[0],0] = bestCentToSplit
- print 'the bestCentToSplit is: ',bestCentToSplit
- print 'the len of bestClustAss is: ', len(bestClustAss)
- centList[bestCentToSplit] = bestNewCents[0,:].tolist()[0]#replace a centroid with two best centroids
- centList.append(bestNewCents[1,:].tolist()[0])
- clusterAssment[nonzero(clusterAssment[:,0].A == bestCentToSplit)[0],:]= bestClustAss#reassign new clusters, and SSE
- return mat(centList), clusterAssment
这个算法比K均值聚类算法效果好一些,比如(图三)是K均值算法在随机初始化不好的情况下聚类的效果示意图:

(图三)
而用二分K均值聚类跑出的效果图如(图四)所示:

(图四)
到此为止,关于K均值的聚类算法也就说完了,虽然有二分K均值聚类算法改进了K均值聚类算法的不足,但也不是没缺点,它们的共同的缺点就是必须事先确定K的值,现实中的数据我们有可能不知道K的值,如何确定K的值也是学术界一直在研究的问题,现在常用的解决办法是用层次聚类(Hierarchical Clustering),或者借鉴下LDA中的话题聚类分析。最后多说一句:谱聚类也是图像中的一个大成员,用途很广,典型的就是图像分割。
机器学习理论与实战(十一)关联规则分析Apriori
《机器学习实战》的最后的两个算法对我来说有点陌生,但学过后感觉蛮好玩,了解了一般的商品数据关联分析和搜索引擎智能提示的工作原理。先来看看关联分析(association analysis)吧,它又称关联规则学习(association rule learning),它的主要工作就是快速找到经常在一起的频繁项,比如著名的“啤酒”和“尿布”。试想一下,给我们一堆交易数据,每次的交易数据中有不同的商品,要我们从中发掘哪些商品经常被一起购买?当然穷举法也可以解决,但是计算量很大,这节的算法Apriori就是解决此类问题的快速算法。Apriori在拉丁语中表示“from before”(来自以前)的意思,在这里就是来自于子集的频繁信息咯,不懂,别着急。下面给出几个简单的交易数据,如(表一)所示:
| 交易号 | 商品 | |||
| 0 | 豆奶 | 生菜 |
|
|
| 1 | 生菜 | 尿布 | 葡萄酒 | 甜菜 |
| 2 | 豆奶 | 尿布 | 葡萄酒 | 橙汁 |
| 3 | 生菜 | 豆奶 | 尿布 | 葡萄酒 |
| 4 | 生菜 | 豆奶 | 尿布 | 橙汁 |
(表一) 商品交易数据
我们的目标就是找到经常在一起出现的频繁子集,集合哦。我们用大括号“{}”来表示集合。为了表示某个子集是否是频繁子集,我们需要用一些量化方法,光计数也不行,因为不同量的交易数据出现的次数差别很大,一般用支持度(support)和置信度(confident)这两个指标来量化频繁子集、关联规则。这两个指标的计算都很简单:
支持度=(包含该子集的交易数目)/总交易数目
比如{豆奶}的支持度为4/5,有四条交易数据都有豆奶,而{豆奶,尿布}的支持度为3/5。
置信度只是针对像{尿布}->{葡萄酒}的关联规则来定义的:
尿布到葡萄酒的置信度=支持度({尿布,葡萄酒})/支持度(尿布)
比如在(表一)中,{尿布,葡萄酒}的支持度为3/5,而尿布的支持度为4/5,那么尿布->葡萄酒的可信度为3/4=0.75。
尽管有了量化指标,但是要让我们在大规模的数据中计算所有的组合的支持度和关联规则的支持度工作量也很大,如(图一)所示:
(图一) 商品{0,1,2,3}所有可能的组合
(图一)中只有四种商品,而其子集则有2^4-1=15个(空子集除外),每计算一个子集的两个指标都需要遍历一下数据,要遍历15次,如果有100种商品,则有1.26*10^30个子集,这个计算量很大,所幸的是研究人员发现了一种Apriori原理:Apriori原理是说如果某个子集不是频繁的,那么包含它的所有超集也不是频繁的,这样一下就砍掉了一大半,如(图二)所示:

(图二) Apriori原理
(图二)中{2,3}不是频繁的,那么所有包含它的超子集都不是频繁的,有了这个原则,就使得我们计算频繁子集的变成可行的。有了Apriori原理作为指导,我们还需要一些trick 来实现代码,这些trick就构成了Apriori算法:
先生成所有单个商品构成子集列表
然后计算每个子集的支持度
剔除不满足最小支持度的子集
接下来对满足要求的子集进行两两组合(但组合一定是某个交易的数据的子集)
然后再计算组合的支持度
再剔除,
依次按上述循环直到剔除完毕。
返回频繁子集
上述算法的伪代码如(图三)所示:

(图三)
下面直接给出代码,没有公式,就根据上面的伪代码,读者都可以尝试自己写代码:
- from numpy import *
- def loadDataSet():
- return [[1, 3, 4], [2, 3, 5], [1, 2, 3, 5], [2, 5]]
- def createC1(dataSet):
- C1 = []
- for transaction in dataSet:
- for item in transaction:
- if not [item] in C1:
- C1.append([item])
- C1.sort()
- return map(frozenset, C1)#use frozen set so we
- #can use it as a key in a dict
- def scanD(D, Ck, minSupport):
- ssCnt = {}
- for tid in D:
- for can in Ck:
- if can.issubset(tid):
- if not ssCnt.has_key(can): ssCnt[can]=1
- else: ssCnt[can] += 1
- numItems = float(len(D))
- retList = []
- supportData = {}
- for key in ssCnt:
- support = ssCnt[key]/numItems
- if support >= minSupport:
- retList.insert(0,key)
- supportData[key] = support
- return retList, supportData
- def aprioriGen(Lk, k): #creates Ck
- retList = []
- lenLk = len(Lk)
- for i in range(lenLk):
- for j in range(i+1, lenLk):
- L1 = list(Lk[i])[:k-2]; L2 = list(Lk[j])[:k-2]
- L1.sort(); L2.sort()
- if L1==L2: #if first k-2 elements are equal
- retList.append(Lk[i] | Lk[j]) #set union
- return retList
- def apriori(dataSet, minSupport = 0.5):
- C1 = createC1(dataSet)
- D = map(set, dataSet)
- L1, supportData = scanD(D, C1, minSupport)
- L = [L1]
- k = 2
- while (len(L[k-2]) > 0):
- Ck = aprioriGen(L[k-2], k)
- Lk, supK = scanD(D, Ck, minSupport)#scan DB to get Lk
- supportData.update(supK)
- L.append(Lk)
- k += 1
- return L, supportData
另外多说一点关于关联规则的挖掘,有了频繁子集,就是挖掘关联规则,和频繁子集一样的道理,只不过改计算置信度(confident),计算方法也同上,如(图四)所示:

(图四) 关联规则挖掘
(图四)和(图二)中的否定关系是反过来的,关联规则挖掘的原则是:如果某个关联规则不满足最小置信度,那么它的所有子集都要被舍去,都是不满足最小置信度的,这点并不是绝对的,读者可以根据自己的需求个性化修改。代码如下:
- def generateRules(L, supportData, minConf=0.7): #supportData is a dict coming from scanD
- bigRuleList = []
- for i in range(1, len(L)):#only get the sets with two or more items
- for freqSet in L[i]:
- H1 = [frozenset([item]) for item in freqSet]
- if (i > 1):
- rulesFromConseq(freqSet, H1, supportData, bigRuleList, minConf)
- else:
- calcConf(freqSet, H1, supportData, bigRuleList, minConf)
- return bigRuleList
- def calcConf(freqSet, H, supportData, brl, minConf=0.7):
- prunedH = [] #create new list to return
- for conseq in H:
- conf = supportData[freqSet]/supportData[freqSet-conseq] #calc confidence
- if conf >= minConf:
- print freqSet-conseq,'-->',conseq,'conf:',conf
- brl.append((freqSet-conseq, conseq, conf))
- prunedH.append(conseq)
- return prunedH
- def rulesFromConseq(freqSet, H, supportData, brl, minConf=0.7):
- m = len(H[0])
- if (len(freqSet) > (m + 1)): #try further merging
- Hmp1 = aprioriGen(H, m+1)#create Hm+1 new candidates
- Hmp1 = calcConf(freqSet, Hmp1, supportData, brl, minConf)
- if (len(Hmp1) > 1): #need at least two sets to merge
- rulesFromConseq(freqSet, Hmp1, supportData, brl, minConf)
- def pntRules(ruleList, itemMeaning):
- for ruleTup in ruleList:
- for item in ruleTup[0]:
- print itemMeaning[item]
- print " -------->"
- for item in ruleTup[1]:
- print itemMeaning[item]
- print "confidence: %f" % ruleTup[2]
- print #print a blank line
转载请注明来源: http://blog.csdn.net/marvin521/article/details/9751589
参考文献:
[1] machine learning in action.Peter Harrington












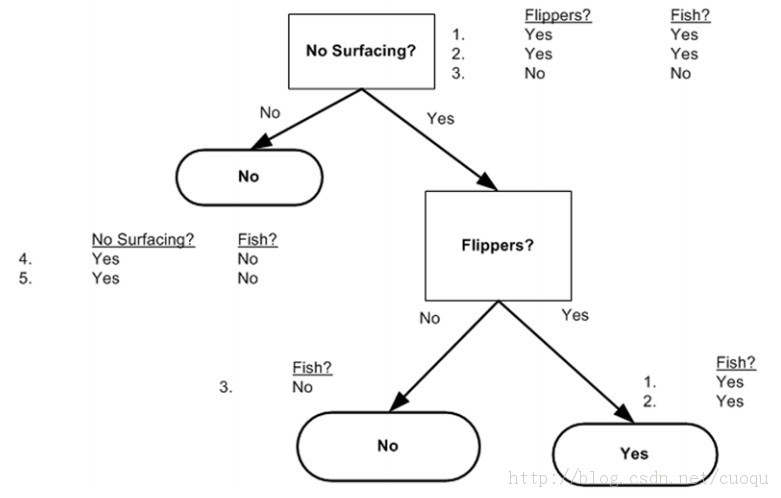


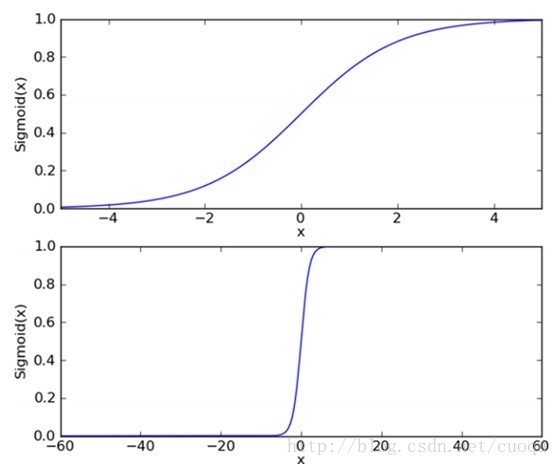

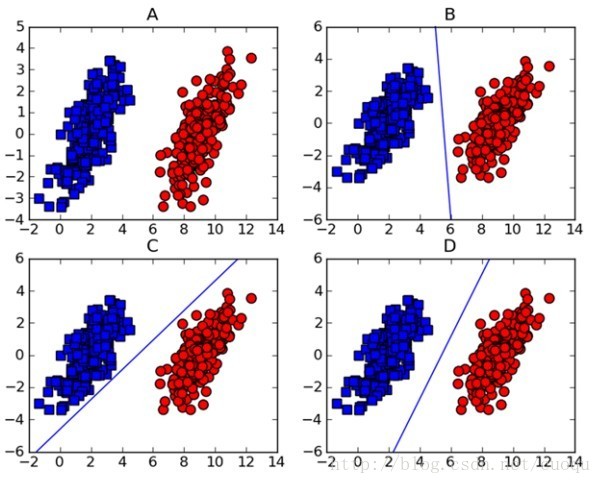
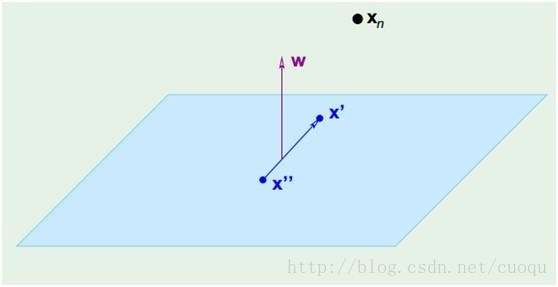
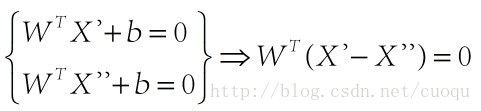




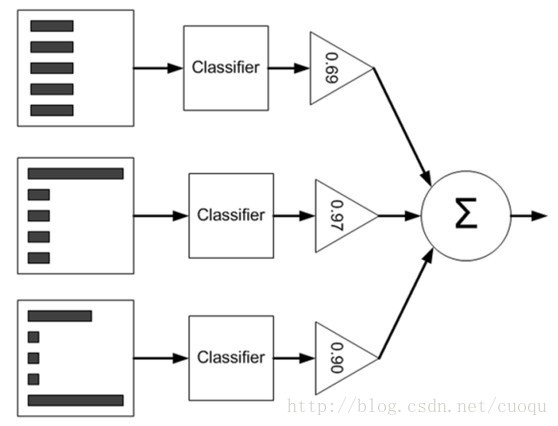








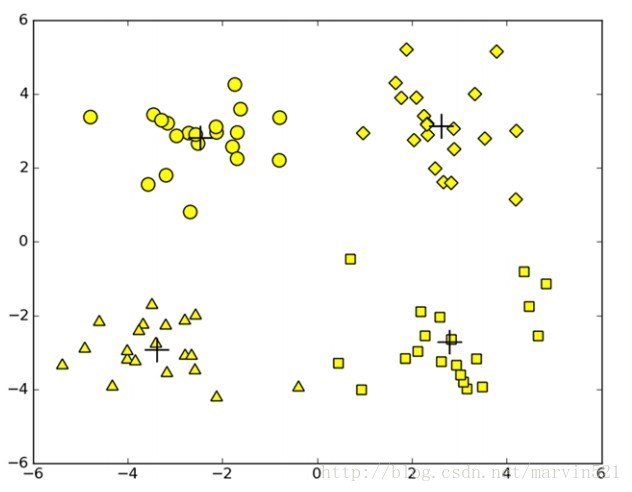















 5850
5850

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








