structural covariance network 结构协方差网络
结构协方差网络是一个较老的概念,只是近年受到了一定的重视。
大佬 Aaron Alexander-Bloch 在2013年通过一篇综述描述了这种结构协方差网络的应用意义及前景。
既往一般是在bold信号和fiber tracking建立连接,两者也都有研究基础。而皮层结构有那么多指标,例如厚度、体积等,因此也考虑利用起来,就自然而然发展出来了结构网络。
Aaron Alexander-Bloch 认为,某节点的皮层厚度能影响其功能连接或结构连接的节点的厚度。
我的理解:我认为这句话很重要,这里的结构连接指的是fiber network。即:假设某个节点变化,它其实是多维度同步变化的,例如连接它的白质纤维束减少,它的皮层厚度就会降低(突触减少,神经元萎缩),并且它的功能bold信号也会有变化(当然不一定是降低,functional network没办法评估功能活动度降低)。这种假设是符合逻辑也容易理解的,并且大概率也符合实际,所以这就构成了结构网络的基础。
那么问题来了,结构协方差网络是直接做pearson相关吗?
答案是不是。试想一下,以cortical thickness为例,假如跟functional connection 一样直接相关,那么这个应该叫cortical connection,而不是加一个covariance单词。这个单词是有确切含义的。当然,最近我看了几篇文献,也不乏几篇不错的二区杂志的文章用的就是cortical correlation network。
事实上,结构协方差网络,做的应该是偏相关,而不是简单相关,即需要加入协变量。当我们需要做基于种子点的结构协方差网络时,是需要回归掉其他所有ROI的影响,只留下这个种子点的效应。因为也有文献用全偏相关去计算结构协方差网络。这种做法会漏掉很多关键信息。诚然,简单相关会产生许多虚假连接,不过后面大家提出random network和small world,通过划分多个阈值来综合评价网络,这样可以减少因简单相关导致的虚假连接误差。所以类似的,结构协方差网络也不必使用全偏相关建立矩阵。
Aaron Alexander-Bloch在他的文献中指出了,建议纳入age和gender去做结构协方差网络,因为皮层厚度与性别和年龄的关系最大(其中年龄为主要因素),因此这样形成的结构协方差网络,就在多个数据集或者多个被试之间具有了可比性。这个我觉得是比较有道理的。
当然,那为啥functional connection不去与年龄性别回归,做一个functional covariance network呢?我认为有2个原因,一个是functional activity与年龄和性别的关系没有那么明确,至少目前来说没有那么明确,因为它毕竟是评价的两个节点协同性。二个是以前大家考虑的比较少,大部分研究结果都是基于此,因为也就延续下来,这样使结果具有可比性。
但是谁又能说一定就没有影响呢,万一得出了有意思的结论呢,科研就是不断的试错,functional covariance network & structural covariance network。当然也有人会说,那为啥fiber network不去做协方差?因为fiber理论上来说就是实际的纤维束数量,它是真实的物理存在,相比相关性分析,它的虚假连接要少很多,所以可以直接使用raw network。
以上都是自己看文献后的一点总结和所思所想。
更正:今天手撸了一套分析代码,才发现由于自己的思想幼稚,数据处理经验缺乏,导致思维太简单。
更正的地方是functional connect 是不能做方差的。因为functional connect是用两个ROI的230时间点做pearson相关,而age和gender只有被试的数量,所以每个被试的functional connect相关性矩阵不能与基础信息做协方差回归。而只有皮层数据可以,因为皮层不包含多个时间点。
(补之前挖下的坑)
结构协方差网络
首先用aparcstats2table获取所有被试的ROI的皮层指标(这里均以thickness为例)。
得到thickness:
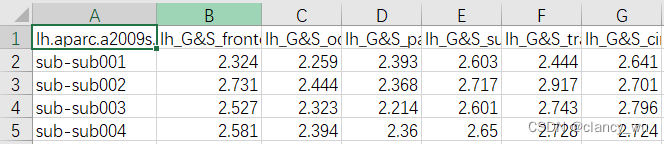
这样就是N个被试,每个被试148个ROI,形成table。
通过brainGraph构建结构协方差网络:
options(bg.subject_id='participant_id', bg.group='group')
data.l.file <- fread('aparc.a2009s_lh_thickness.csv')
data.r.file <- fread('aparc.a2009s_rh_thickness.csv')
data.l <- data.l.file[,2:75]; data.r <- data.r.file[, 2:75]
lhrh <- cbind(data.l, data.r)
# set new name for matching the atlas
library(stringr)
oldname <- colnames(lhrh)
newname <- str_replace_all(oldname, c('h_'='', '&'='_and_', '_thickness'='', '-'='.'))
colnames(lhrh) <- newname
# build structural covariance network
covars.all <- fread('subjects_71_info.csv')
covars.all[, gender := as.factor(gender)]
covars <- covars.all[,c('age', 'gender', 'group')]
covars.id <- paste0('sub-', covars.all$participant_id)
lhrh$participant_id <- covars$participant_id <- covars.id
myResids <- get.resid(lhrh, covars, atlas = 'destrieux')
densities <- seq(0.01, 0.34, 0.01)
corrs <- corr.matrix(myResids, densities=densities)
g <- lapply(seq_along(densities), function(x)
make_brainGraphList(corrs[x], modality='thickness'))
dt.G <- rbindlist(lapply(g, graph_attr_dt))
dt.V <- rbindlist(lapply(g, vertex_attr_dt))
一步一步来看这个代码:
首先是读取freesurfer生成的thickness,然后左右半球合并成一个文件。接下来是用get.resid函数得到myResids。plot(myResids,region=‘lS_calcarine’)
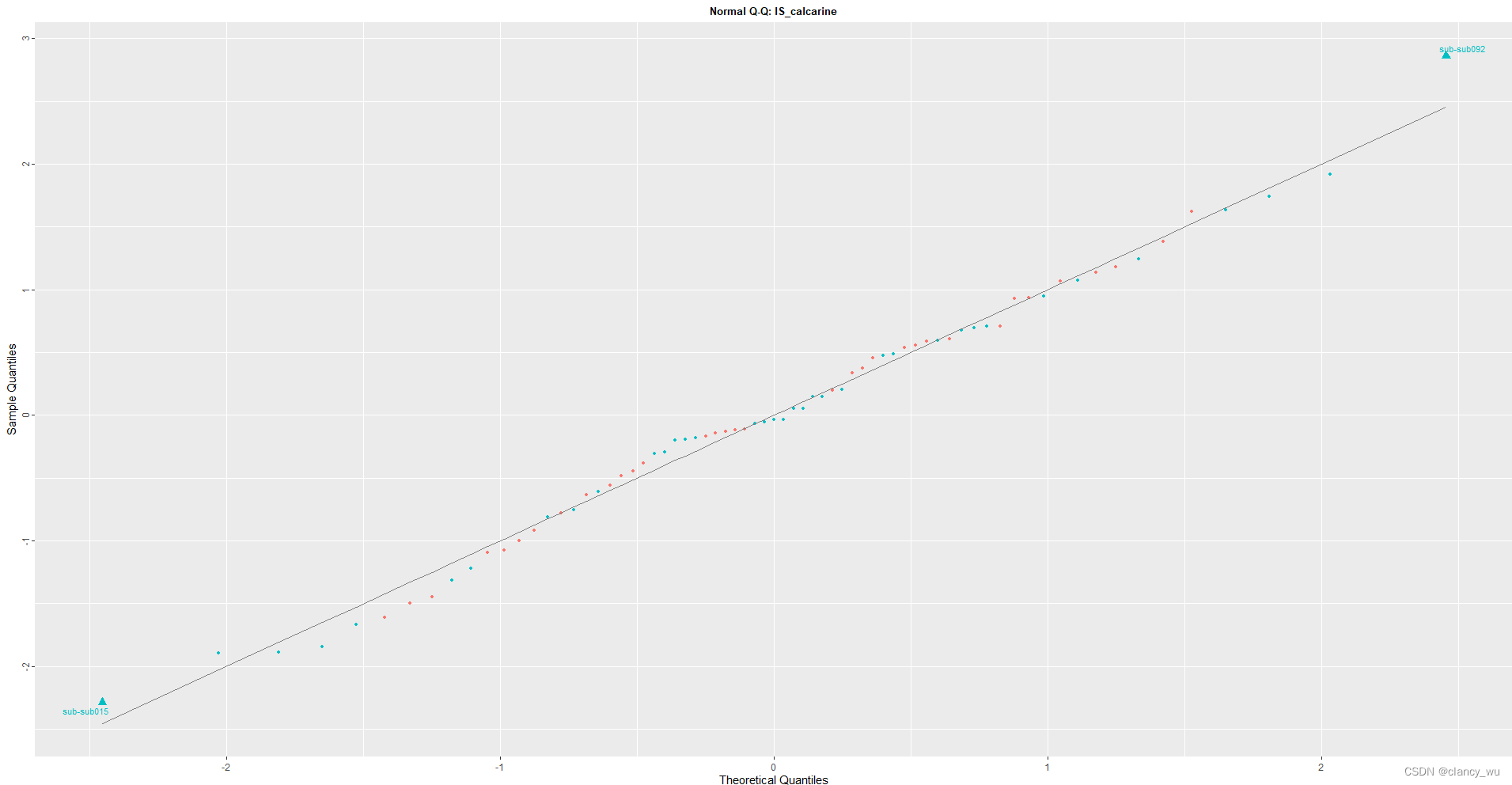
可以看出基本是呈现正态分布的。有两个偏离点(蓝色)都是患者组的。
myResids是list格式,有rawdata和resids.all:
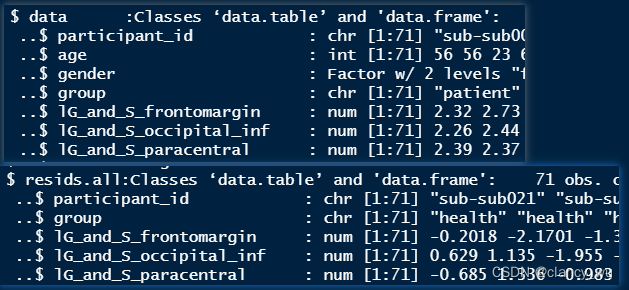
这里的rawdata就是原始数据了,然后经过使用自定义的fastLmBG(X, Y)函数,这个函数使用的是一般线性回归模型,把rawdata放进fastLmBG中,通过把年龄、性别变量回归掉,就得到resids.all矩阵了。
说到这里要提一句,一般研究是用简单皮尔逊相关或者偏相关来做的,但是这里brainGraph作者建议的是用拟合线性模型后的残差做,这个残差有专门的名字,叫studentized residuals,有时候也被称为 leave-one-out residuals。当然,如果要用偏相关做,也是没有问题的。(可能偏方差比较严格,皮尔逊相关又过于宽松。)
随后就是用corr.matrix函数生成矩阵网络。这个函数是使用Hmisc::rcorr(X$res.all)的命令来做的,格式如下:
# format
corr.matrix(resids, densities, thresholds = NULL, what = c("resids",
"raw"), exclude.reg = NULL, type = c("pearson", "spearman"),
rand = FALSE)
# default
corr.matrix(resids, densities, thresholds = NULL, what = "resids", exclude.reg = NULL, type = "pearson",
rand = FALSE)
默认是用残差做皮尔逊相关,因为在上一个阶段已经把年龄和性别回归掉了,所以再用皮尔逊,整体会比直接偏相关更柔和一些。
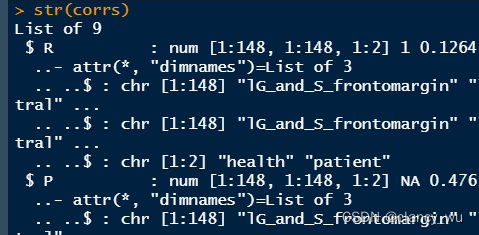
corrs经过相关计算,71X148就变成了148X148了,但是有个方便的在于,它会自动根据group分为患者组的矩阵网络和健康人的矩阵网络,这就挺好的。生成的有R值矩阵,P值矩阵,R值阈值矩阵。值得注意的是,这里的阈值,是高于阈值的部分变为1,低于阈值的部分变为0,所以整体属于无权重的无向图,非权重图,这里切记。
后面的就很简单了,把组图按照阈值切成34份,然后就可以进行图论分析了:
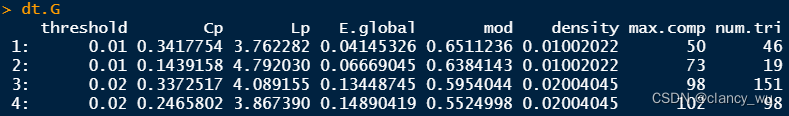
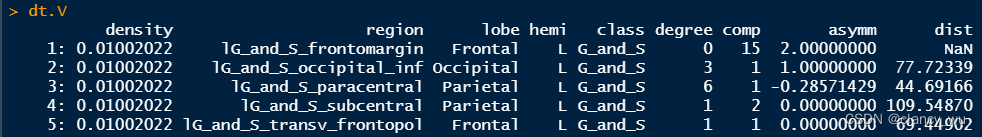
dim(dt.G)=[68,22],这里的68就是342,阈值X组别,dim(dt.V)=[10064, 30],这里的10064就是342*148,阈值X组别X节点数。
需要特别注意的是,由于每个阈值下只有1个值,无法做参数检验,只能使用置换检验来比较两组差异。这是一个特别注意的点。
当然,现在还有格兰杰因果结构协变量网络,2017年提出的,那就稍微更复杂了一点点。
具体看南京大学的一篇文献,作者开发了matlab工具包BCCT(2021年,DOI:10.3389/fnhum.2021.641961),专门用于一些结构协方差网络的分析。但是,缺点在于,这个包的manual还未发布,所以去理解这个包会比较困难。。。。。






















 5672
5672











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










