背景:
SmallRNA(小分子RNA)存在于真核生物细胞核和细胞质中,SmallRNA在细胞的生长、发育、代谢等基础生物学过程中扮演着重要的角色,甚至在癌症等相关疾病形成过程中起着关键的作用。
SmallRNA是一类高度保守的长度在18-30 nt的RNA分子,主要有两种类型的小分子RNA:一类是snRNA(small nuclearRNA),存在于细胞核中;另一类是scRNA(small cytoplasmicRNA),存在于细胞质中。小分子RNA通常与蛋白质组成复合物, 在细胞的生命活动中起重要的作用,某些snRNPs和剪接作用密切相关,它们分别与供体和受体剪接位点以及分支顺序相互补。scRNA参与蛋白质的合成和运输,如SRP颗粒就是一种由一个7SRNA和蛋白质组成的核糖核蛋白体颗粒,主要功能是识别信号肽,并将核糖体引导到内质网。Small RNA主要包括miRNA、siRNA、piRNA。
小RNA(SmallRNA,sRNA)主要是一大类功能各异的调控分子,主要包括:miRNA,ncRNA,siRNA,snoRNA,piRNA,rasiRNA(重复相关siRNA,repear-associatedsiRNA)等,它们分别在转录水平、转录后水平及表观遗传水平等方面控制基因的表达,通过多种多样的作用途径,包括mRNA降解、翻译抑制、异染色质形成以及DNA去除,广泛参与调控生物体的生长发育和疾病发生。
Smallinterfering RNA (siRNA):是一种小RNA分子(约21-25核苷酸),由Dicer(RNAaseⅢ家族中对双链RNA具有特异性的酶)加工而成。SiRNA是siRISC的主要成员,激发与之互补的目标mRNA的沉默。
MicroRNA(miRNA): 是一类由内源基因编码的长度约为22 个核苷酸的非编码单链RNA分子,它们在动植物中参与转录后基因表达调控。到目前为止,在动植物以及病毒中已经发现有4361 个miRNA 分子(Release 910 , October 2006) 。大多数miRNA基因以单拷贝、多拷贝或基因簇(cluster) 的形式存在于基因组中(Lagos2Quintanaet al , 2001 ; Lauet al , 2001) 。
piRNA:与Piwi蛋白相作用的RNA称为piRNAs。piRNAs在基因组中显示出与众不同的定位类型,主要成群地分为长20–90kb的基因簇,其中的长片断的小分子RNA只能来源于单链。相似的piRNAs在人类和小鼠中均有发现,大部分基因簇出现在同一染色体的位置上。虽然piRNA的功能仍然需要研究阐明,但是生殖细胞中的piRNA富集现象和Miwi突变导致的男性不育表明piRNA在配子形成的过程中起作用。
高通量测序技术可以对样本中所有SmallRNA家族进行测序和表达定量,从而解析miRNA、siRNA、piRNA、其它非编码RNA以及相应的靶基因序列。借助于第二代测序技术,为小分子RNA功能研究提供有力工具,可以在没有生物体基因组参考序列信息的前提下,一次性获得数百万条小分子RNA序列,从而快速全面检测小分子RNA,用于新的小分子RNA发现,或小分子RNA表达谱研究。基于Illumina高通量测序技术的小RNA数字化分析,采用边合成边测序(sequencingby synthesis,SBS),具有所需样品量少,高通量,高精确性,拥有简单易操作的自动化平台和功能强大等特点。
 派森诺生物的IlluminaMiSeq/HiSeq两大测序平台能提供优质的Small RNA测序服务。
派森诺生物的IlluminaMiSeq/HiSeq两大测序平台能提供优质的Small RNA测序服务。
优选平台:Illumina MiSeq平台具有测序周期短(1×35bp模式的测序时间仅8小时)、分辨率高、精确度高、重复性好等优点,7个工作日即可完成,可以为您节省时间。IlluminaHiSeq平台具有费用低廉、通量高等优点,为Small RNA分子的发现和研究提供了极高的覆盖深度,能够检测丰度极低的稀有SmallRNA。派森诺生物将根据合作伙伴的实际需要,选择合适的测序平台为您提供个性化的高品质Small RNA测序服务。
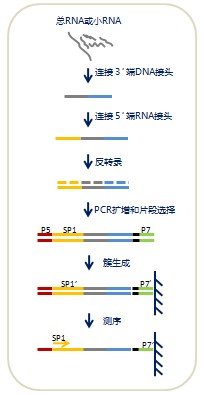
一、技术路线
|
|
|
总RNA样品提取 接3’RNA接头并纯化 |
|
连接5’RNA接头并纯化 |
|
RT-PCR合成双链cDNA序列 |
|
PCR扩增富集,制备测序文库 PAGE纯化文库 验证文库 |
|
IlluminaMiSeq/HiSeq测序 |
|
数据质控 |
|
生物信息学分析 |
|
高质量测序数据 |
二、生物信息学分析
a)
b)
c)
d)
e)
f)
g)
h)
i)
j)
k)
三、常见问题解答
1、
A: 因为SmallRNA的长度一般为18~30nt,因此我们一般选用Illumina1×35bp模式进行深度测序,所以得到的结果连接有一段3’接头序列。污染指的是5’接头分子,由于加接头时接头分子都是过量加入的,因此会有空载现象,结果造成数据中含有少量的5’接头序列污染。通常污染的比例会低于5%,属正常现象。
2、
A: 需要提供相应物种的基因组和相关的外显子、内含子以及重复信息。如果没有本物种的基因组,需要提供近缘物种的相关信息。
3、
A: 基于高通量测序技术的Small RNA测序突破了目前研究技术手段上的局限性,使研究人员能够直接对样本中的SmallRNA进行高通量测序,其主要优势有:
(1) 可以直接从核苷酸水平上研究SmallRNA分子,不存在传统芯片杂交的荧光模拟信号带来的交叉反应和背景噪音问题,非常利于区分相同家族以及序列极为相似的不同SmallRNA分子;
(2) 可以对任意物种进行高通量分析,无需任何预先的序列信息以及二级结构信息;
(3) 灵敏度高,测序通量大,为SmallRNA分子的发现和研究提供了极大的数据深度与覆盖率,能够检测丰度极低的稀有转录;
(4) 测序产生的原始数据可以与多种分析软件兼容,可以注释SmallRNA的基因组信息,并分析其表达水平,能够随时使用Small RNA数据库注释已知的SmallRNA,还可以进一步分析未匹配的数据,发现新的Small RNA种类及异构体,寻找更深入的研究信息。
4、Q: 有哪些miRNA预测软件?
A:
| 软件名 | 适用物种 | 网址 |
| PalGrade | 人 | — |
| miRscan | 线虫 | |
| Srnaloop | 线虫 | |
| miRseeker | 果蝇 | |
| findMiRNA | 拟南芥 | |
| miR-abela | 动物 | |
| BayesMiRNAfind | 动物 | |
| ProMiRⅡ | 动物 | |
| RNAz+RNAmicro | 动物 | |
| Microprocessor SVM | 动物 | |
| ERPIN | 动植物 | |
| MiRAlign | 动植物 | http://bioinfo.au.tsinghua.edu.cn/miralign/ |
| microHARVESTER | 植物 | |
| MIRFINDER | 植物 | |
| Vmir | 病毒 |
5、Q: 有哪些miRNA靶基因预测软件?
A:miRNA靶基因预测软件分为第一代预测软件和第二代预测软件,两者的区别是所采用的算法不同,前者大多是从种子互补这一规则出发设计算法,其次才考虑miRNA靶基因跨物种间的保守性;而后者更倾向于采用机器学习方法训练参数进行靶基因预测。现在,多采用第二代target预测方法对miRNA靶基因进行预测。
| 软件 | 适用范围 | 网址 |
| 第一代miRNA target预测软件: | ||
| MicroInspector | 哺乳动物 | |
| DIANA-microT | 哺乳动物 | http://www.diana.pcbi.upenn.edu/ |
| RNAhybrid | 哺乳动物 | http://bibiserv.techfak.uni-bielefeld.de/rnahybrid/ |
| miRanda | 脊椎动物 | http://www.microrna.org/ |
| TargetScan | 脊椎动物 | http://tiger.dbs.nus.edu.sg/microtar/ |
| 第二代miRNA target预测软件: | ||
| microTar | 线虫、果蝇和小鼠 | http://tiger.dbs.nus.edu.sg/microtar/ |
| TargetBoost | 线虫和果蝇 | https://demo1.interagon.com/targetboost/ |
| miTarget | 哺乳动物 | http://cbit.snu.ac.kr/~miTarget/ |
| RNA22 | 哺乳动物 | |
| PicTar | 哺乳动物 | |























 995
995

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








