AlphaFold3|万字长文解读
AlphaFold3预测所有分子相互作用准确结构
AlphaFold3
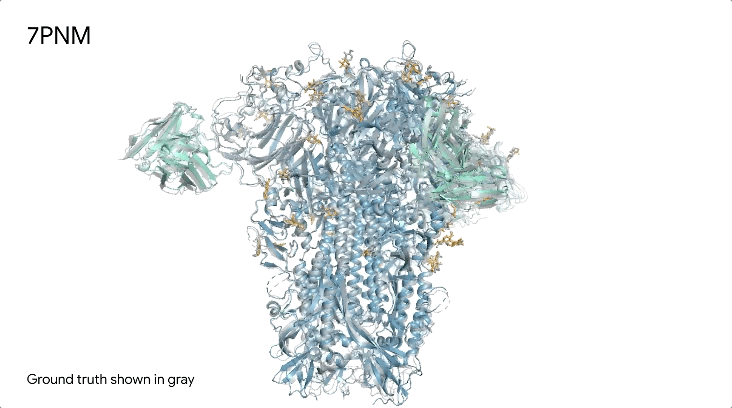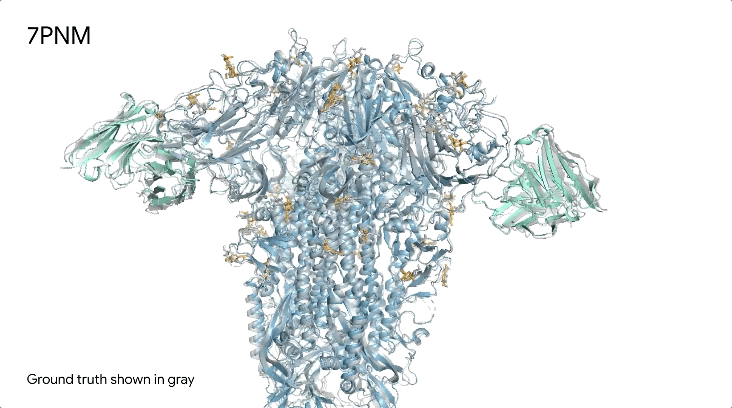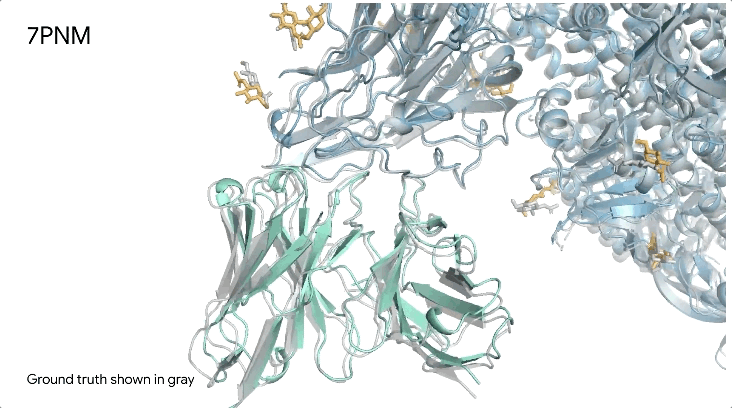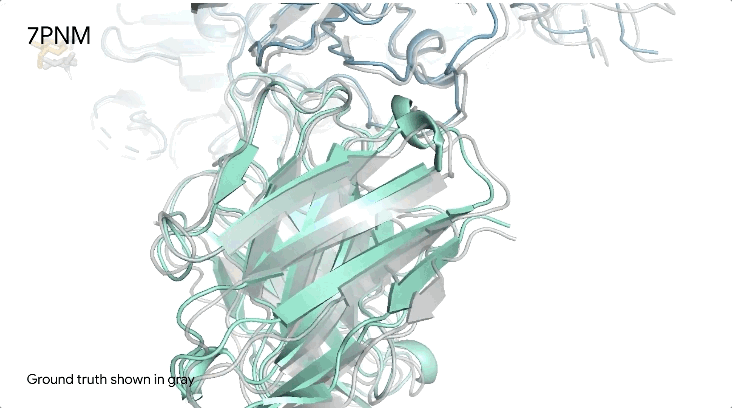
自2021年AlphaFold2问世以来,科研工作者们便开始利用这一蛋白结构预测模型来详细描绘众多蛋白质的结构、探索新药。近日,Google DeepMind公司推出了其最新产品,AlphaFold3模型。该模型不仅能够预测蛋白质结构,还能对核酸、小分子、离子以及化学修饰等和蛋白组成的复合物进行结构预测。
AlphaFold3在各种场景准确性上有巨幅提升,这些场景包括蛋白质-小分子、蛋白质-核酸、以及抗体-抗原相互作用等。这表明,通过单一深度学习框架,完全可以预测所有生物分子相互作用的高精确度结构。DeepMind团队还推出了AlphaFold Server,用户可以免费访问使用。AlphaFold3在生物学界具有革命性的意义,将使得结构生物学的研究变得更加普及。这项研究的成果以《Accurate structure prediction of biomolecular interactions with AlphaFold 3》为题,于2024年5月8日刊登在《Nature》杂志上。
目录索引
-
背景介绍
-
模型架构
-
- 输入信息的嵌入
- 序列局部原子注意力机制
- MSA模块
- 对表示的三角形更新
- 模版模块
- PairFormer模块
- 扩散模块
- 置信度模块
-
模型表现
-
- 蛋白-小分子配体
- 蛋白核酸或RNA单体
- 共价修饰(键合配体、糖基化、RNA/DNA/蛋白修饰)
- 蛋白多体/单体
-
AF3准确度与置信度指标正相关
-
PairFormer模块
-
AF3模型局限性
正文内容
背景介绍
准确预测的生物分子复合物结构,对于理解细胞功能和疾病治疗至关重要。AlphaFold2使得蛋白结构预测已经能达到实验精度,基于AF2也迸发出很多算法和进展。
这就引出科学问题:是否能用AI算法准确预测所有生物分子,包括:DNA、RNA、小分子、离子、化学修饰等,的复合物结构。虽然最近也有类似的工作RFAA,但精度还欠佳。
本文作者提出AlphaFold 3(AF3),该模型能够高精度预测各种分子类型,包括PDB数据库中的几乎所有分子类型(下图)。

图1|**a,**DNA和cGMP复合物;b,冠状病毒OC43刺突蛋白;灰色为共晶。
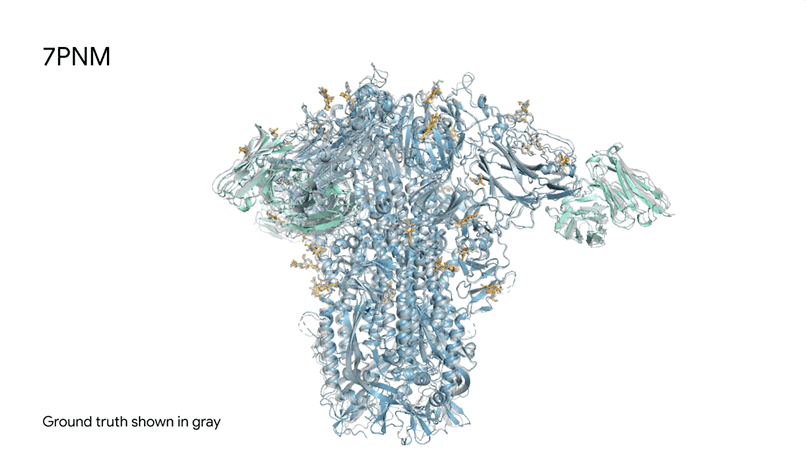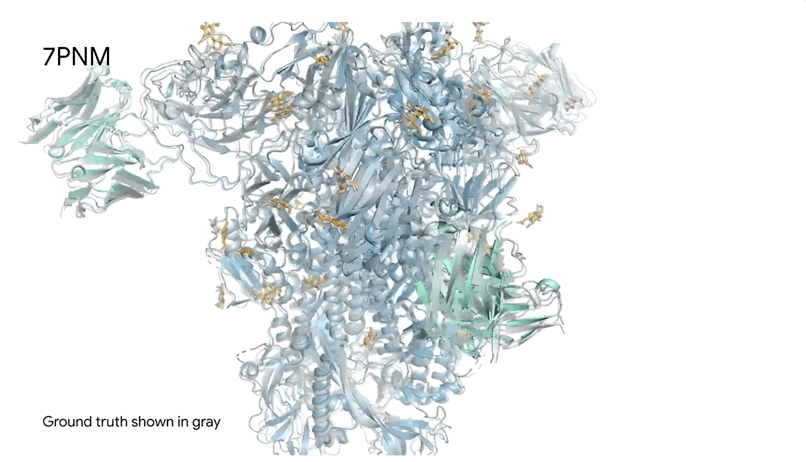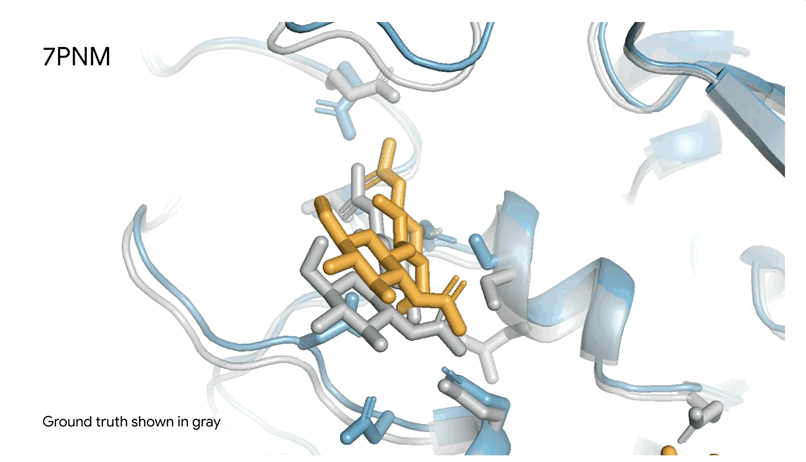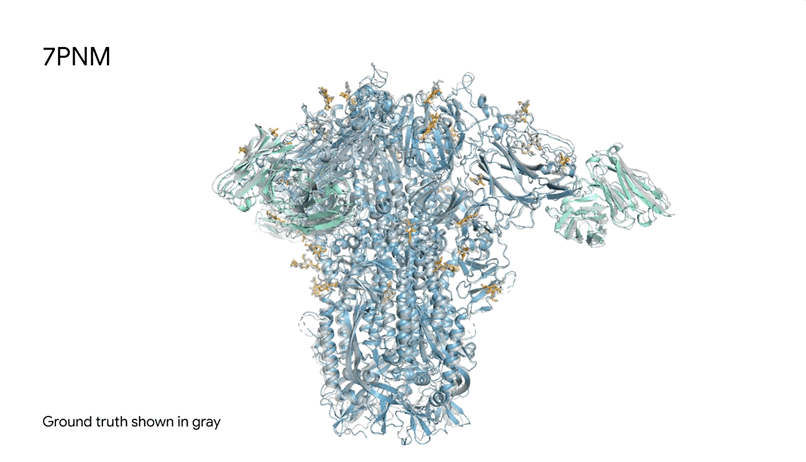
7PNM|一种普通感冒病毒的刺突蛋白(冠状病毒OC43),AlphaFold 3预测结构与共晶结构对比。其中刺突蛋白(蓝色)、抗体(青绿色)和简单糖(黄色),共晶结构为灰色。结构有助于更好地理解冠状病毒,包括COVID-19,为改进治疗提供可能。
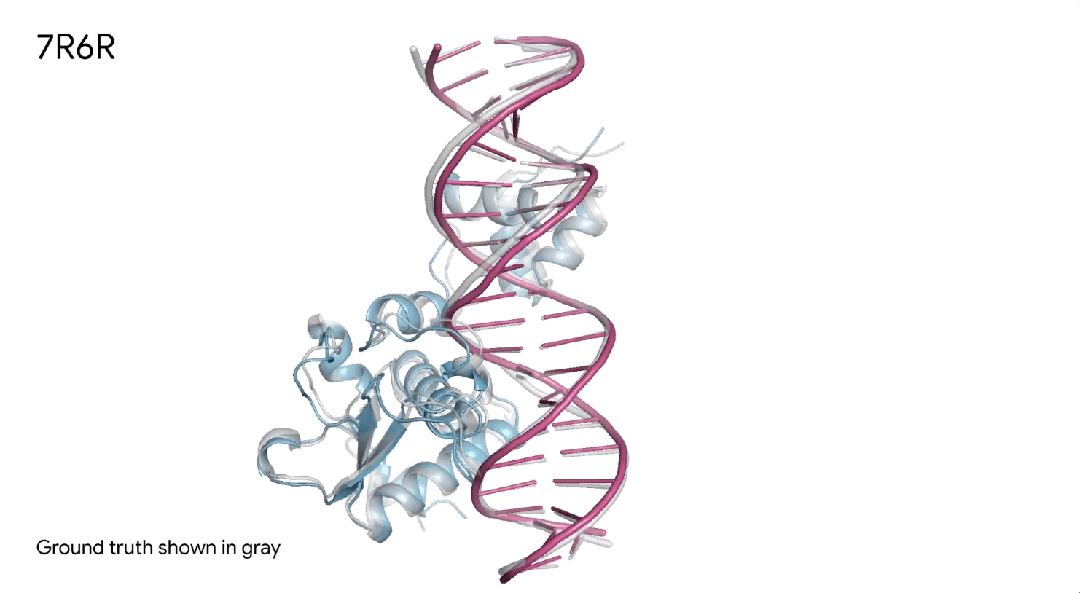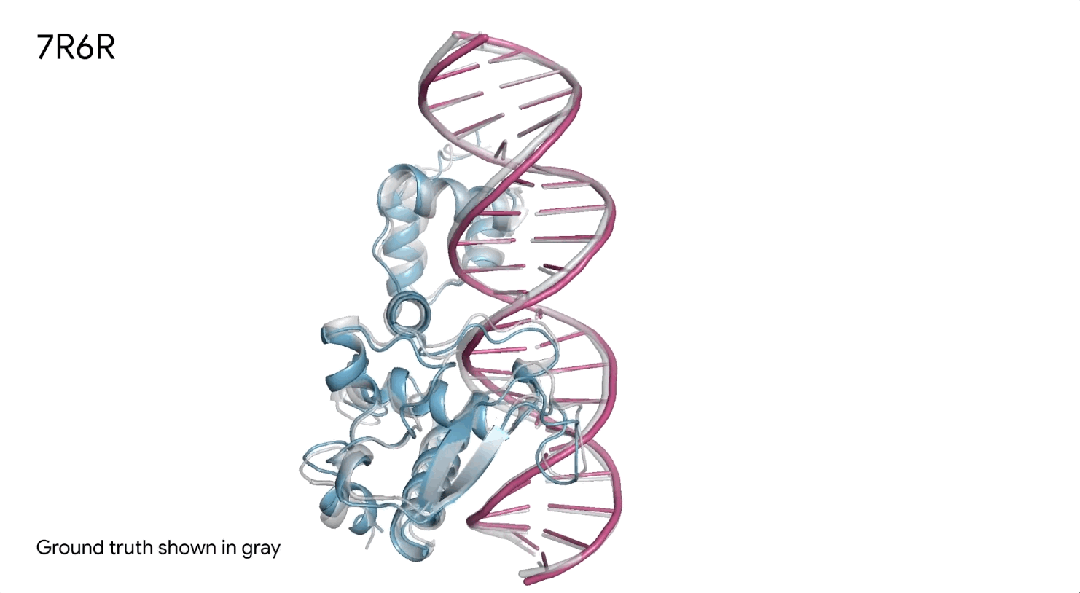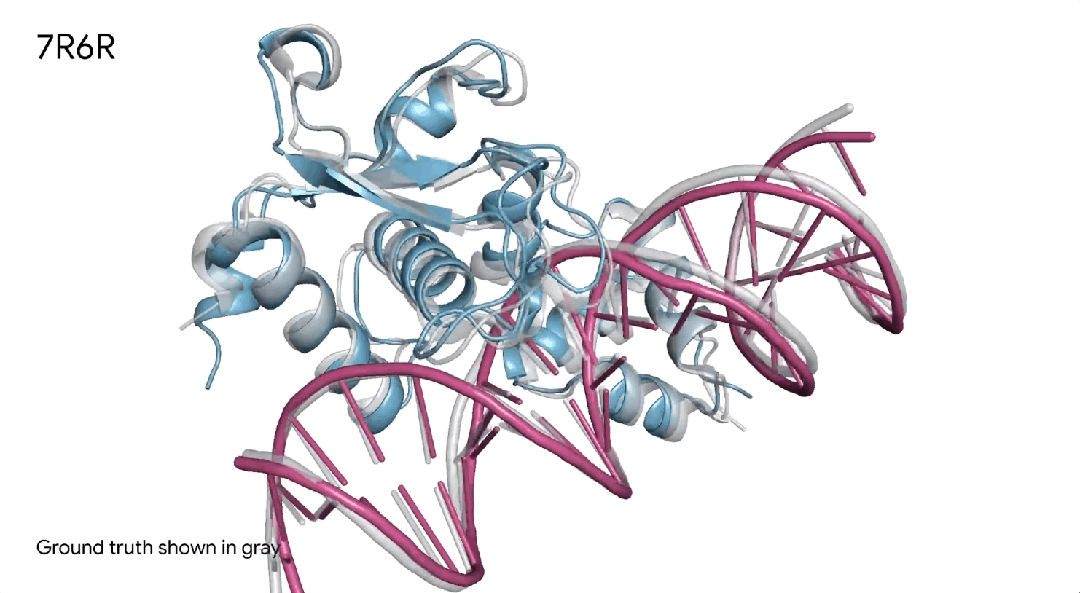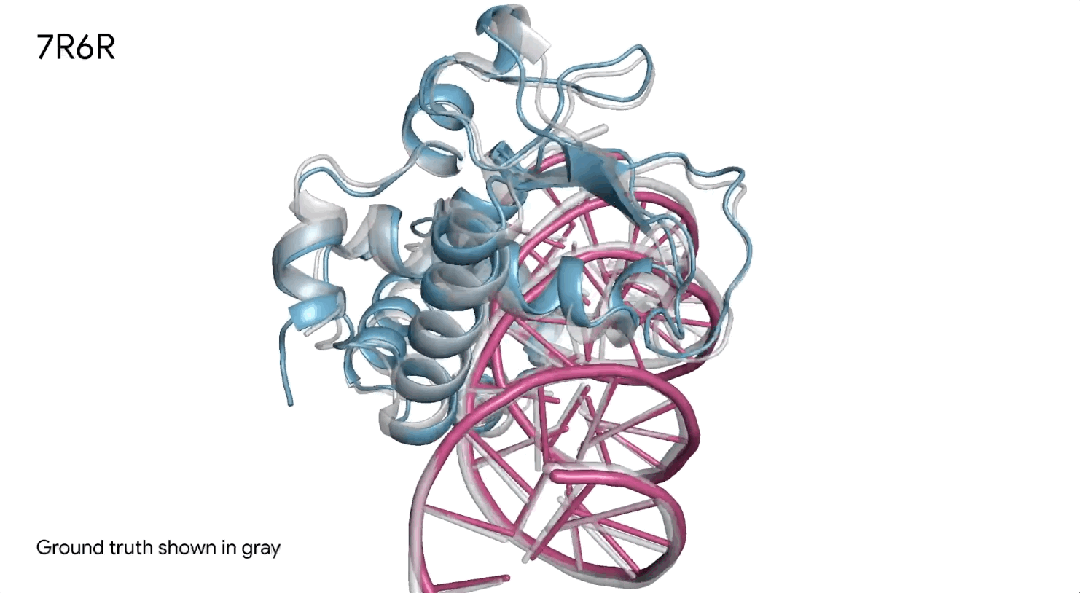
7R6R|AlphaFold 3对DNA结合蛋白分子复合物的预测,其中蓝色蛋白质与紫色DNA双螺旋结合,几乎完美地匹配灰色真实结晶结构。
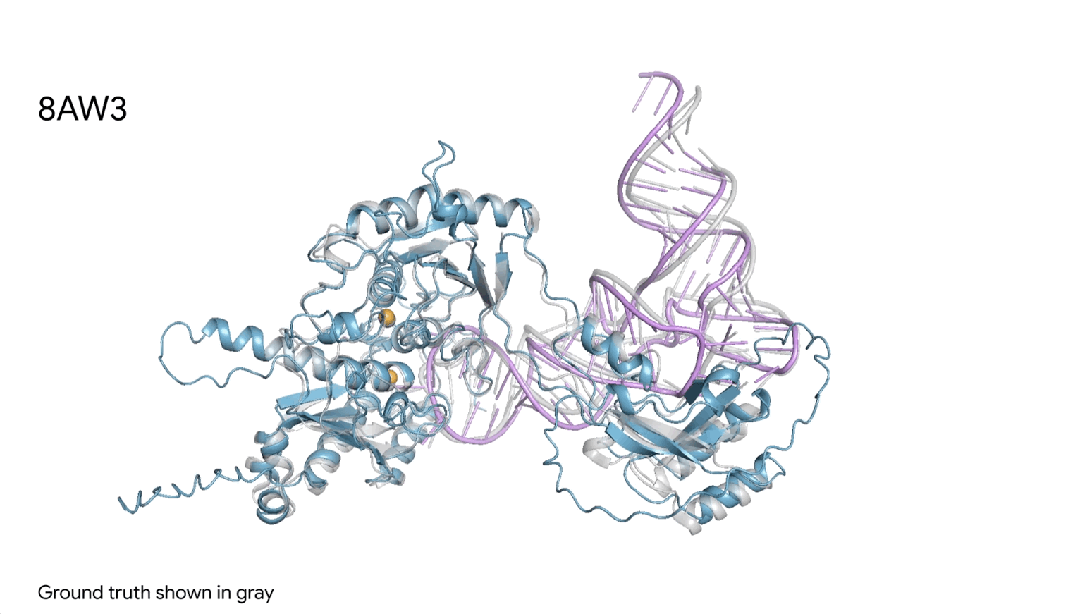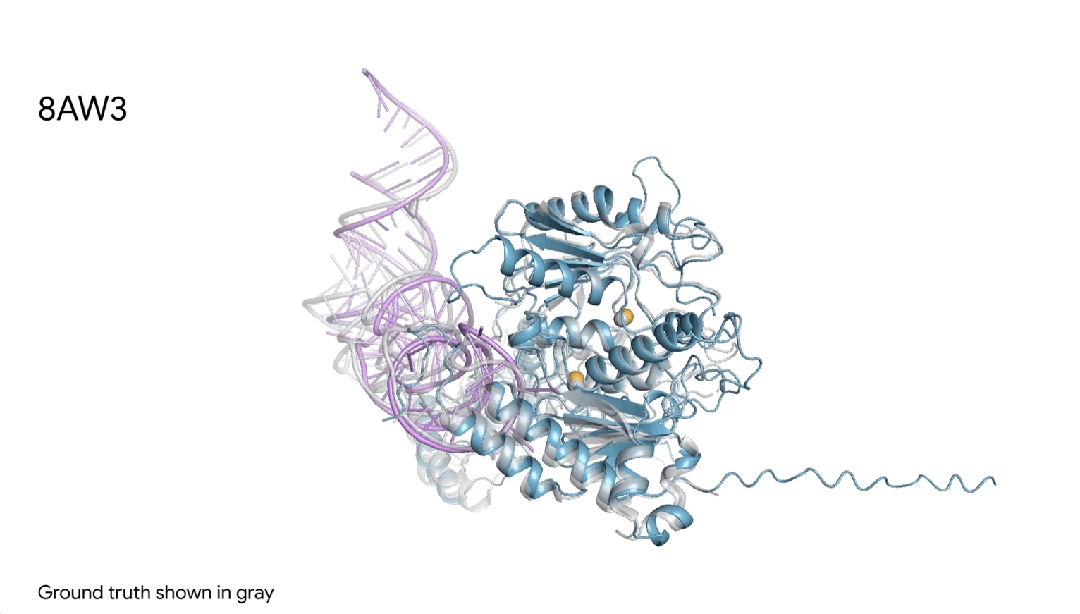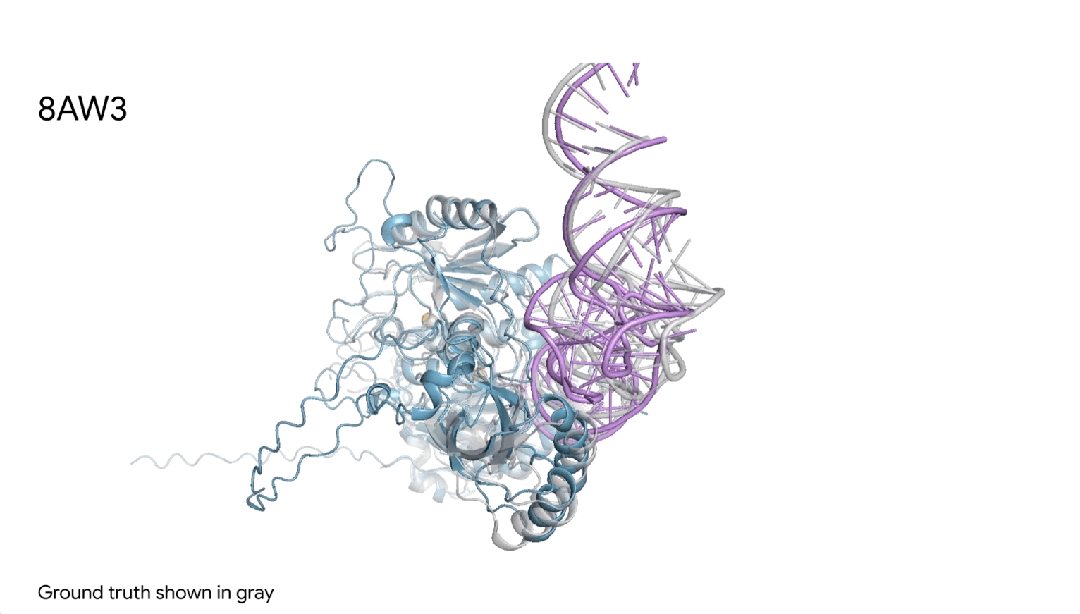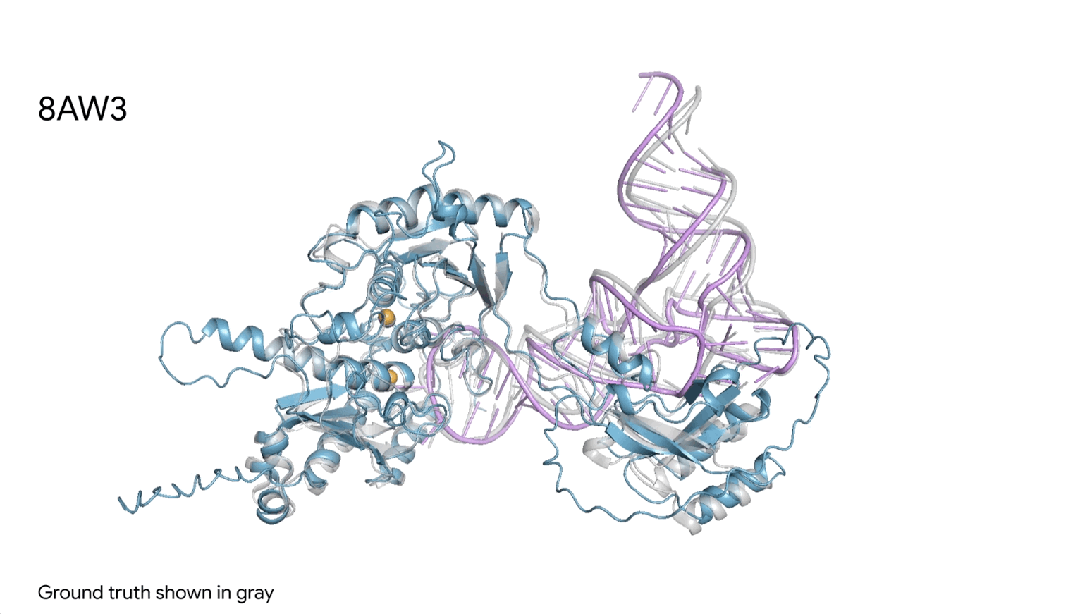
图18AW3 |AlphaFold 3对一个蛋白+RNA修饰分子复合物的预测,其中包含一个蓝色蛋白质、一根紫色RNA链、两个黄色离子,可见与真实灰色结构非常相似。
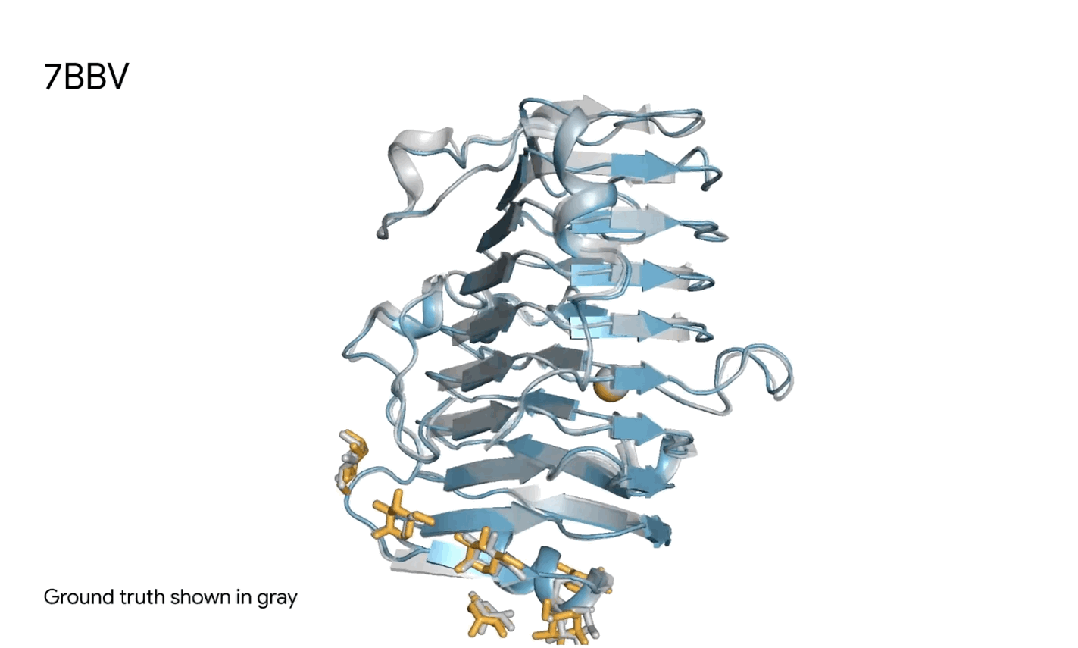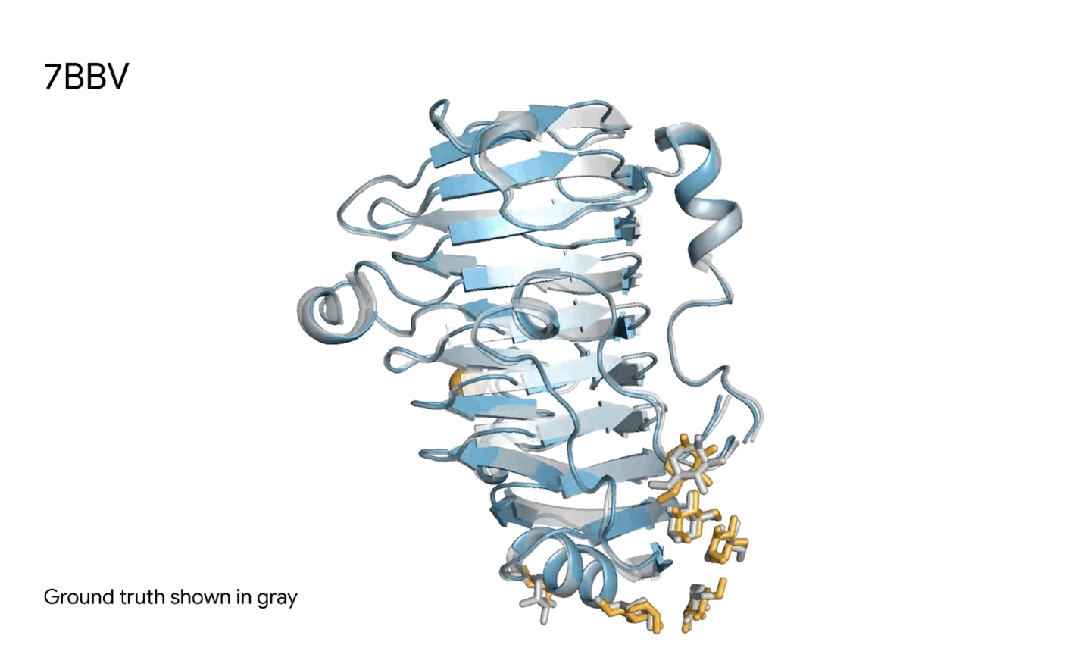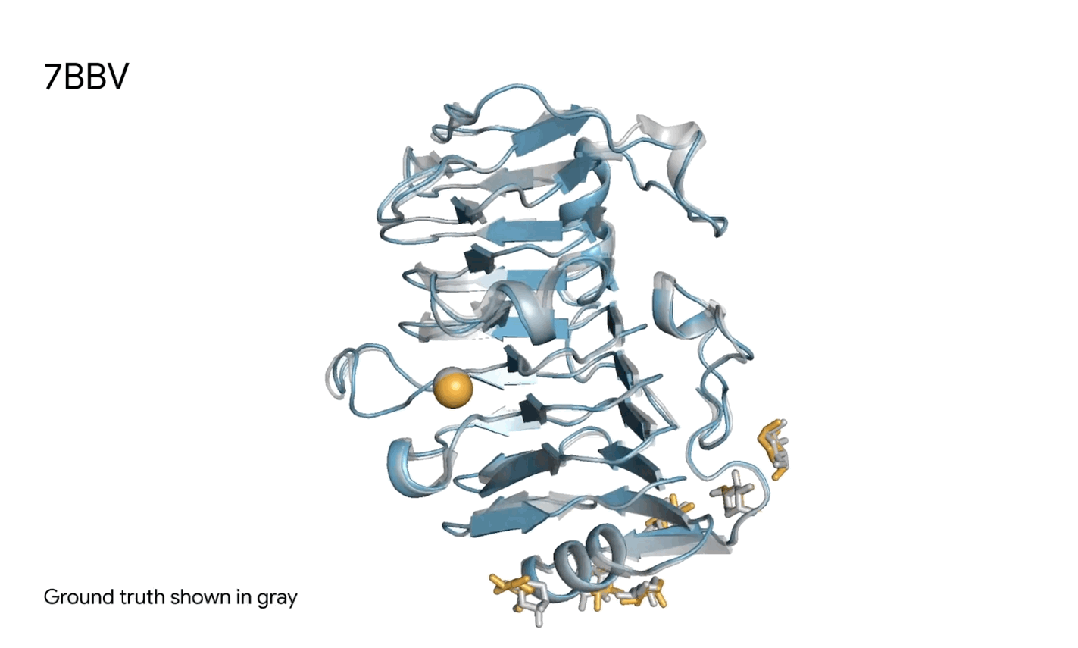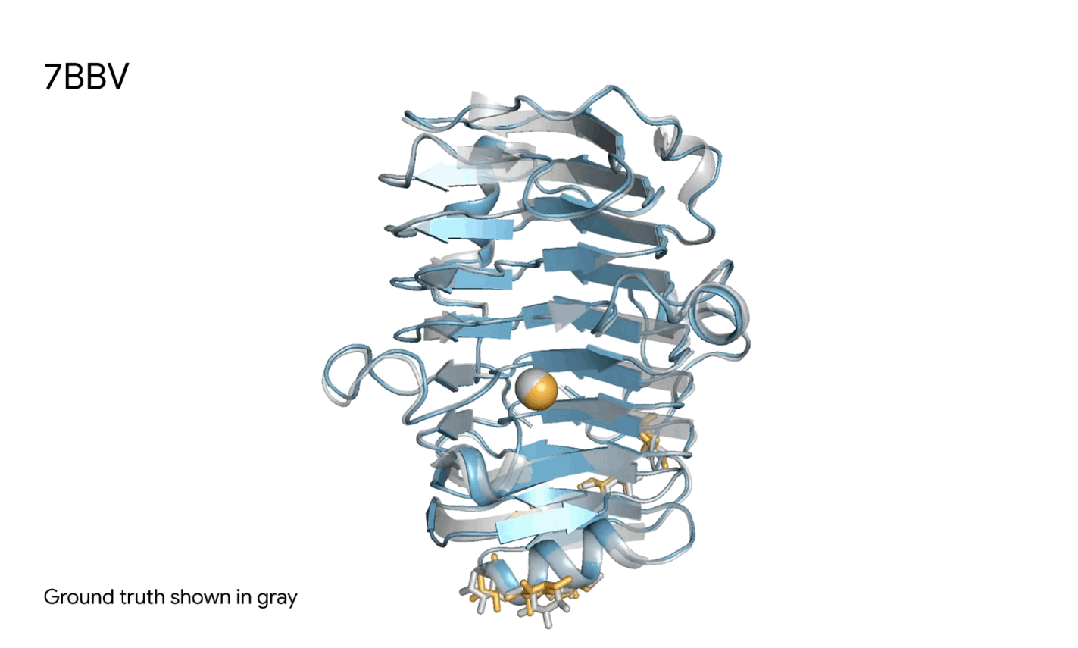
7BBV|AlphaFold 3对一个酶分子复合体的预测,其中包括一个蓝色酶蛋白,一个黄色离子、和黄色简单糖,以及真实灰色结构。这种酶存在于一种土壤传播的黄萎镰孢菌,会对各种植物造成损害。了解这种酶如何与植物细胞相互作用,有助于研究人员开发更健康、更具抗性的作物。
模型架构
AF3出色的结果是通过改进AlphaFold2架构,然后进行训练实现的。AF3既能输入更多种类的分子数据,也优化了学习效率。这些改进点有:
- AF3用更简单的Pairformer模块替换Evoformer,减少了多序列比对MSA处理的数量,图2a。
- AF3使用扩散模块直接预测原始原子坐标,取代了AF2中的IPA结构模块(图2b)。扩散的多尺度性质(低噪声引导神经网络,改善分子的局部结构),也能消除立体化学损失 ,还能特殊处理网络的键合模式(bonding patterns)。

图1d|AF3的架构。矩形表示处理模块,箭头表示数据流向。黄色:输入数据,蓝色:抽象网络激活,绿色:输出数据。彩色代表分子物理原子坐标。
前面的模版/基因搜索&构象生成细节如下:
第一种是基因搜索(Genetic search),蛋白质链的序列搜索用了2种搜索方法,搜索了5个库(与AF2类似);RNA链在3个数据库(Rfam\RNACentral\Nucleotide collection)上进行搜索。
第二种是模板搜索(Template search),该搜索提供的是模板结构信息(与AF2类似),选择搜索到的前4个模板作为输入。需要注意的是模板搜索仅针对单链的蛋白质进行,不提供多链的模板信息。在预测复合物时,模型不会尝试从相同PDB中选择模板,从而获得有关链间的相互作用信息。
第三种是构象生成(Conformer generation),该功能提供了氨基酸、核苷酸或配体的参考构象(AF3新增)。给定输入的CCD代码(一种用于标识化学组分的简短字符串编码系统)或SMILES字符(一种用来描述分子结构的字符串表示方法),使用RDKit(一种开源的分子信息学工具包)的ETKDGv3方法(一种分子构象生成算法)生成构象。
–摘自《力文所》
AF3 的整体结构(图1d)与AF2的整体结构相呼应,其中一个变化是复合








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1032
1032

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








