
#这里需要对非模式物种制作ORG.DB包,如果是模式物种,“https://bioconductor.org/packages/release/BiocViews.html#___OrgDb”该网站有自带的成熟的包,自行下载使用就行。
#对上一个教程中得到的out.emapper.annotations文件,对表头修整下:

#windows上的R运行
library(dplyr)
library(stringr)
library(clusterProfiler)
library(AnnotationForge)
library(tidyr)
options(stringsAsFactors = F)#keep character but not factor conversion
emapper <- read.delim("out.emapper.annotations")
emapper[emapper=="-"] <- NA#change "-" to "NA"
emapper <- emapper[-(49584:49586),]#remove the final 3 rows
gene_info <- dplyr::select(emapper, GID=query, Gene_Name=seed_ortholog)%>%
dplyr::filter(!is.na(Gene_Name))#gene_info表格

#提取GO信息
gene2go <- dplyr::select(emapper,GID=query, GO=GOs)%>%
filter(!is.na(GO))%>%
mutate(EVIDENCE='IEA')%>%
separate_rows(GO, sep = ',', convert = F)#gene2go表格,其实和实战8中,TBTOOLS做出来的是一样的。

#构建orgdb包
AnnotationForge::makeOrgPackage(gene_info=gene_info,
go=gene2go,
maintainer = 'LJH',
author = 'LJH',
outputDir = "./",
tax_id = 0000,
genus = 'P',
species = 'tri',
goTable = "go",
version = "1.0")#对新生成的org.Ptri.eg.db包中的DESCRIPTION,进行修改,Maintainer: LJH <abc@cba.com>,
#打包
pkgbuild::build('./org.Ptri.eg.db', dest_path = './')
#生成org.Ptri.eg.db_1.0.tar.gz将这个R包放到平时R包安装的路径中,本地安装
install.packages('your_path', repos = NULL)
library(org.Ptri.eg.db)
#将实战5中的差异基因自行excel修改下基因名,使其与gene_info中的GID相对应
#差异分析
DE <- read.delim("DE_genes_filter.txt")
ego <- enrichGO(gene = DE$GID,
OrgDb = org.Ptri.eg.db,
keyType = 'GID',
ont = 'ALL',
pvalueCutoff = 0.05,
qvalueCutoff = 0.05)
#以下是自带的clusterprofiler的画图函数
dotplot(ego)
barplot(ego)
cnetplot(ego)
#这个富集文件要自己用EXCEL修改,我自己选了15条BP-4条CC-15条MF。GeneRatio自己做成百分比。#以下是用GGPLOT2画条形图了,各种函数,自己调节参数即可。write.table(ego, file = "Ptri_GO_test",sep = '\t',quote = F)ego2 <- read.delim("Ptri_GO_test")
library(ggplot2)
library(GOplot)
ggplot(ego2, aes(Description, -log10(p.adjust))) +
geom_col(aes(fill = ONTOLOGY), width = 0.5, show.legend = FALSE) +
scale_fill_manual(values = c('#D06660', '#5AAD36', '#6C85F5')) +
facet_grid(ONTOLOGY~., scale = 'free_y', space = 'free_y') +
theme(panel.grid = element_blank(), panel.background = element_rect(color = 'black', fill = 'transparent')) +
scale_y_continuous(expand = expansion(mult = c(0, 0.1))) +
coord_flip() +
labs(x = '', y = '-log10(p.adjust)')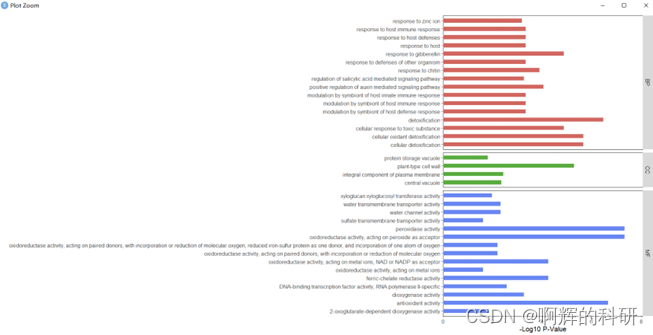
#GGPLOT2画气泡图
pp <- ggplot(ego2, aes(GeneRatio, Description))
pp + geom_point() +
geom_point(aes(size = Count)) +
geom_point(aes(size = Count, color = -1 * log10(qvalue))) +
scale_colour_gradient(low = "green", high = "red") +
labs(color = expression(-log[10](Qvalue)), size = "Gene Number", x = "Rich Factor", y = "Pathway Name", title = "Top 30 of Pathway Enrichment") +
theme_bw()
##########图片自己微调吧#######
#小林家的龙女仆






















 7189
7189











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










