字数 558,阅读大约需 3 分钟
ReverseDock:基于分子对接的反向找靶
先前我们介绍过一款基于药效团的反向找靶工具:
本期将介绍另一款基于分子对接的反向找靶工具——ReverseDock
网址:https://reversedock.biologie.uni-freiburg.de/
1. 上传ligand

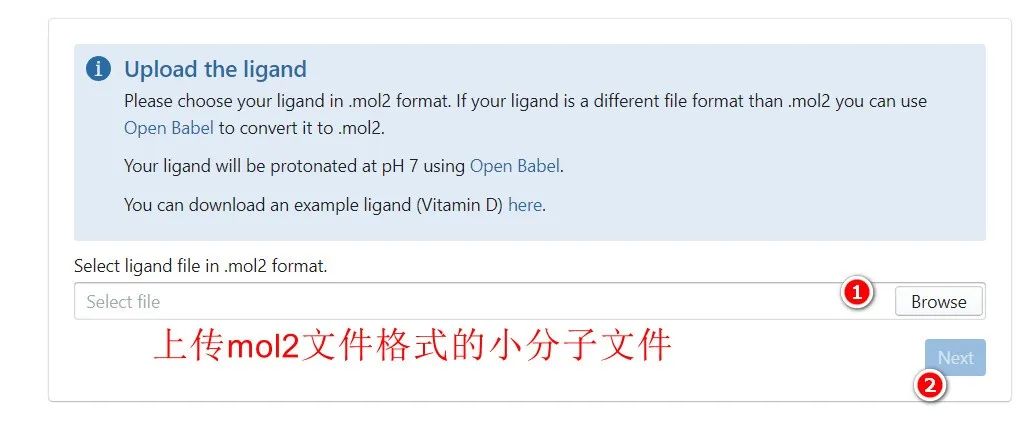
进入 Submit 页面,选择拟用于对接的配体文件(需为.mol2 格式)。完成选择后,点击 “Next”,进入目标蛋白选择环节。
2. 上传筛选的靶点数据
在此步骤,存在两种选择路径。其一,可上传最多 100 个目标蛋白,要求每个蛋白文件小于 1000 个氨基酸;其二,提交最多 100 个 UniProt ids,用于 AlphaFold 预测,同样限制每条序列不超过 1000 个氨基酸。

上传pdb文件(最多100个)
上传最多 100 个以.pdb 格式存储的目标蛋白文件,每个文件对应的蛋白序列需小于 1000 个氨基酸。
这一步它会自动对蛋白文件进行预处理:
①清除 DNA/RNA;
②应用 PDBFixer 处理:添加缺失残基;替换非标准残基;移除杂原子(包括水);添加缺失重原子
③使用 pdb2pqr 进行质子化处理

或者选择alphafold模式并输入uniprot id
提交最多 100 个 UniProt ids 进行 AlphaFold 预测,只有氨基酸数量少于 1000 的序列可用于对接。
目前,reverse dock 工具仅支持以下几个物种中的alphafold预测的蛋白:
-
人类(Homo sapiens)
-
大肠杆菌(Escherichia coli strain K12)
-
小鼠(Mus musculus)
-
果蝇(Drosophila melanogaster)
-
线虫(Caenorhabditis elegans)
-
拟南芥(Arabidopsis thaliana)
-
酿酒酵母(Saccharomyces cerevisiae)
-
挪威大鼠(Rattus norvegicus)

3. 提交任务
提交过程中,可选择填写邮箱地址(非必须),以便任务完成时接收通知,及时获取结果信息。

4. 查看任务状态
提交成功后,系统会自动跳转至任务状态页面。在此,可实时掌握任务进度,一旦结果生成,便能第一时间查看。任务运行期间,也可随时回顾进度、浏览中间结果。

为便于后续查看,建议将该页面添加至书签收藏。

5. 查看与下载结果
对接结果以表格形式呈现,包含文件名及计算所得的结合能数据。若需更直观地观察结果,点击小眼睛图标,即可进行 3D 可视化展示。

在分子查看器中,点击 “Screenshot” 按钮,可将可视化状态保存为图片。

此外,点击对应图标,能够下载每次运行的对接结果,其中涵盖排名前五的对接构象(查看器中仅展示最优构象)及目标蛋白文件。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










