基因家族成员筛选与分析
基因家族
基因家族是来源于同一个祖先,由一个基因通过基因重复而产生两个或更多的拷贝而构成的一组基因,它们在结构和功能上具有明显的相似性,编码相似的蛋白质产物。
研究基因家族的意义
- 基因家族的基因在物种之间都是比较保守的,通过基因家族分析可以得到某物种特有的家族基因,而这些基因则有可能与该物种的特异性有关。(筛选)
- 通过对多物种构建系统发育树,从而得到物种起源进化或亲缘关系方面的信息,并为后续遗传操作提供参考。(进化树)
- 通过分析家族基因在进化过程受到的正向选择,确定与该物种环境适应性相关的基因。(Ka/Ks)
主要工具:TBtools (https://www.sciencedirect.com/science/article/pii/S1674205220301878)

TBtools下载及安装
https://tbtools.cowtransfer.com/s/0a9cbf41b47b4a

1. 数据库下载
1.1 EnsemblPlants (http://plants.ensembl.org/info/data/ftp/index.html)


1.2 Phytozome 13 (https://phytozome-next.jgi.doe.gov/)


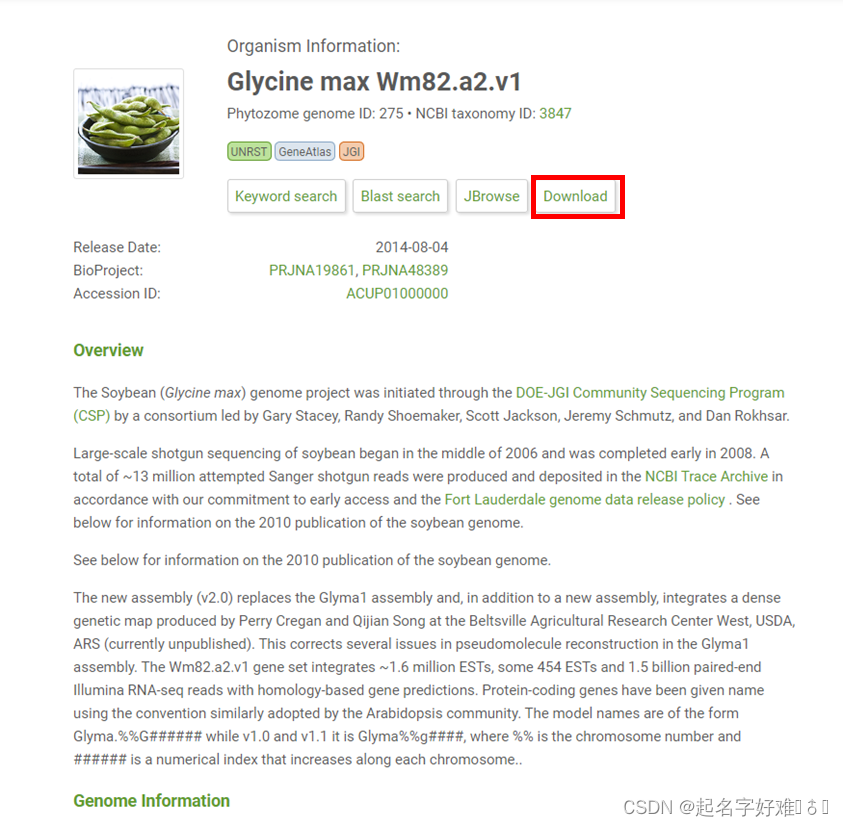




2. 去除可变剪切
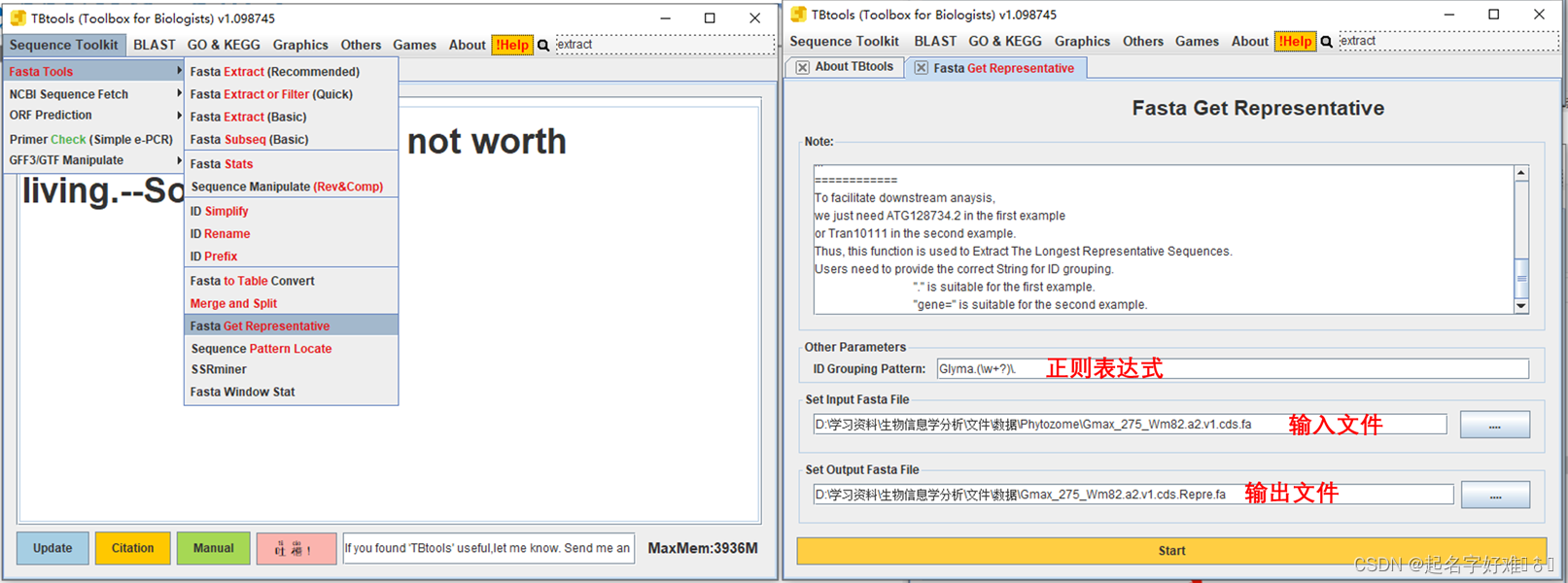
3. 隐马尔可夫模型 (HMM: Hidden Markov Model)
HMM(隐马尔可夫模型) 是一种统计模型,从可观察的参数中确定该过程的隐含参数,然后利用这些参数来作进一步的分析。
https://www.ebi.ac.uk/interpro/entry/pfam/#table

4. 基因家族成员筛选
4.1 利用HMM进行筛选

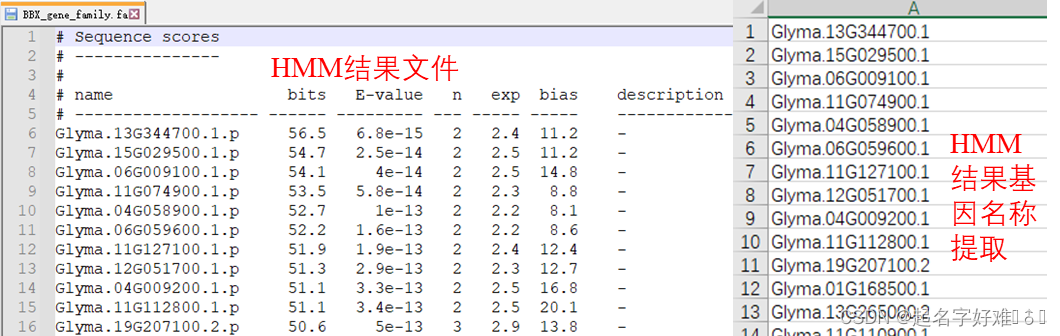
fasta序列提取


4.2 本地blast筛选
4.2.1 下载:https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/

4.2.2 安装

4.2.3 用户环境变量设置
此电脑-属性-高级系统设置-环境变量
 在用户变量下方:新建-变量名:balstdb,变量值为电脑安装好新建的db文件夹的路径
在用户变量下方:新建-变量名:balstdb,变量值为电脑安装好新建的db文件夹的路径

在系统变量下方:Path-添加变量值为电脑上bin文件夹位置

4.2.4 本地blast筛选
将整理好的蛋白组数据(fasta)放入到db文件夹中,在Windows PowerShell中运行以下代码:
#第一步,格式化数据库
makeblastdb.exe -in Gmax_275_Wm82.a2.v1.protein.Repre.fa -parse_seqids -hash_index -dbtype prot
在db文件夹下创建target.txt的文本文件,将用来blast的fasta序列放入

在db 文件夹下创建out.txt的文本文件,用于记录blast结果
blastp.exe -task blastp -query target.txt -db Gmax_275_Wm82.a2.v1.protein.Repre.fa -out out.txt -evalue 1e-10 -outfmt 6 -num_threads 2

合并HMM结果和blast结果,提取fasta序列,用于后续分析。
5. 结构与预测
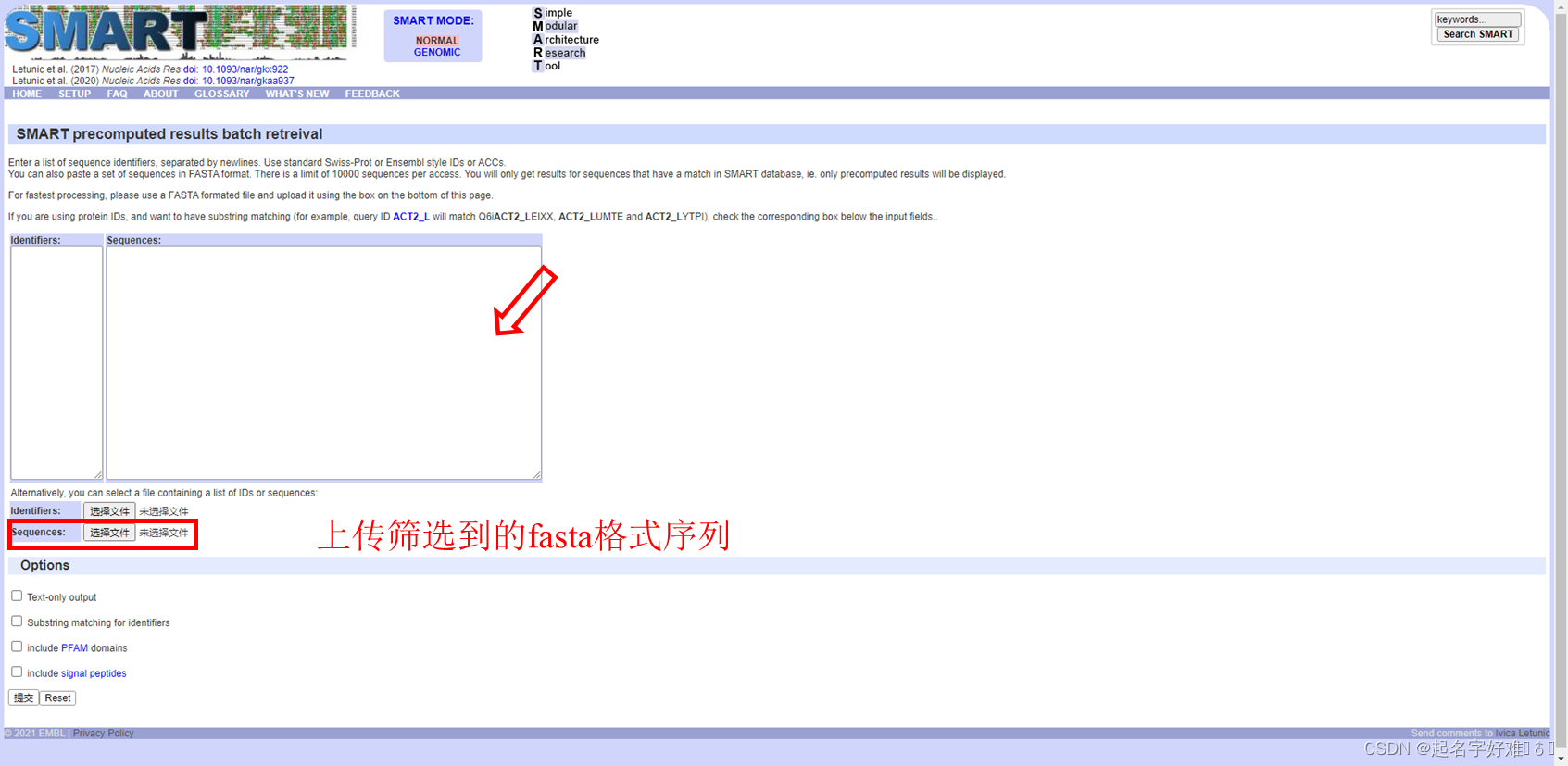
5.1 SMART
http://smart.embl-heidelberg.de/smart/set_mode.cgi?NORMAL=1


5.2 CDD
https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi




6. 结构域可视化

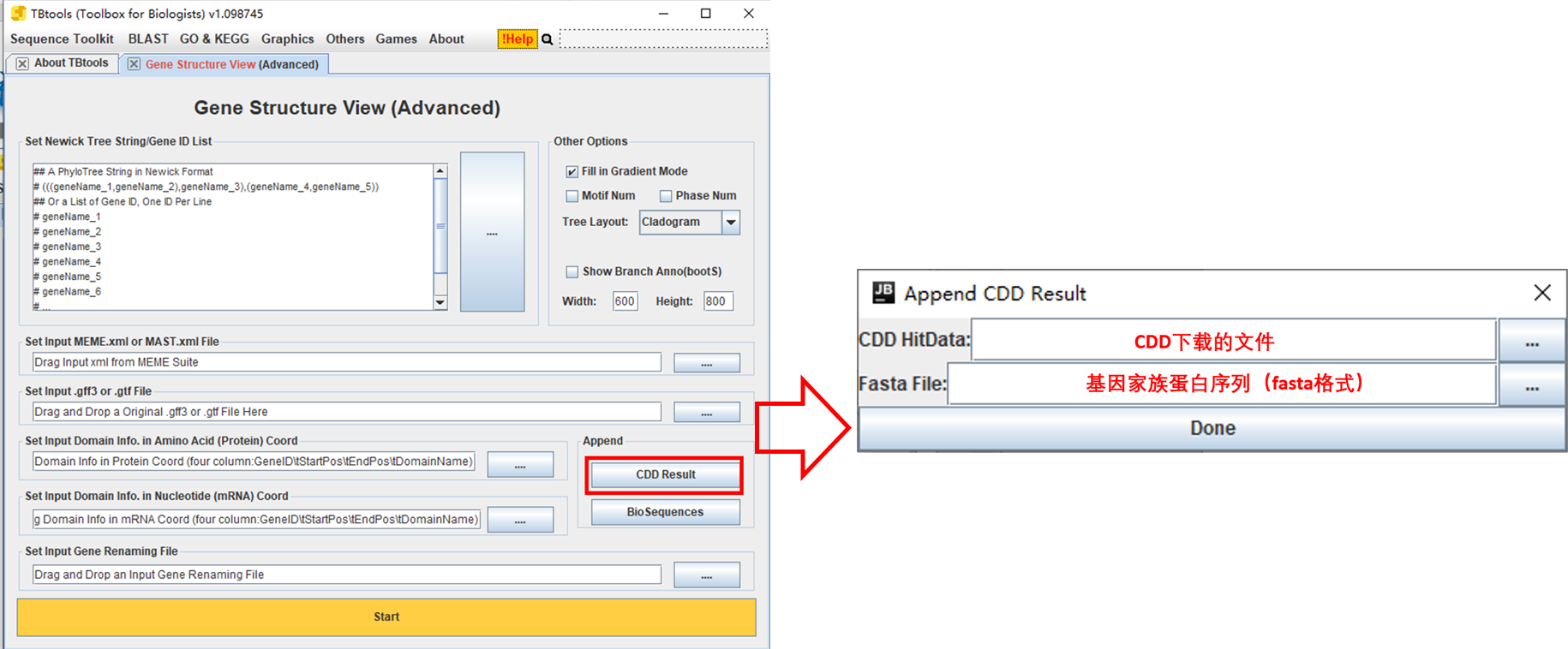

6. 系统发育进化树

6.1 工具:MEGA
6.1.1 构建进化树








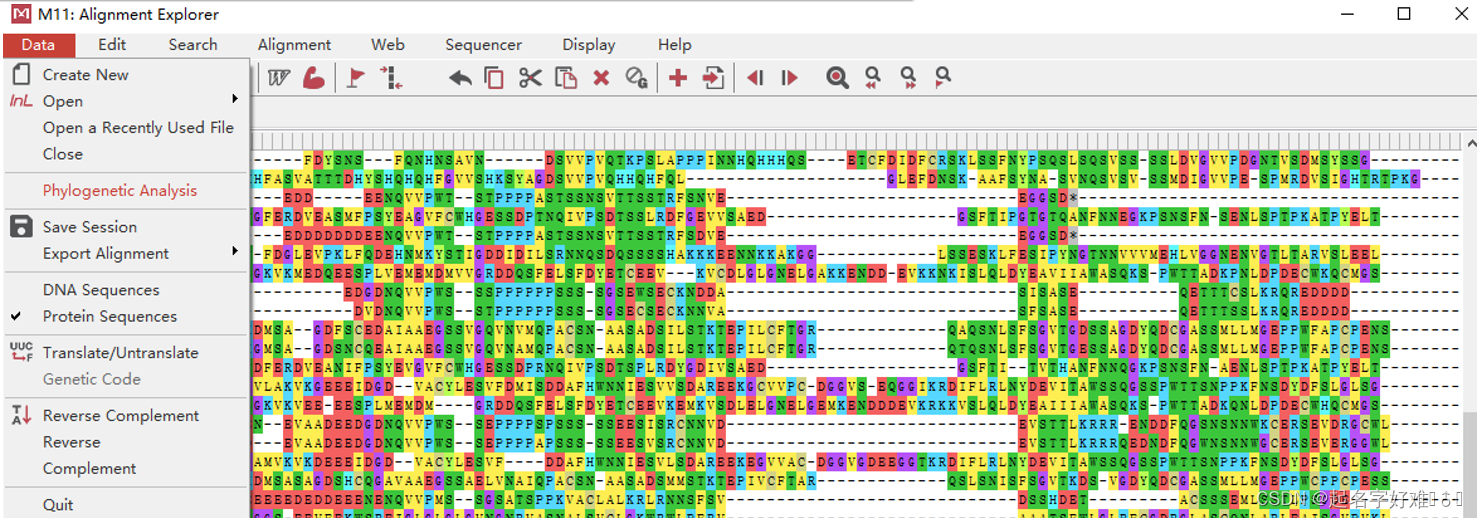




6.1.2 进化树可视化

























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








