首先
 参考教程
参考教程
https://docs.scvi-tools.org/en/0.6.3/tutorials/basic_tutorial.html
其中输入
首先需要安装pytorch
pip install torch==1.10.2
pip install scvi[notebooks]
即可,然后下载scVI自带数据需要注意
例如
import sys
show_plot = True
test_mode = False
save_path = "data/"
if not test_mode:
save_path = "data/"
import os
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from scvi.dataset import CortexDataset, RetinaDataset
from scvi.models import VAE
from scvi.inference import UnsupervisedTrainer, load_posterior
from scvi import set_seed
import torch
# Control UMAP numba warnings
import warnings; warnings.simplefilter('ignore')
%matplotlib inline
# Sets torch and numpy random seeds, run after all scvi imports
set_seed(0)
retina_dataset = RetinaDataset(save_path=save_path)
这里数据总是下载失败,可以去源码里找到下载loom文件的原始链接,然后浏览器下载保存在data文件里,就可以离线家在,但是我发现加载这个数据会出现一个问题,就是
 解决办法
解决办法
pip install h5py==2.10.0
使用教程参考
https://docs.scvi-tools.org/en/0.6.3/tutorials/basic_tutorial.html
我今天刚发现,scVI是有两个,其实可能不止两个,我上面的安装是做单batch 分析的
svVI-tools(integration)安装
反正我到是没搞懂,为社么scVI不弄成一个环境,
安装教程
https://docs.scvi-tools.org/en/stable/installation.html
即
conda create -n scvi-env python=3.9
conda activate scvi-env
pip install scvi-tools[tutorials]
pip install ipykernel
python -m ipykernel install --name scvi_env --user
** 这个环境叫scvi_env,而之前安装的环境叫scVI**
这个两个不同的环境,就离谱为啥要弄成两个环境,感觉作者是有病
使用教程参考
https://docs.scvi-tools.org/en/stable/tutorials/notebooks/api_overview.html
我发现一个问题,就是最新的scvi-tools里面的scanpy是1.9.1,但是1.9.1写入的h5ad在scanpy1.7.2中读取不了,很奇怪,我暂时不管这个问题了,所以我在python3.6.10中装了scvi-tools的0.7.1,已经是很旧的版本了,下面记录一下怎么使用旧版本的教程
首先google scvi-tools
第一个点入进去,是
 点击Docs->tutorials
点击Docs->tutorials

这里显示的是最新版的教程,有函数已经被弃掉了,就是说scvi-tools最新版的教程用scvi-tools 0.7.1是会报错的,

所以切换之前的版本的教程

但是这样点其实是没用的
真正的做法是点击 Project Home,然后出现的界面是

然后点击自己对应的版本,就可以进入之前版本的scvi-tools的教程了,最终的界面如下

然后点击User guide,然后点击
 这个就就可以进入scvi-tools==0.8.1 的RNA-seq的data integration的教程了
这个就就可以进入scvi-tools==0.8.1 的RNA-seq的data integration的教程了
scvi-tools(0.8.1 tutorial)
import scvi
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
import pandas as pd
import scanpy as sc
print(scvi.__version__)
print(sc.__version__)
import sys
print(sys.version)
import umap
print(umap.__version__)
sc.set_figure_params(figsize=(4, 4))
结果如下

adata=sc.read("4batch_4celltype_multi.h5ad")
print(adata)

adata.layers["counts"] = adata.X.copy()
sc.pp.normalize_total(adata, target_sum=1e4)
sc.pp.log1p(adata)
adata.raw = adata # keep full dimension safe
sc.pp.highly_variable_genes(
adata,
flavor="seurat_v3",
n_top_genes=1000,
layer="counts",
batch_key="BATCH",
subset=True
)
# sc.tl.pca(adata)
# sc.pp.neighbors(adata)
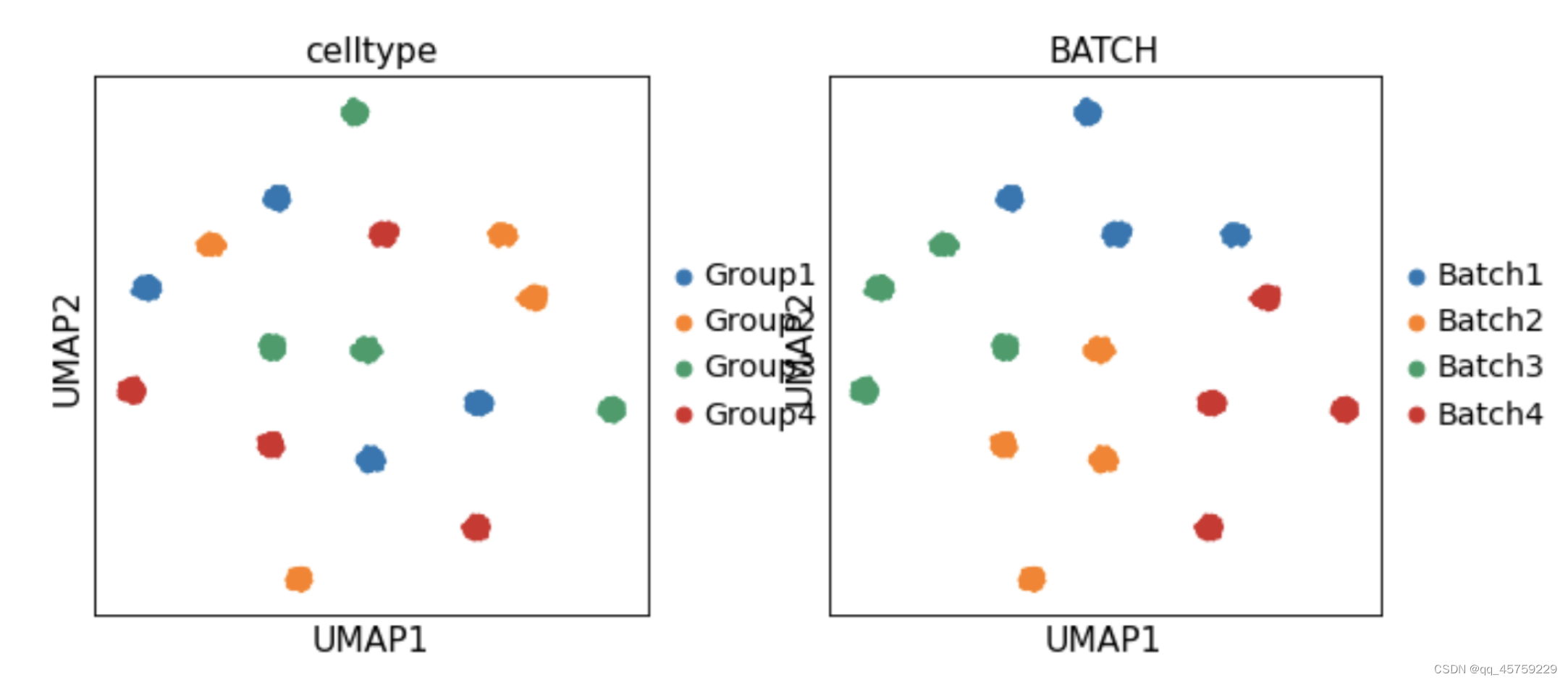
# sc.tl.umap(adata)
# sc.pl.umap(adata,color=["celltype","BATCH"])
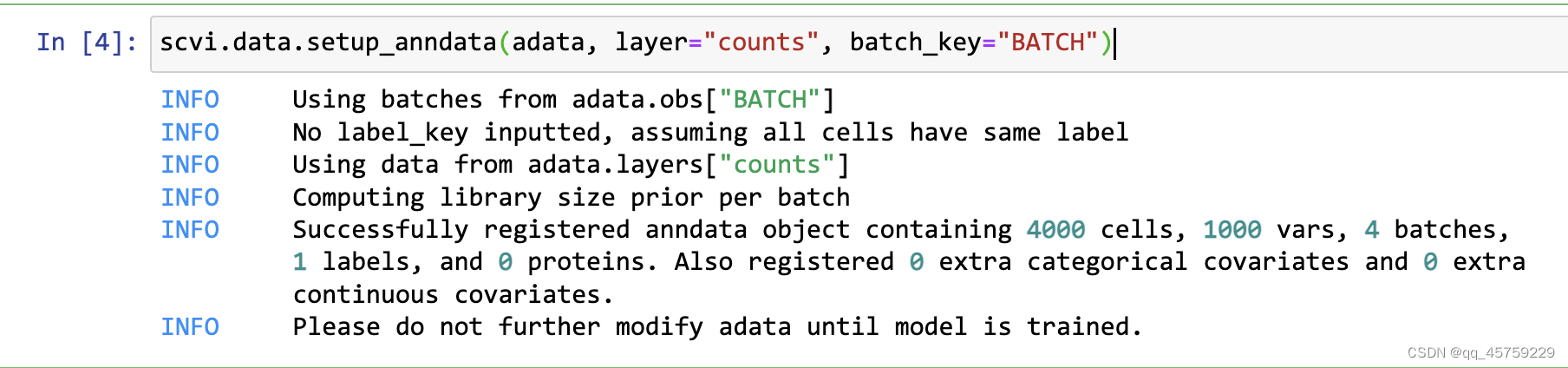
scvi.data.setup_anndata(adata, layer="counts", batch_key="BATCH")
vae = scvi.model.SCVI(adata)
vae.train()
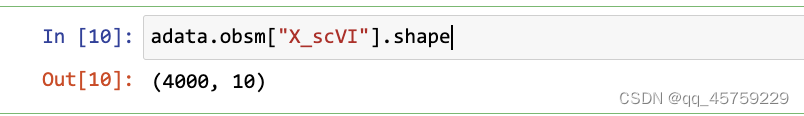
adata.obsm["X_scVI"] = vae.get_latent_representation()
sc.pp.neighbors(adata, use_rep="X_scVI")
sc.tl.louvain(adata)
sc.tl.umap(adata)
sc.pl.umap(
adata,
color=["BATCH", "louvain"],
frameon=False,
ncols=1,
)
结果如下


注意这里使用不同版本的scVI默认都是使用GPU加速的,如果想强制设置成cpu运行,那么scVI 0.8.1的方式是
 而scVI 0.17.1使用的方式是
而scVI 0.17.1使用的方式是
 两者不一样
两者不一样

会自动安装pytorch以及cuda
然后安装
pip install scib-metrics
然后运行教程
https://docs.scvi-tools.org/en/stable/tutorials/notebooks/harmonization.html
使用教程2
https://ccbskillssem.github.io/assets/scvi_notebook.html
这里程序感觉是有一个bug
比如


























 870
870

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








