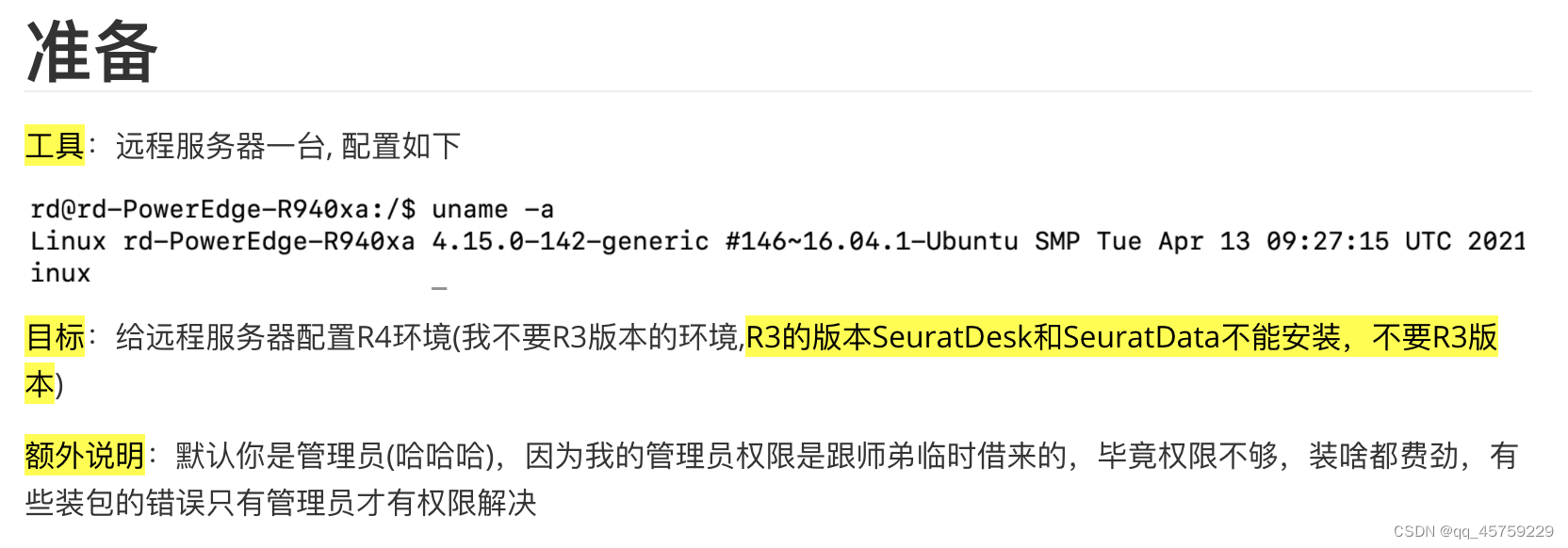
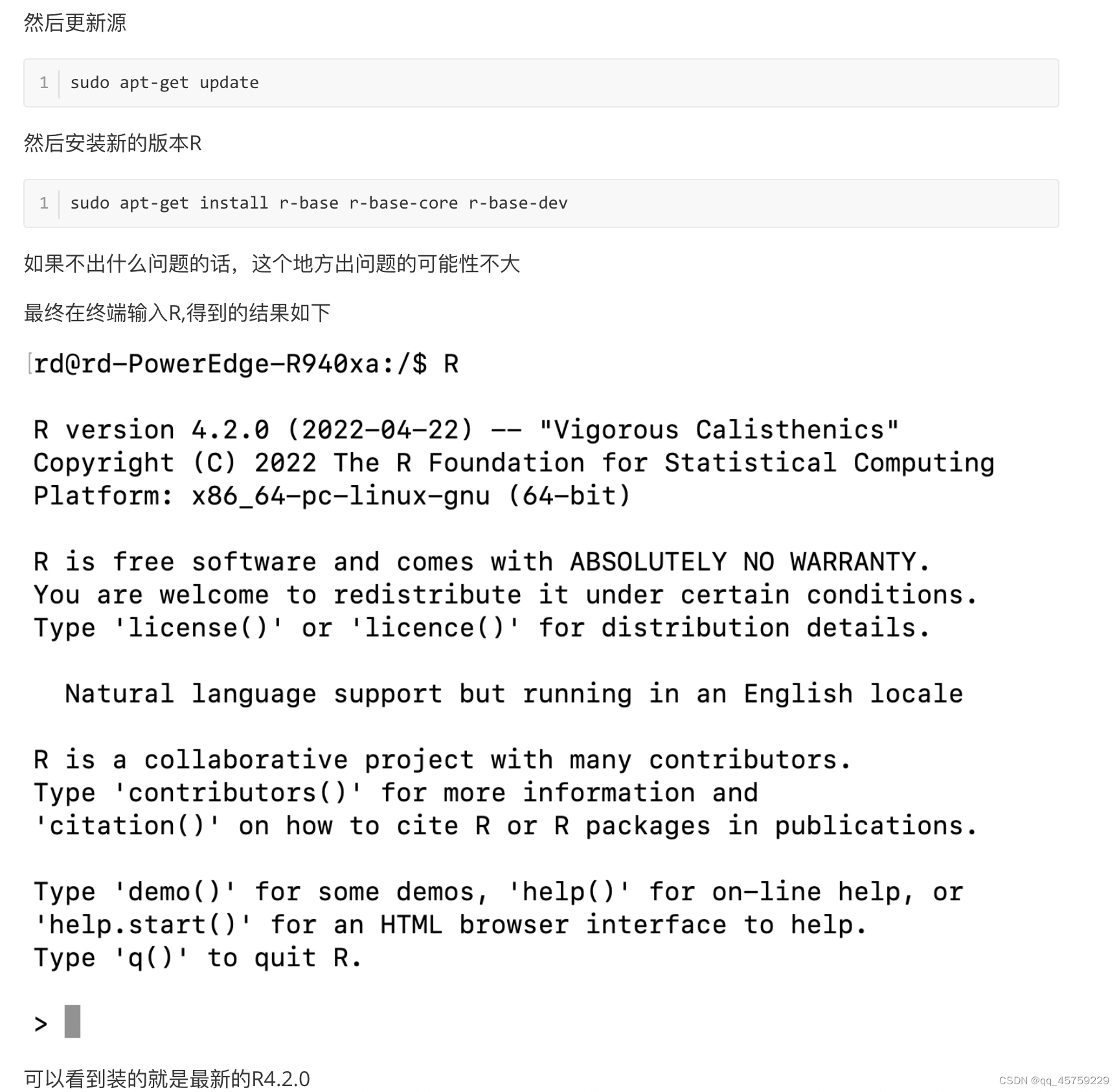
[https://mirrors.tuna.tsinghua.edu.cn/CRAN/]

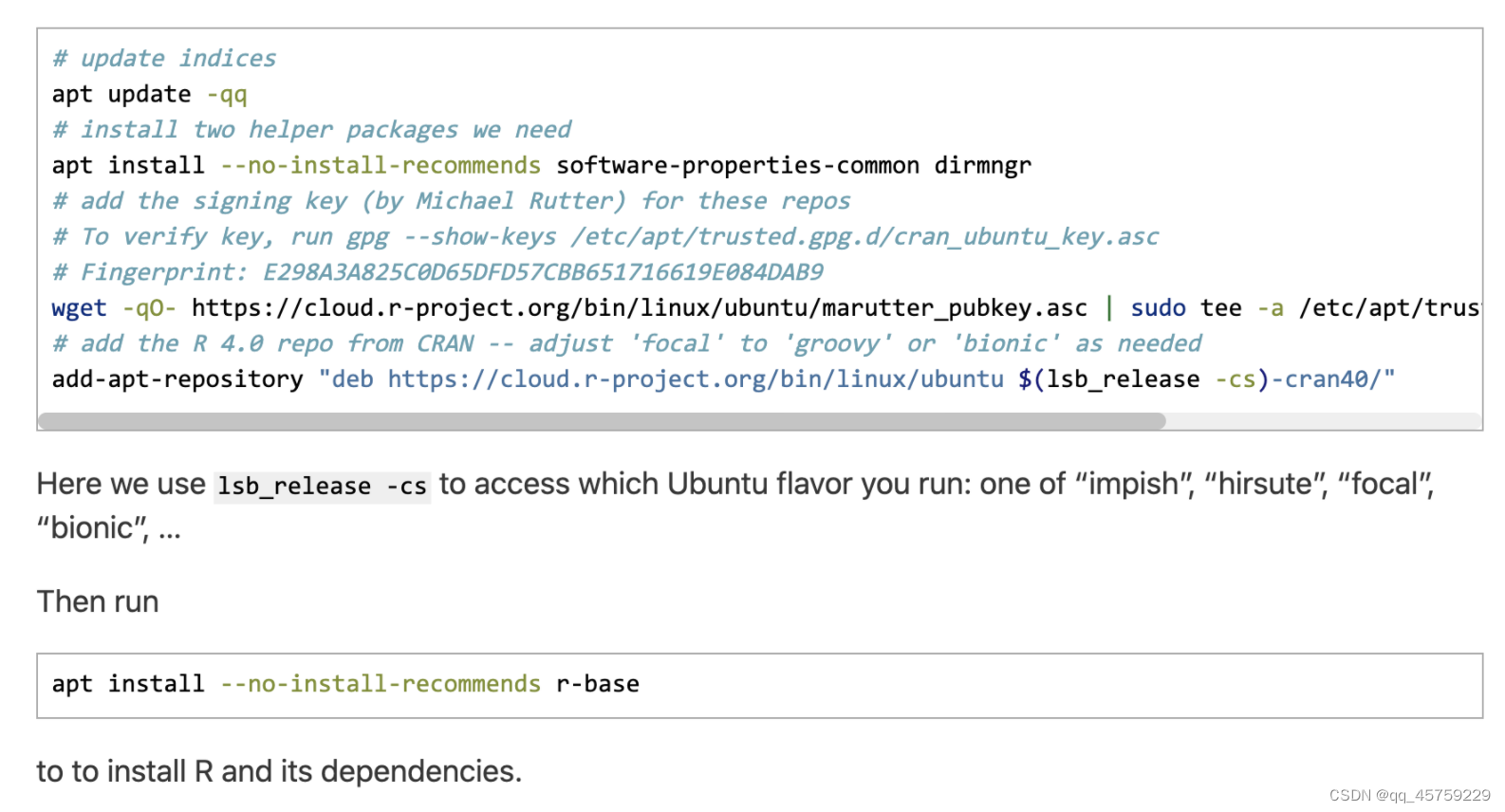
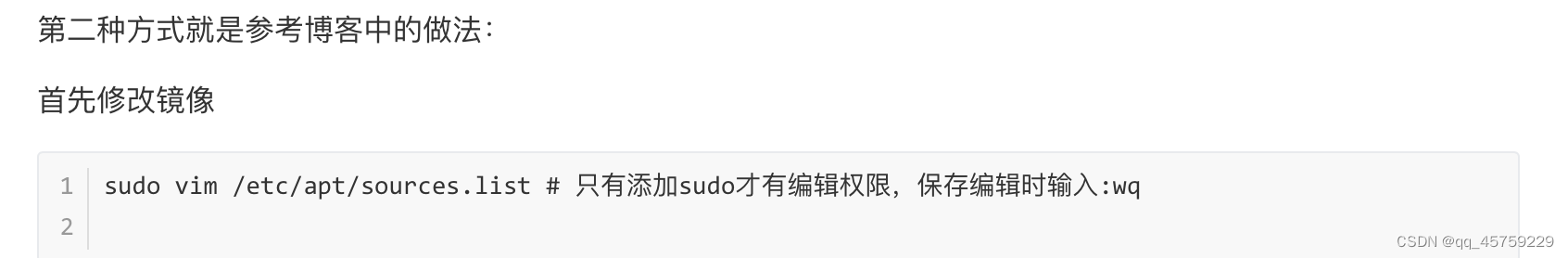

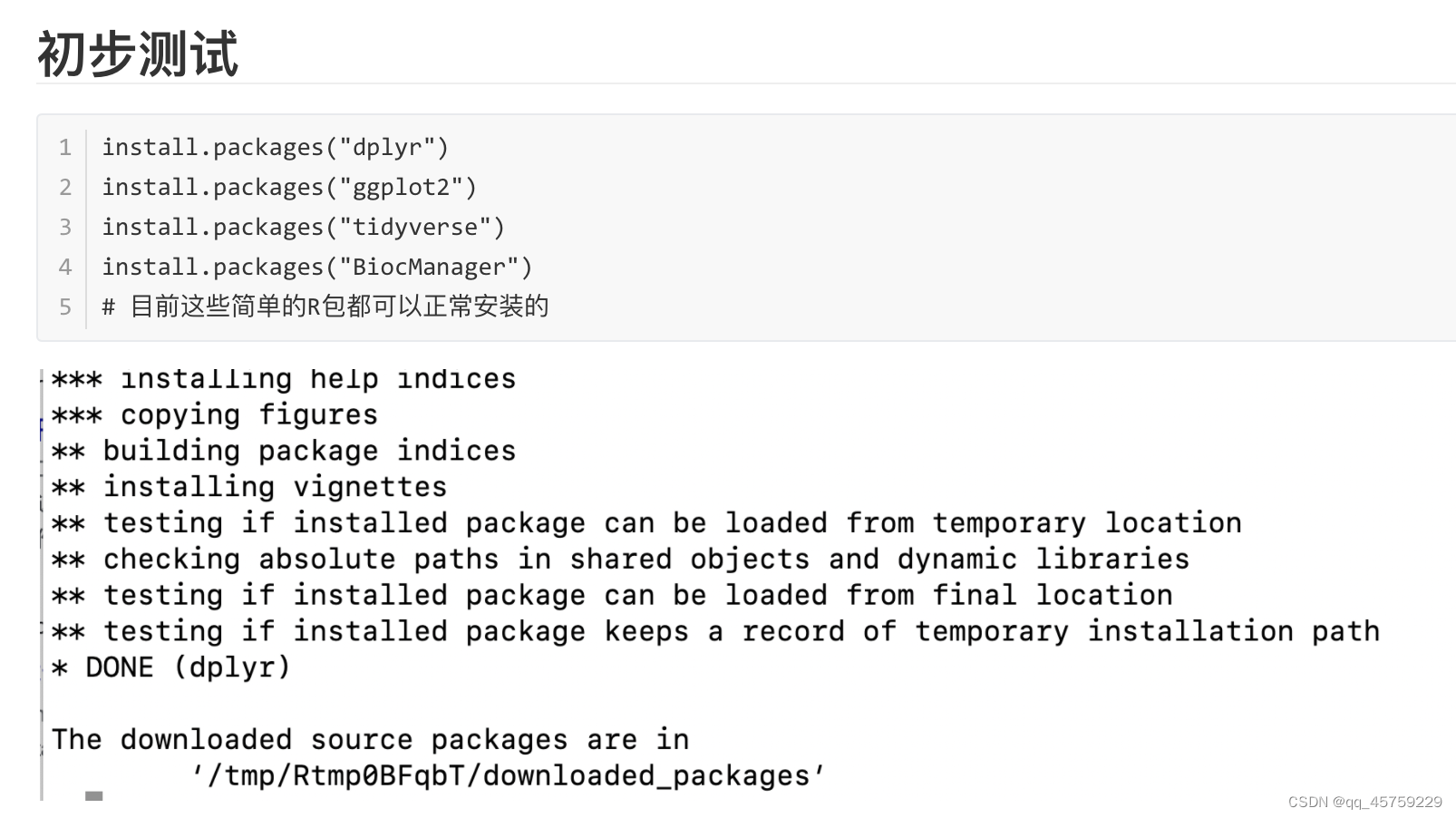

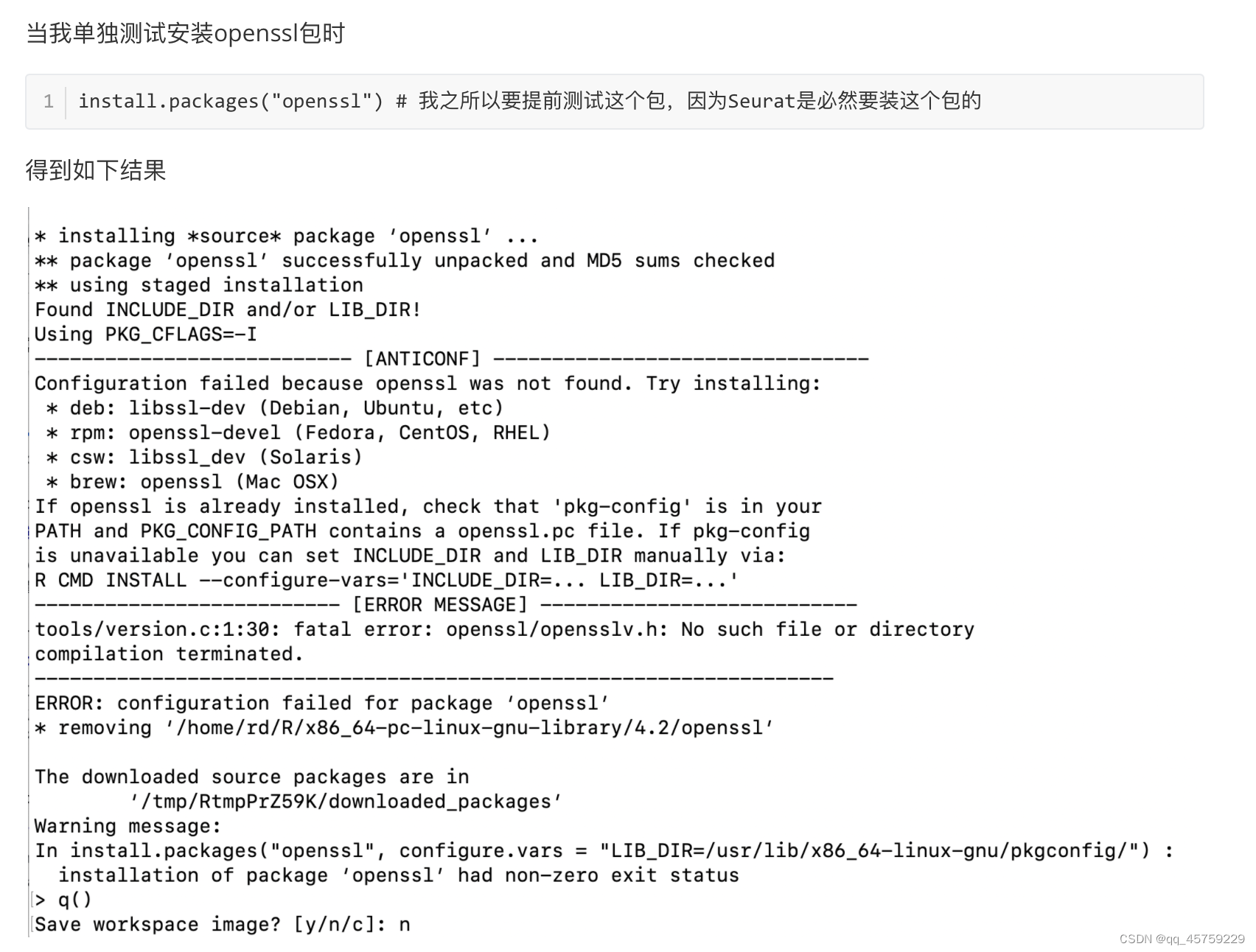
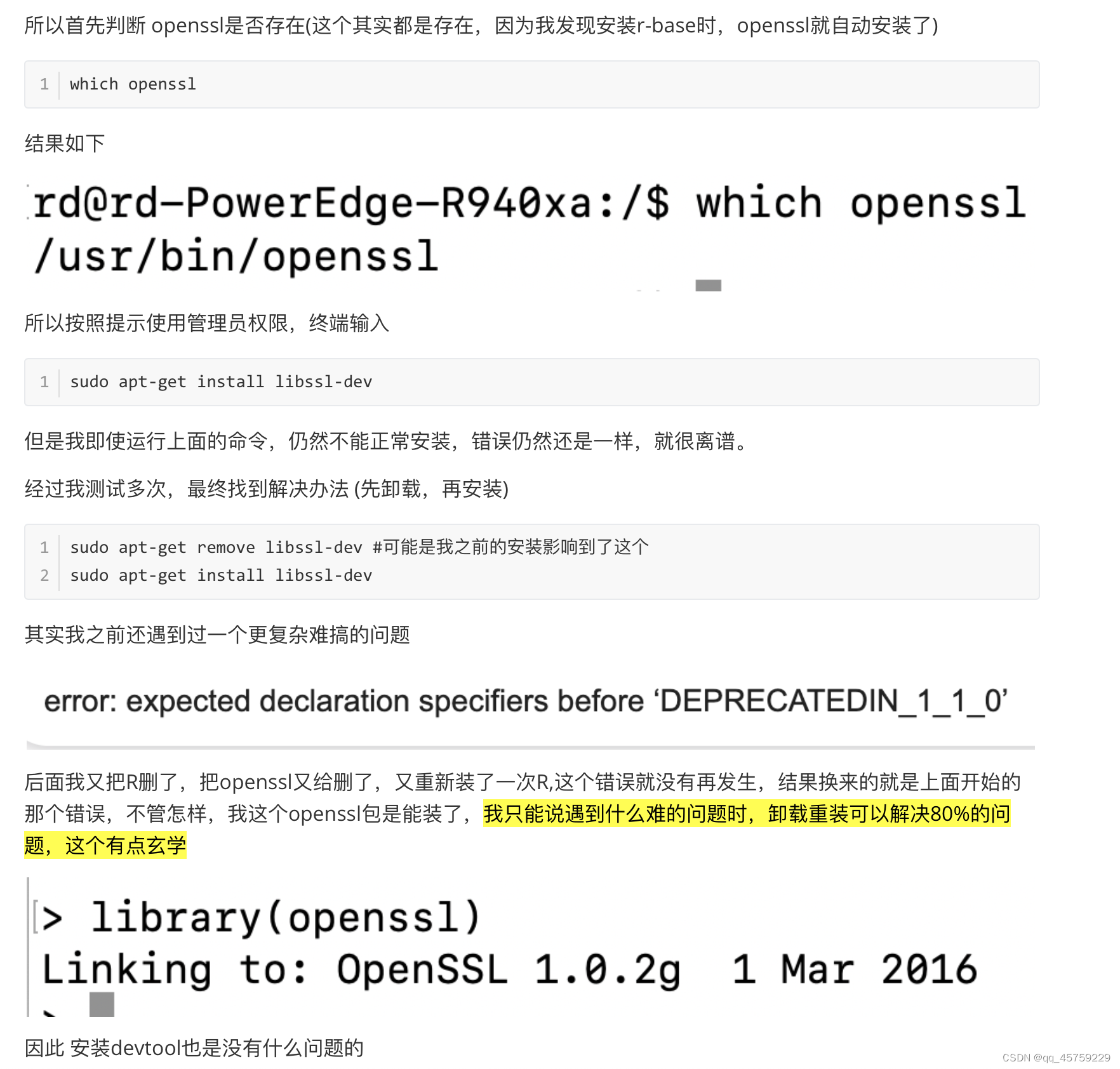




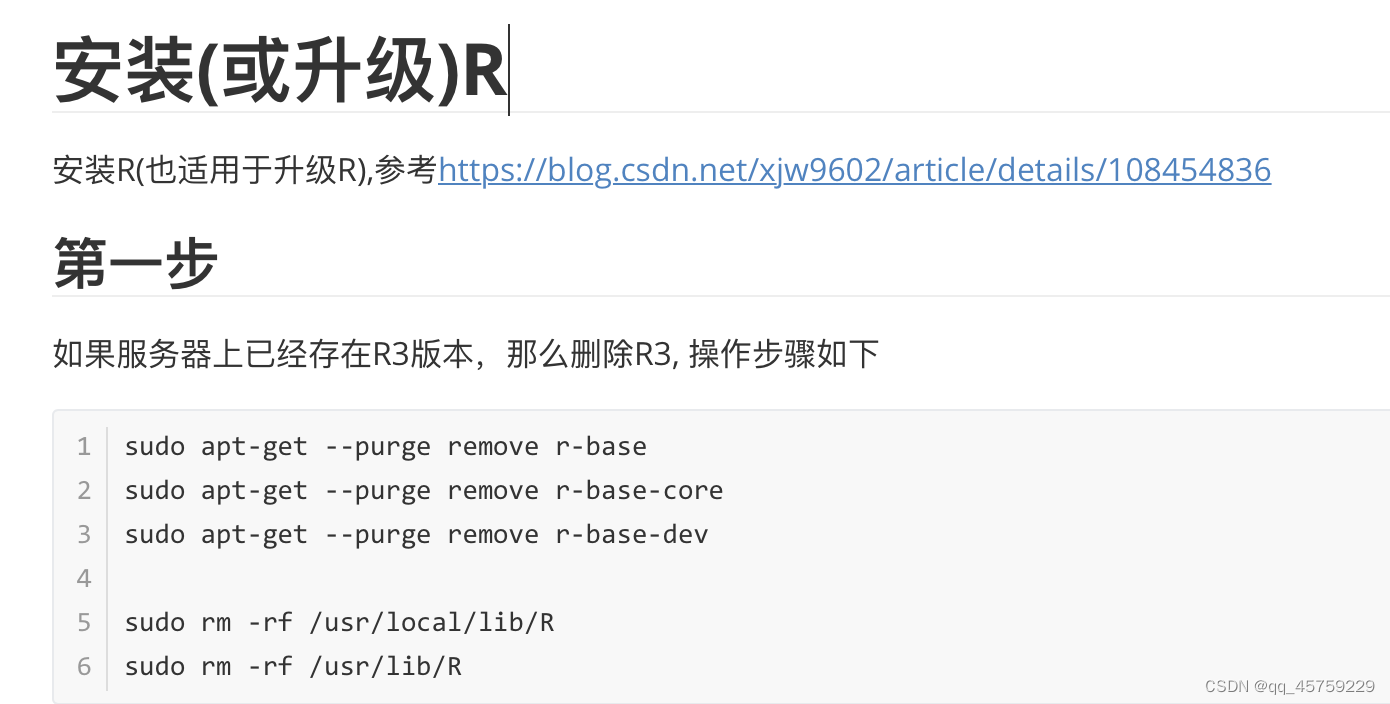
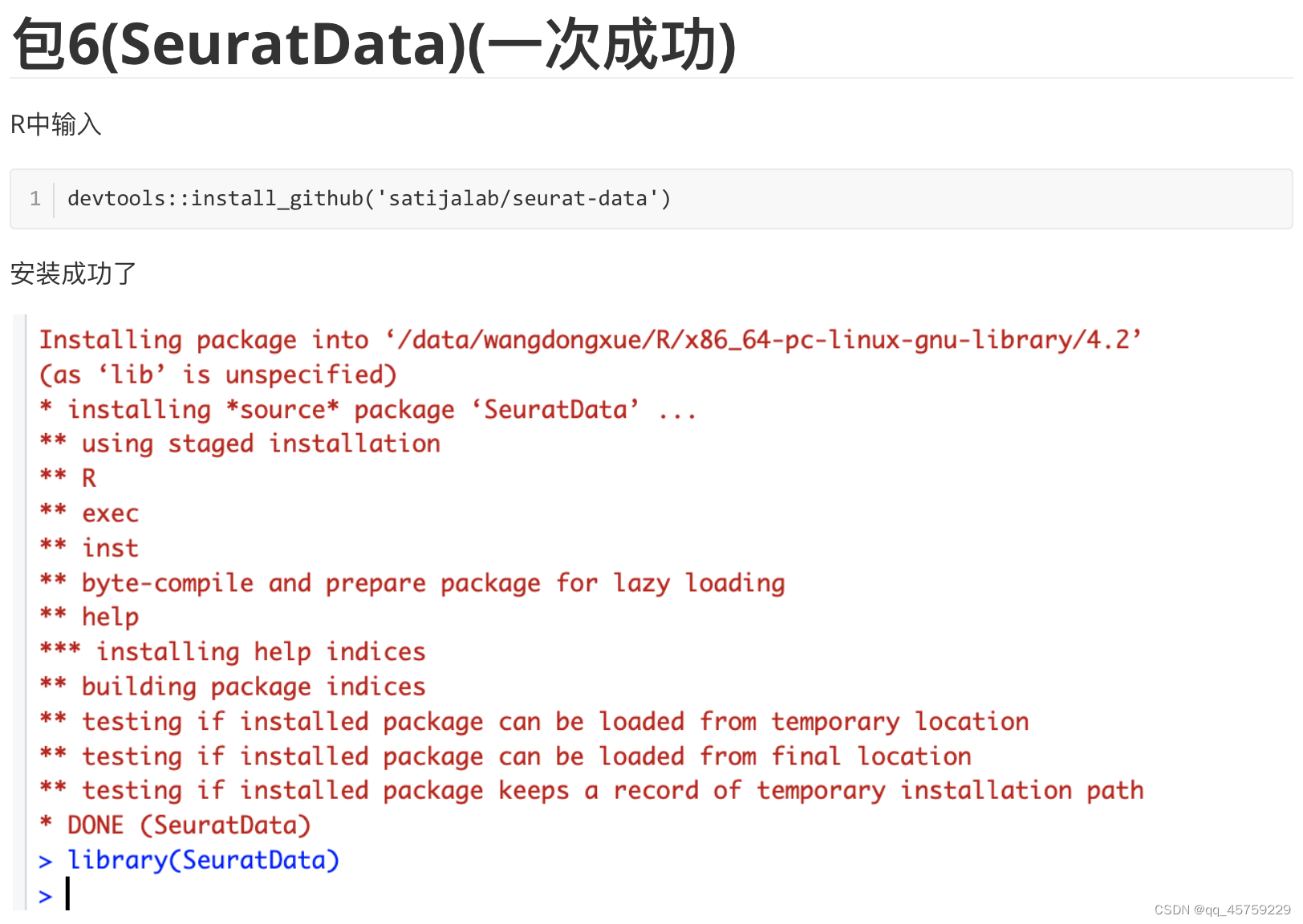
 # 测试Seurat包
# 测试Seurat包
rm(list=ls())
suppressMessages(require(Seurat))
suppressMessages(require(ggplot2))
suppressMessages(require(cowplot))
#suppressMessages(require(scater))
#suppressMessages(require(scran))
#suppressMessages(require(BiocParallel))
#suppressMessages(require(BiocNeighbors))
setwd("/data/wangdongxue/yxk/R学习测试/Seurat3")
pancreas.data <- readRDS(file = "./data/pancreas_v3_file/pancreas_expression_matrix.rds")
metadata <- readRDS(file = "./data/pancreas_v3_file/pancreas_metadata.rds")
pancreas <- CreateSeuratObject(pancreas.data, meta.data = metadata)
#注意这个和其他的情况是不一样的,这里只有metadata的标注信息的
#之前的数据集只是传入count matrix,而并没有同时传入meta.data
# 标准化数据(Filter cells省略了,这个影响不大)
# Normalize and find variable features
pancreas <- NormalizeData(pancreas, verbose = FALSE)
pancreas <- FindVariableFeatures(pancreas, selection.method = "vst", nfeatures = 2000, verbose = FALSE)
# Run the standard workflow for visualization and clustering
pancreas <- ScaleData(pancreas, verbose = FALSE)
pancreas <- RunPCA(pancreas, npcs = 30, verbose = FALSE)
pancreas <- RunUMAP(pancreas, reduction = "pca", dims = 1:30)
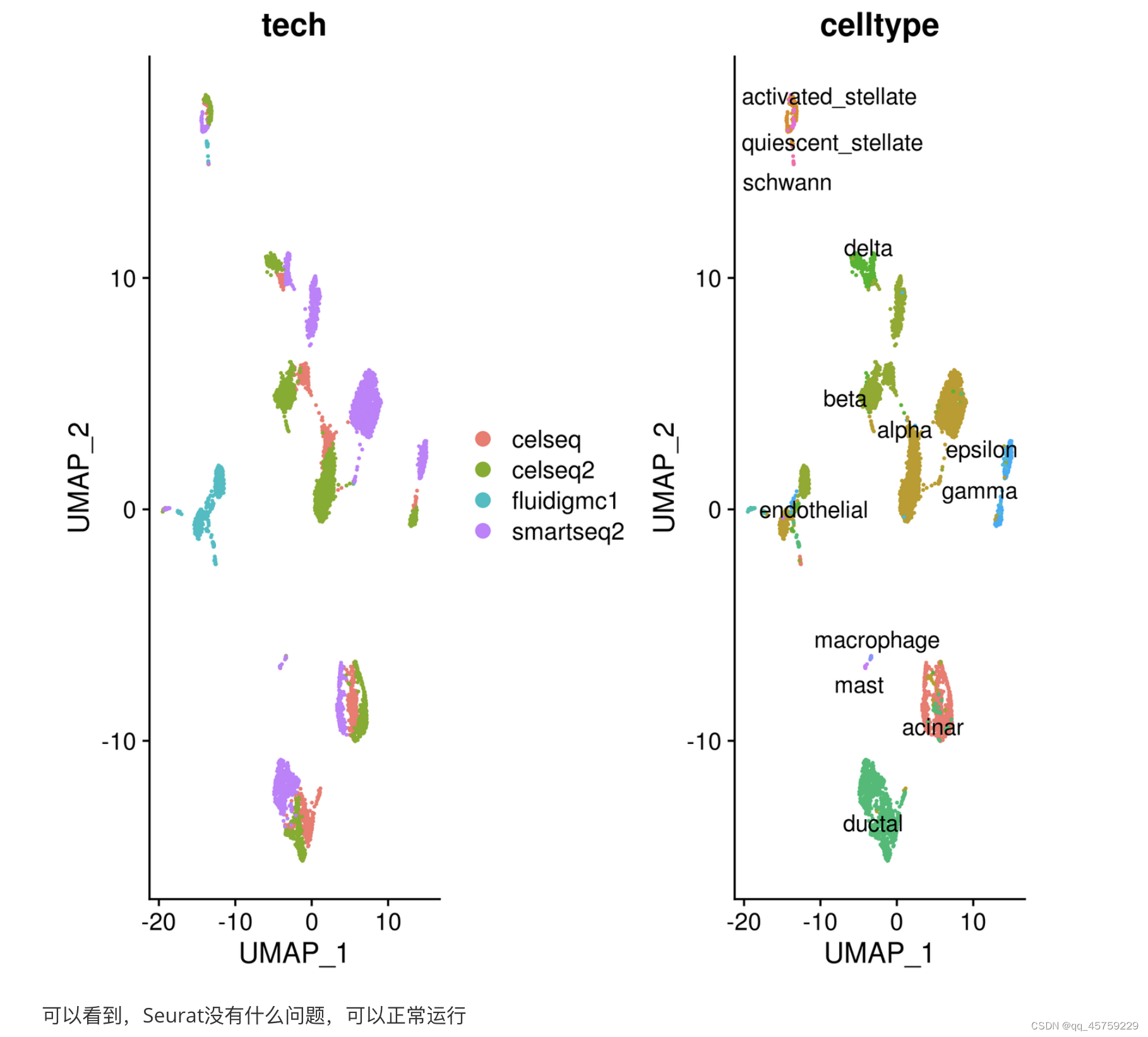
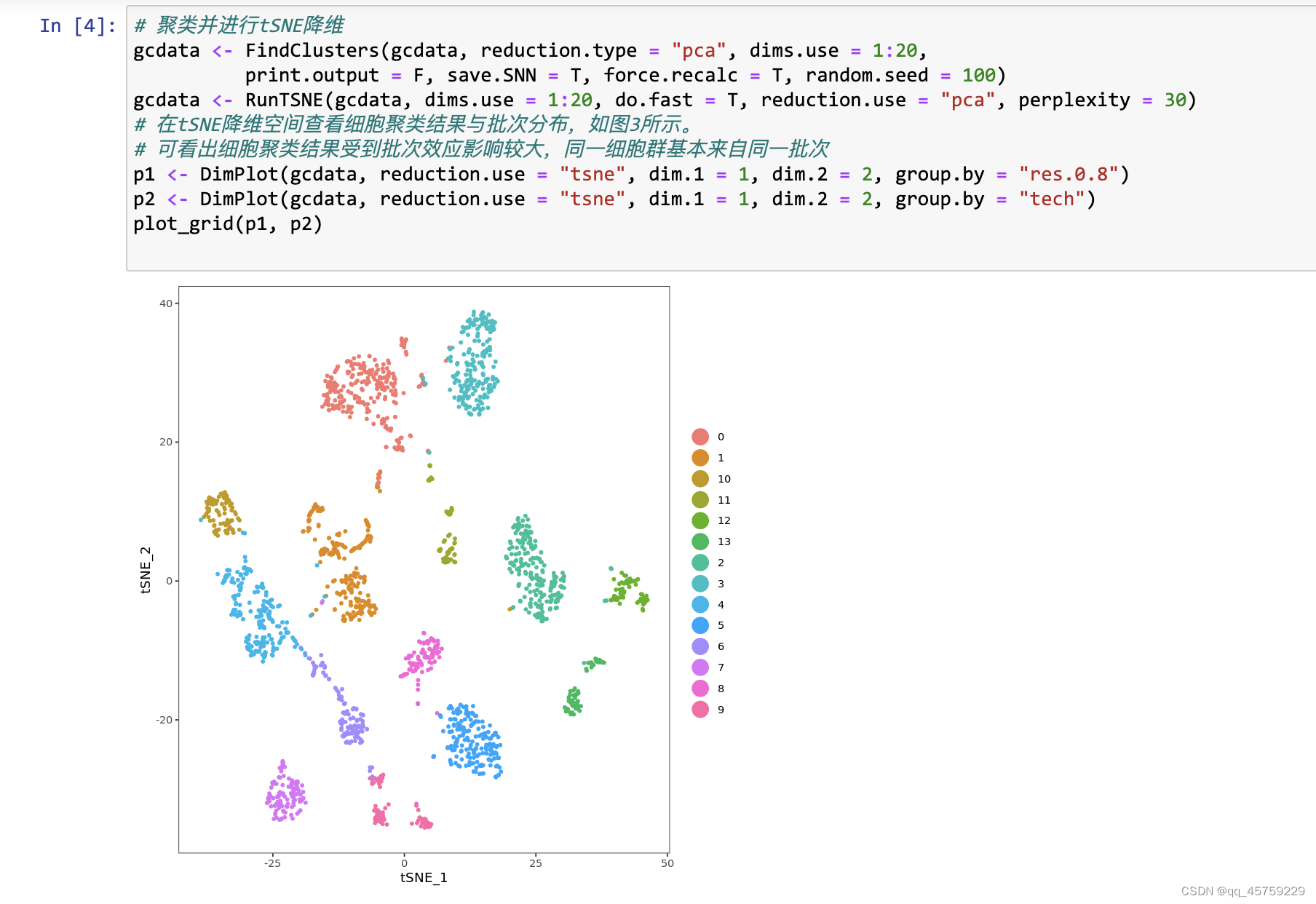
p1 <- DimPlot(pancreas, reduction = "umap", group.by = "tech")#画batch图
p2 <- DimPlot(pancreas, reduction = "umap", group.by = "celltype", label = TRUE, repel = TRUE) +
NoLegend() #画celltype的图
print(p1+p2)
ggsave("vis_pancras.png",plot=p1+p2)
结果如下
remotes::install_github("Oshlack/splatter@RELEASE_3_9")
我最近一次安装发现即使上面的安装仍然是有问题,错误在于locfit包与当前R的版本不匹配,解决问题
install.packages("locfit_1.5-9.2.tar.gz",repos=NULL,type="source")
即可
rm(list=ls())
suppressMessages(library(splatter))
suppressMessages(library(Seurat))
suppressMessages(library(ggplot2))
params <- newSplatParams()
params <- setParam(params, "nGenes", 5000)
params <- setParam(params, "batchCells", c(500,500,500))
params <- setParam(params, "batch.facLoc", 0.5)
params <- setParam(params, "batch.facScale", 0.5)
params <- setParam(params, "group.prob", c(1/3,1/3,1/3))
sim <- splatSimulate(params, method="groups", verbose=FALSE)
## create Seurat object
data <- CreateSeuratObject(counts(sim),meta.data = data.frame(colData(sim)))
#
#注意这个和其他的情况是不一样的,这里只有metadata的标注信息的
#之前的数据集只是传入count matrix,而并没有同时传入meta.data
data <- NormalizeData(data, verbose = FALSE)
data <- FindVariableFeatures(data, selection.method = "vst", nfeatures = 2000, verbose = FALSE)
# Run the standard workflow for visualization and clustering
data <- ScaleData(data, verbose = FALSE)
data <- RunPCA(data, npcs = 30, verbose = FALSE)
data <- RunUMAP(data, reduction = "pca", dims = 1:30)
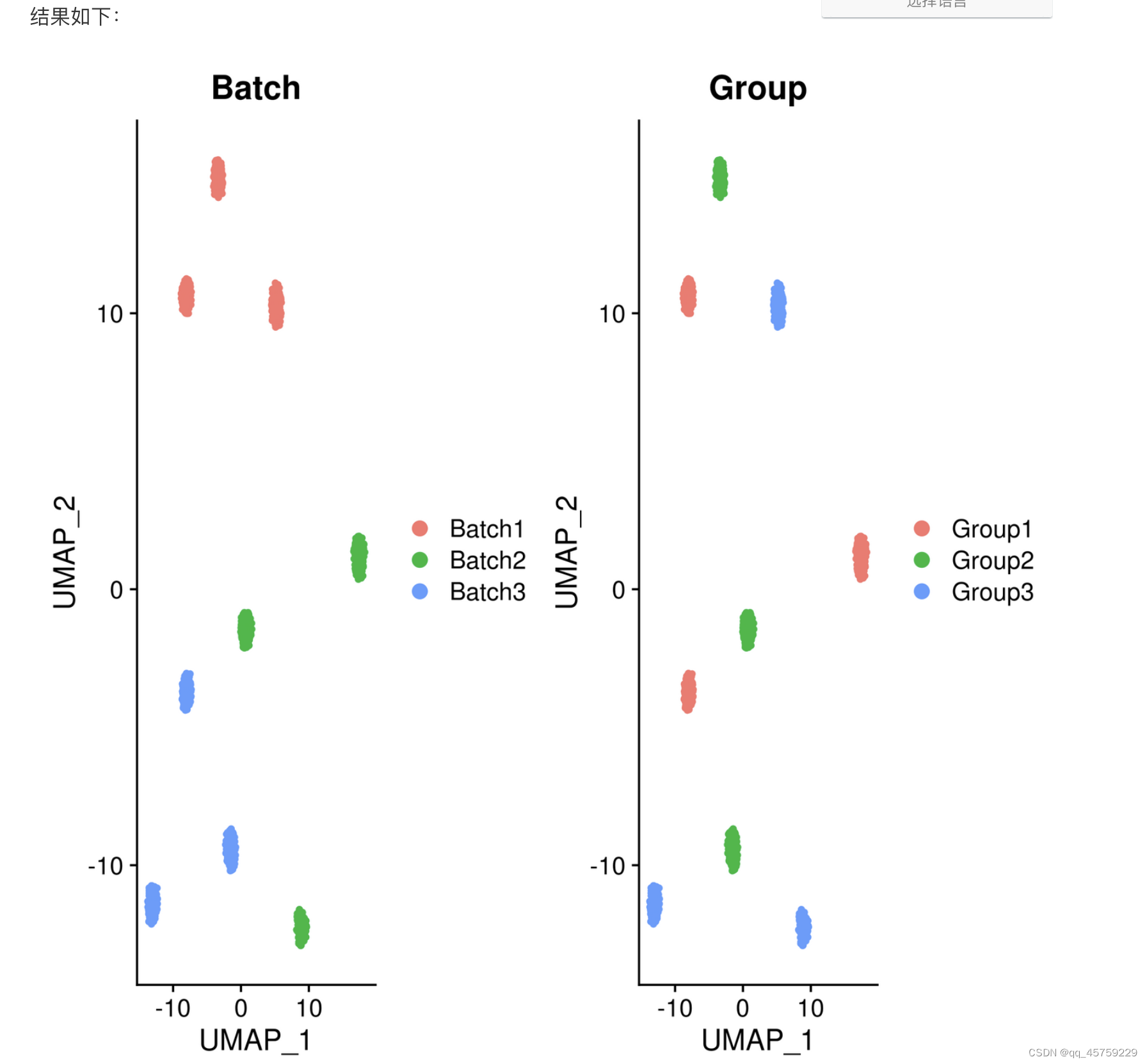
p1 <- DimPlot(data, reduction = "umap", group.by = "Batch")#画batch图
p2 <- DimPlot(data, reduction = "umap", group.by = "Group")
print(p1+p2)
ggsave("vis_splatter.png",plot=p1+p2)
结果如下

 如果在线下载失败,那么使用下面的语句
如果在线下载失败,那么使用下面的语句
install.packages("/Users/xiaokangyu/Desktop/Method_test/Seurat/seurat-wrappers-master/",type="source",repos=NULL)也是可以的

完结,撒花
补充问题1
发现问题
当使用library(reticulate)时,无法导入scipy包,出现以下问题
 解决办法:
解决办法:
https://blog.csdn.net/weixin_44121197/article/details/123018363
#
sudo apt-get install software-properties-common
sudo apt-get update
#
sudo add-apt-repository ppa:ubuntu-toolchain-r/test
sudo apt-get update
sudo apt-get install gcc-4.9
sudo apt-get upgrade libstdc++6
然后问题就解决了
补充问题2
其实这样装完R并不完美,我希望再装一个jupyter的R kernel,但是我上面装的都是主环境的R, 没有办法用jupyter kernel的,所以可以在conda 环境中自己创建一个R4版本的环境,然后加上kernel就可以了,
操作步骤如下
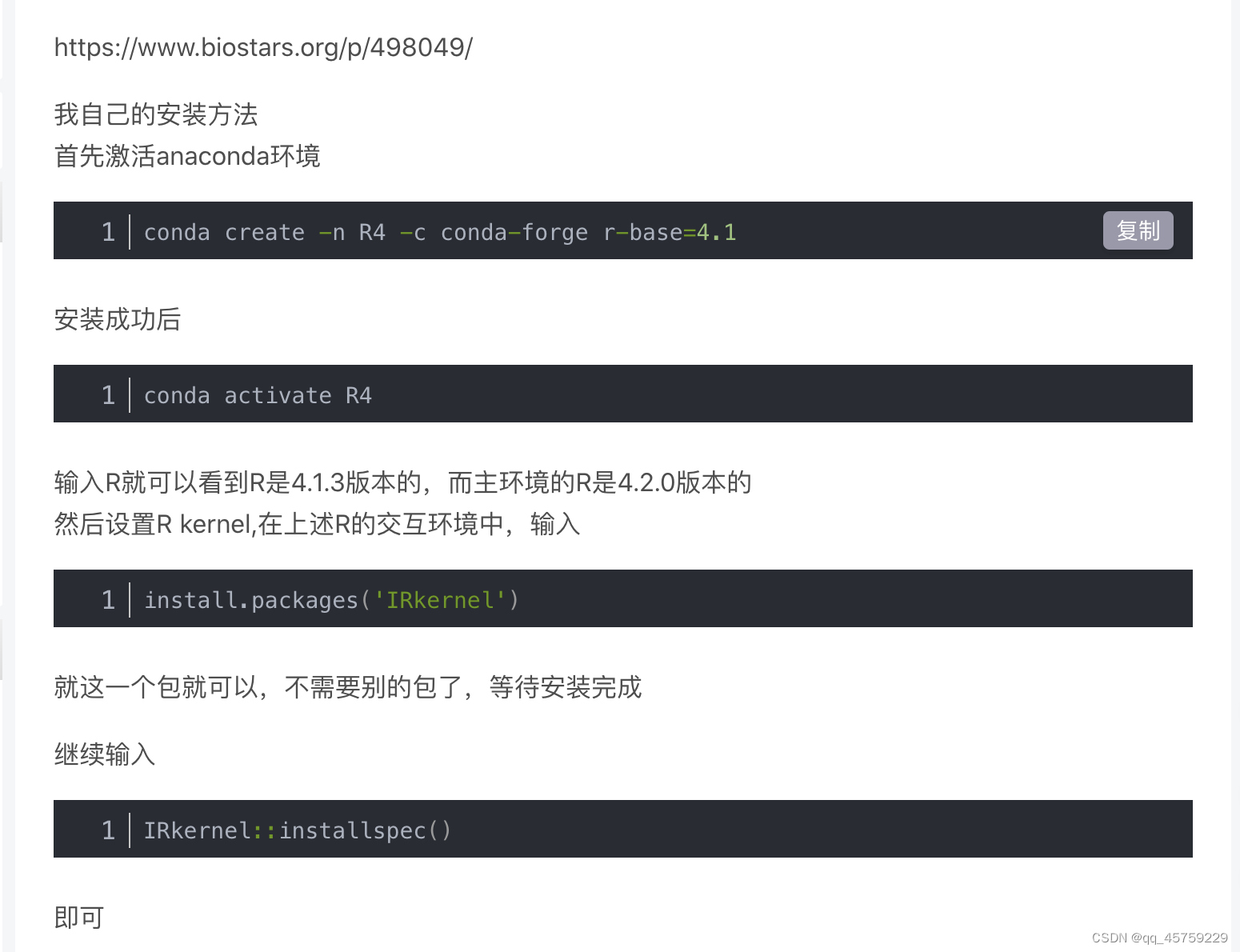
但是我发现在R4环境中装包又会出现问题,我记得主环境中没有这个问题,但是还是要记录一下,
注意现在我是在R4的conda环境中装包,而不是主环境的R,当装devtools包时,发现以下错误
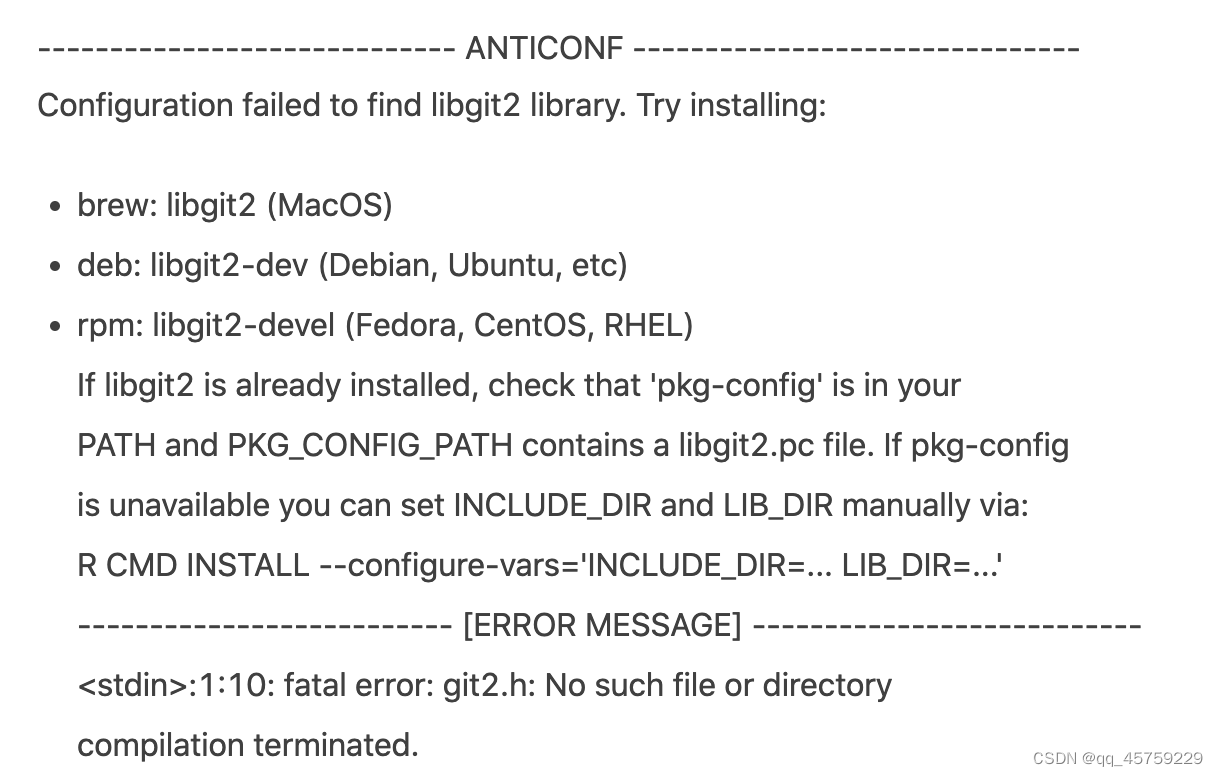 解决办法:在当前R4环境下输入
解决办法:在当前R4环境下输入
conda install libgit2
再补充一个问题
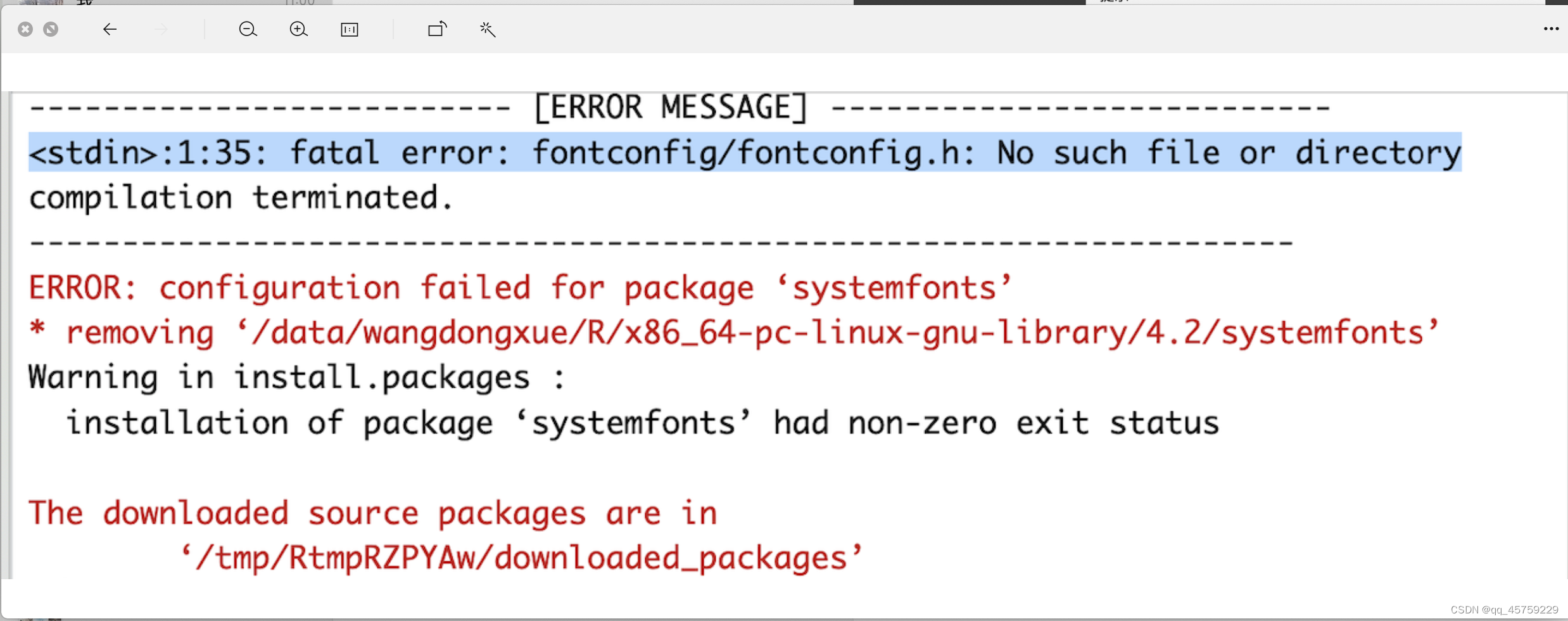
解决办法
https://github.com/r-lib/systemfonts/issues/35
管理员输入
sudo apt -y install libfontconfig1-dev
就没什么问题了
补充stringi安装问题
我今天在mac上尝试自己安装一个conda R4.0.3的环境,因为我的电脑上安装了Seurat2,和Seurat4,唯独没有Seruat3,我想着就试一下,发现还是遇到了stringi的问题
我首先创建了一个conda环境
conda create -n Seurat3 -c conda-forge r-base==4.0.3
安装成功后,
当输入
install.packages("stringi")
总是会发生一下错误,就是下面那个包下载失败
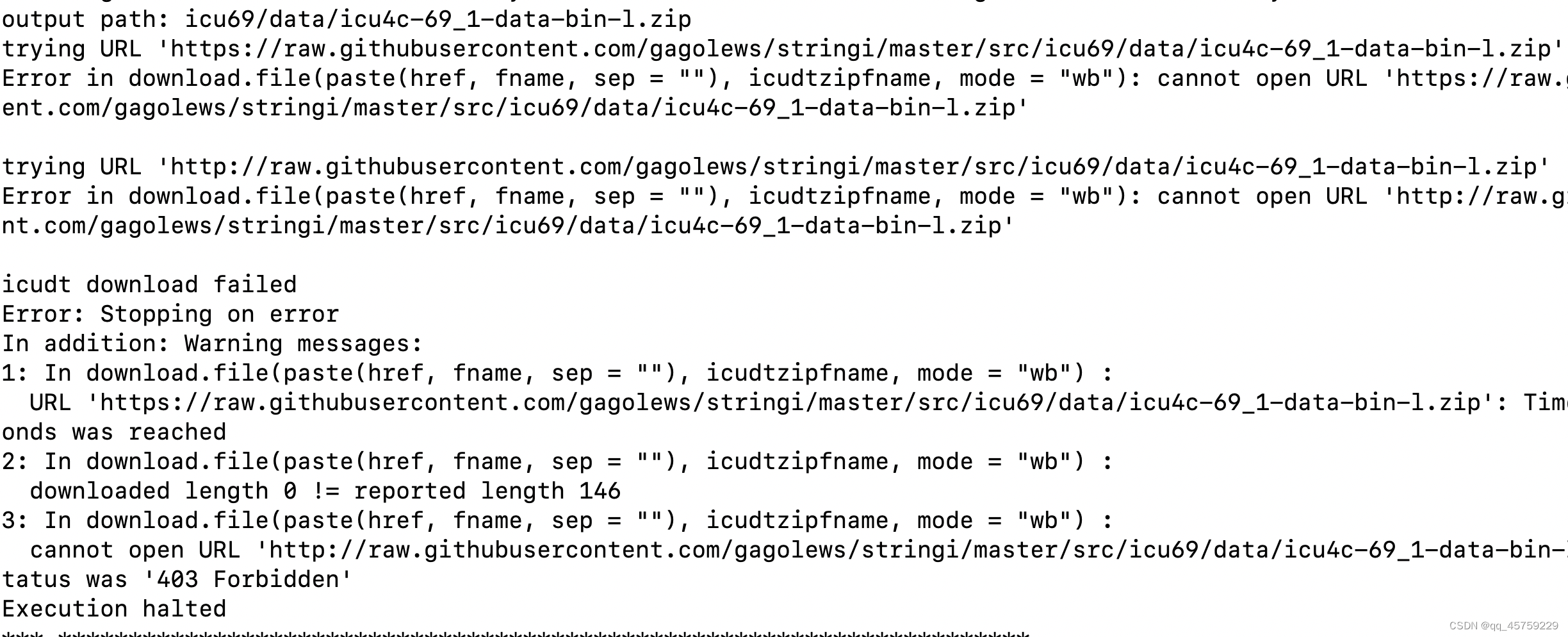 但是我发现在conda环境的R包中,不是这么装stringi包,conda环境有自己的stringi包
但是我发现在conda环境的R包中,不是这么装stringi包,conda环境有自己的stringi包
https://github.com/conda-forge/r-stringi-feedstock
在当前环境中输入
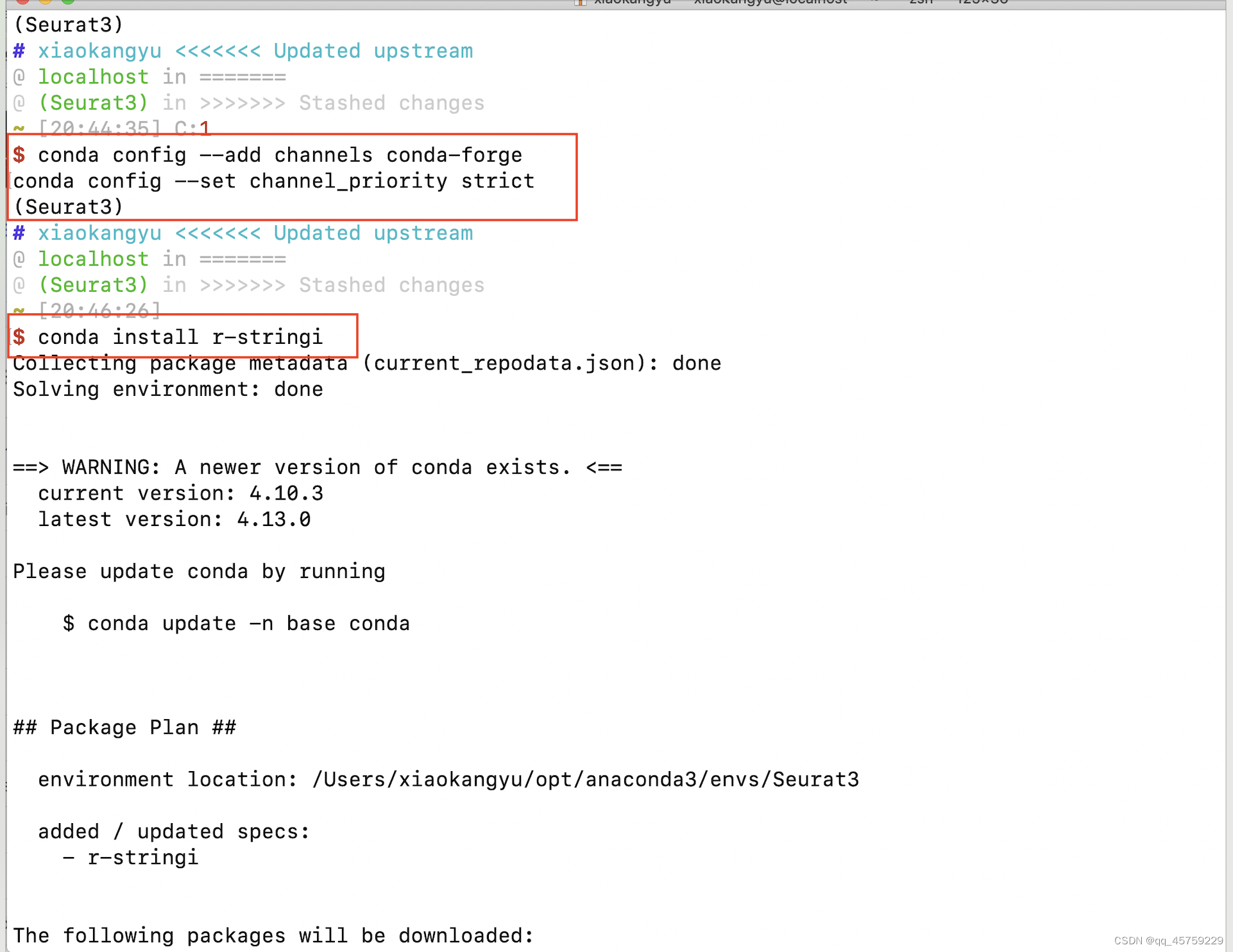

必须提前输入conda config 那句,否则安装还是会失败,不知道是什么原因,很奇怪的感觉, 我待会再到服务器上试一下
安装Seurat3
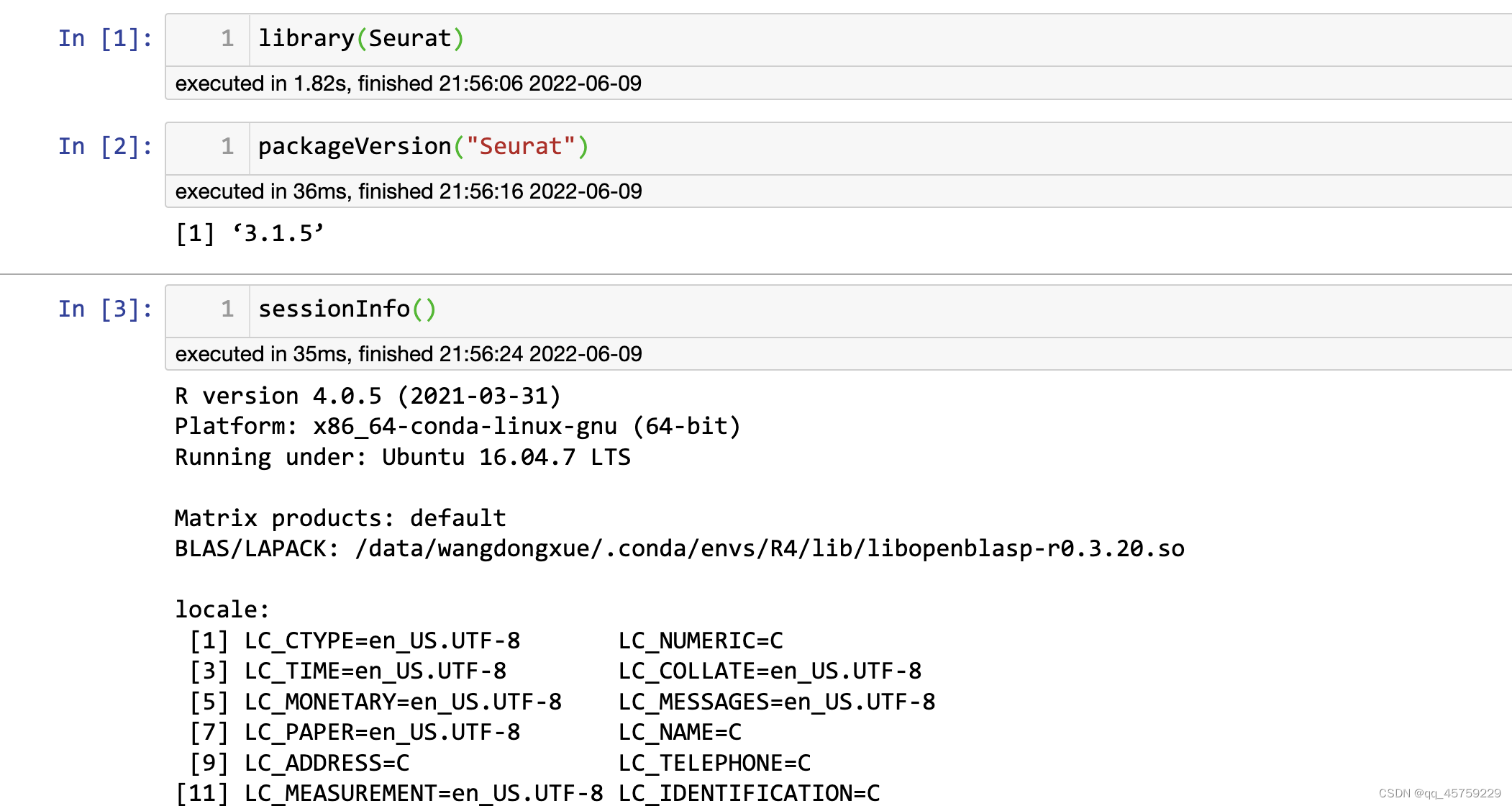
如果想在conda环境中安装Seurat3,那么在R中输入
remotes::install_version("Seurat", version = "3.1.5")
不要安装seurat3.2.3.这个一装就报错,很恶心
然后就是设置kernel的事情了
我之前在服务器上anaconda创建的环境是R4,现在在R4里成功安装了Seurat3,现在我要在jupyter里把这个R4环境的kernel给掉出来
步骤如下
install.packages("BiocManager")
install.packages('IRkernel')
conda install -c conda-forge r-seurat
IRkernel::installspec(name = 'Seurat3', displayname = 'Seurat3')
注意我这里的kernel名字和conda创建的名字是不一样的,不过这个不影响
最终结果如下
rgeos 安装
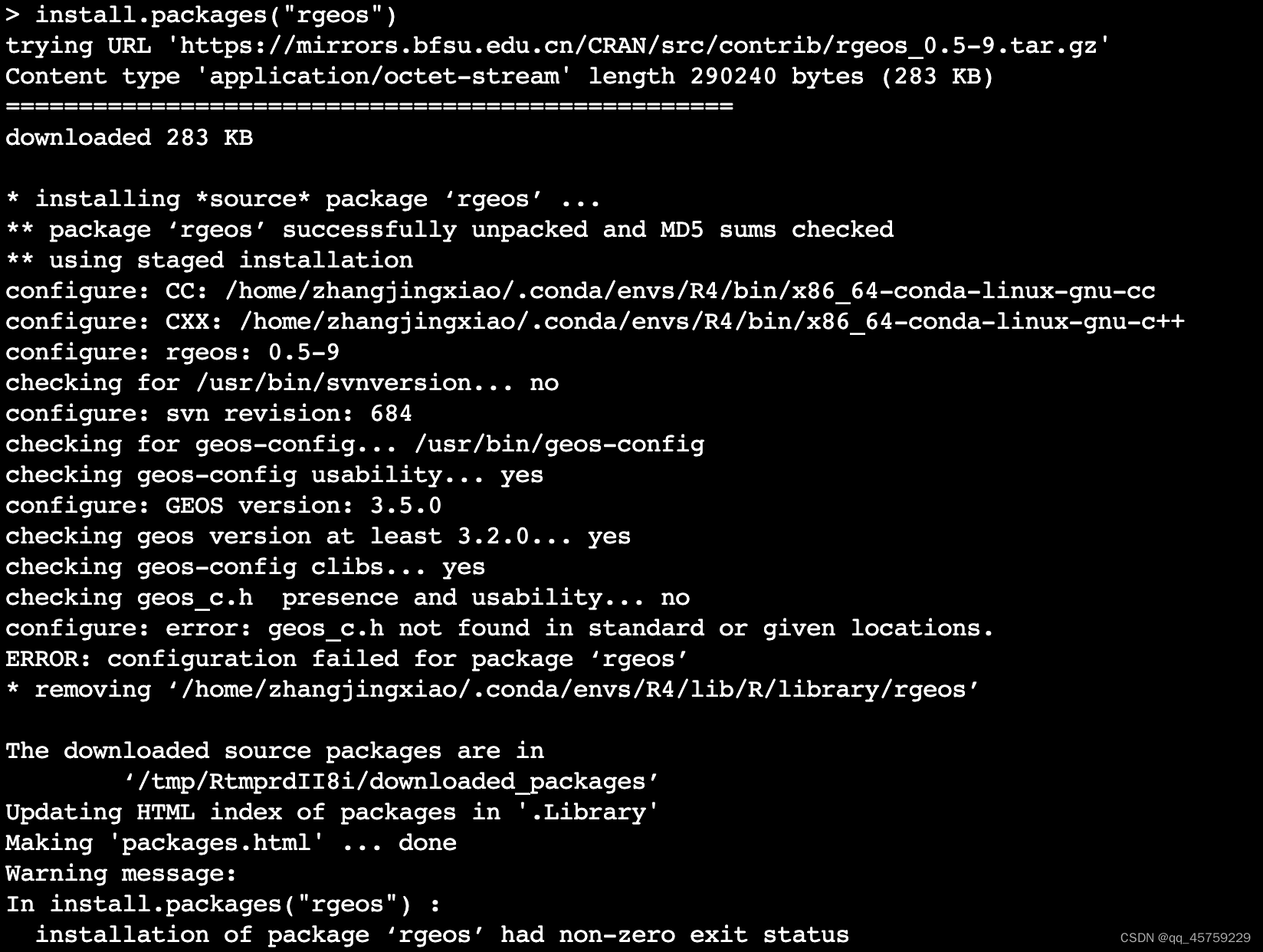 解决办法
解决办法
conda install -c conda-forge r-rgeos
结果如下

conda安装Seurat4
conda install -c conda-forge r-seurat
结果如下
 这个下载的还特别快
这个下载的还特别快
这里需要补充一个非常必要的地方,就是在conda的R环境里添加kernel
我今天尝试了很久怎么在conda创建的R环境里添加R-kernel,一直出现这样的错误
jupyter-client has to be installed but “jupyter kernelspec --version” exited with code 1
怎么弄都不好使,
其中我也是按照网上的教程做的
https://github.com/IRkernel/IRkernel
install.packages('IRkernel')
IRkernel::installspec() # to register the kernel in the current R installation
# 或者
IRkernel::installspec(name = 'R4', displayname = 'R4')
都是一样的错误,
以及我还尝试过
install.packages('devtools')
devtools::install_github('IRkernel/IRkernel')
都是出现问题,
最终的解决方案如下
不要动内部的R环境装IRkernel包,而是要从外部的conda环境中创建,即
conda config --add channels conda-forge
conda config --set channel_priority strict
conda install r-irkernel
不过这个过程估计要等的有点久,大概得半个小时左右,主要在那个solving enviroment,一直在那转,反正执行这几个命令得稍微等一下,然后等正常安装即可,这样也安装好了IRkernel,
然后同样的进入R,再次输入
IRkernel::installspec(name = 'R4', displayname = 'R4')
就可以了
conda环境安装Seurat2
这个装起来有点麻烦,首先我发现并不是说Seurat2过去很久了,好像就必须要求R的版本必须是3.x.x版本的,其实是没有必然联系的,我现在用的就是R4.0.3尝试装Seurat2.3.0
首先还是一样,创建一个R4.0.3的一个环境
conda create -n Seurat2 -c conda-forge r-base==4.0.3
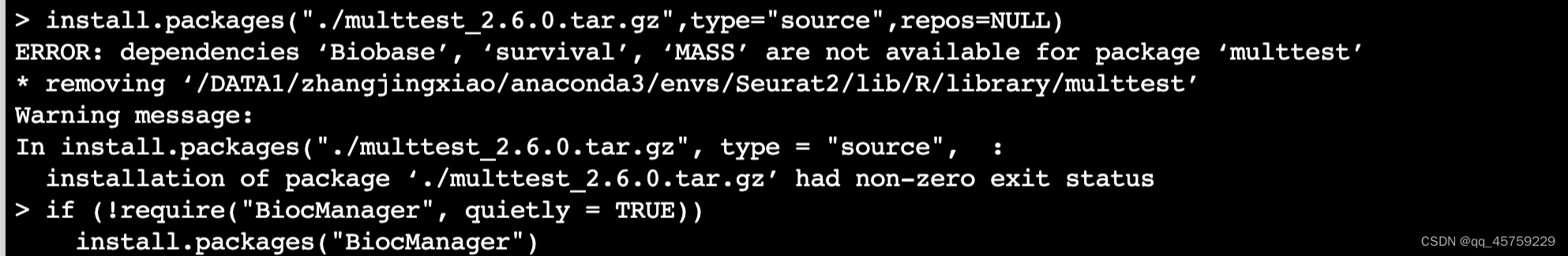
然后注意下载提前下载
-
multtest_2.6.0.tar.gz - R.utils( cran)
- SDMTools_1.1-221.1.tar.gz
如果直接安装Seurat
install.packages('devtools')
# Replace '2.3.0' with your desired version
devtools::install_version(package = 'Seurat', version = package_version('2.3.0'))
还是会出错误的,但是即使我把上面的包安装好了,依然发现有一个包安装不上
metap,这个包的依赖qqconf也是装不上的,但是我发现其实qqconf装不装的上其实不影响Seurat2.3.0的安装,真正有影响的是metap,但是默认下载的metap版本不对,解决的办法是
library(devtools)
install_version("metap", version = "1.4.0")
这个函数我也发现了就是cran安装包并不一定非得安装最新版本的,用这个
install_version函数可以安装cran上包的其他版本的,很nice
但是Seurat2.3.0装完之后,还是有一个问题,就是FindCluster函数,还是会存在问题
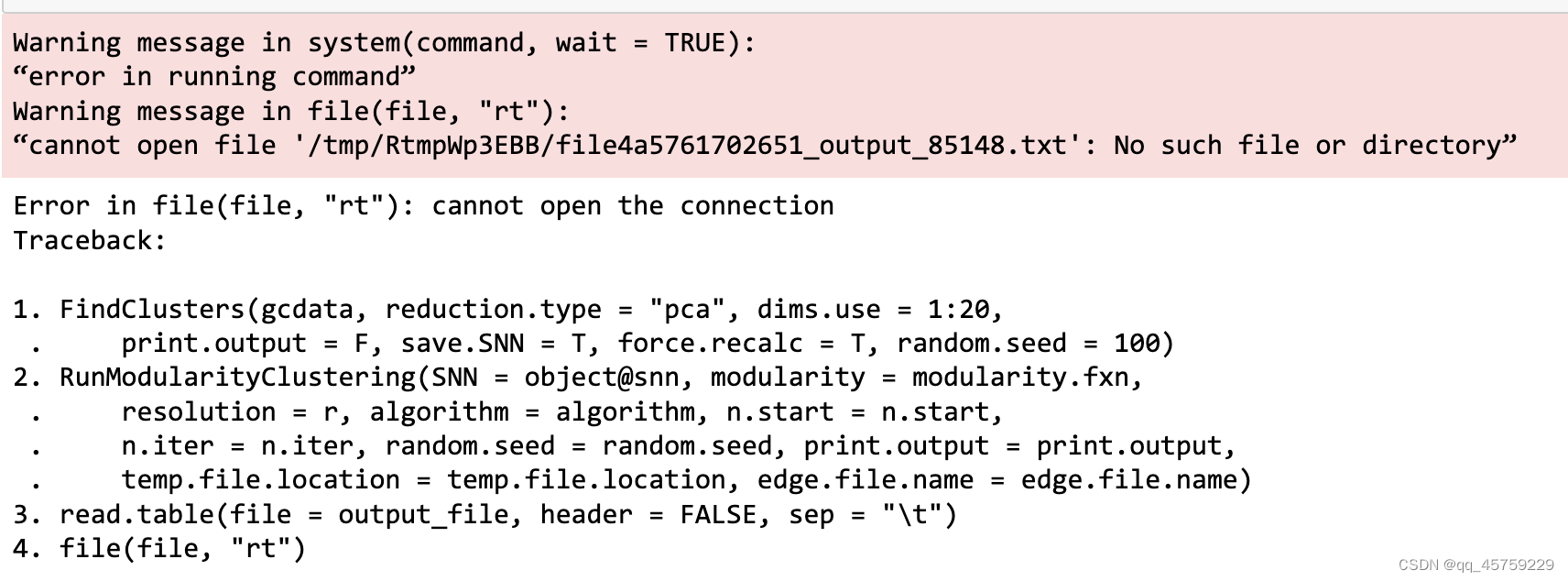 这里我之前遇到过,就是java running time的问题
这里我之前遇到过,就是java running time的问题
问题解决其实很简单,请管理员把java给安上就可以了
最终的测试结果
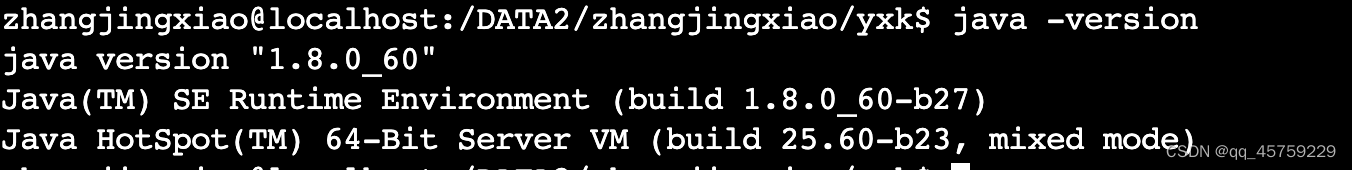 这里有点怪哈,java后面跟的是一个杠,竟然不是两个杠
这里有点怪哈,java后面跟的是一个杠,竟然不是两个杠
,然后Seurat2.3.0里面的FindCluster就可以正常运行了
安装kableExtra包
发生错误
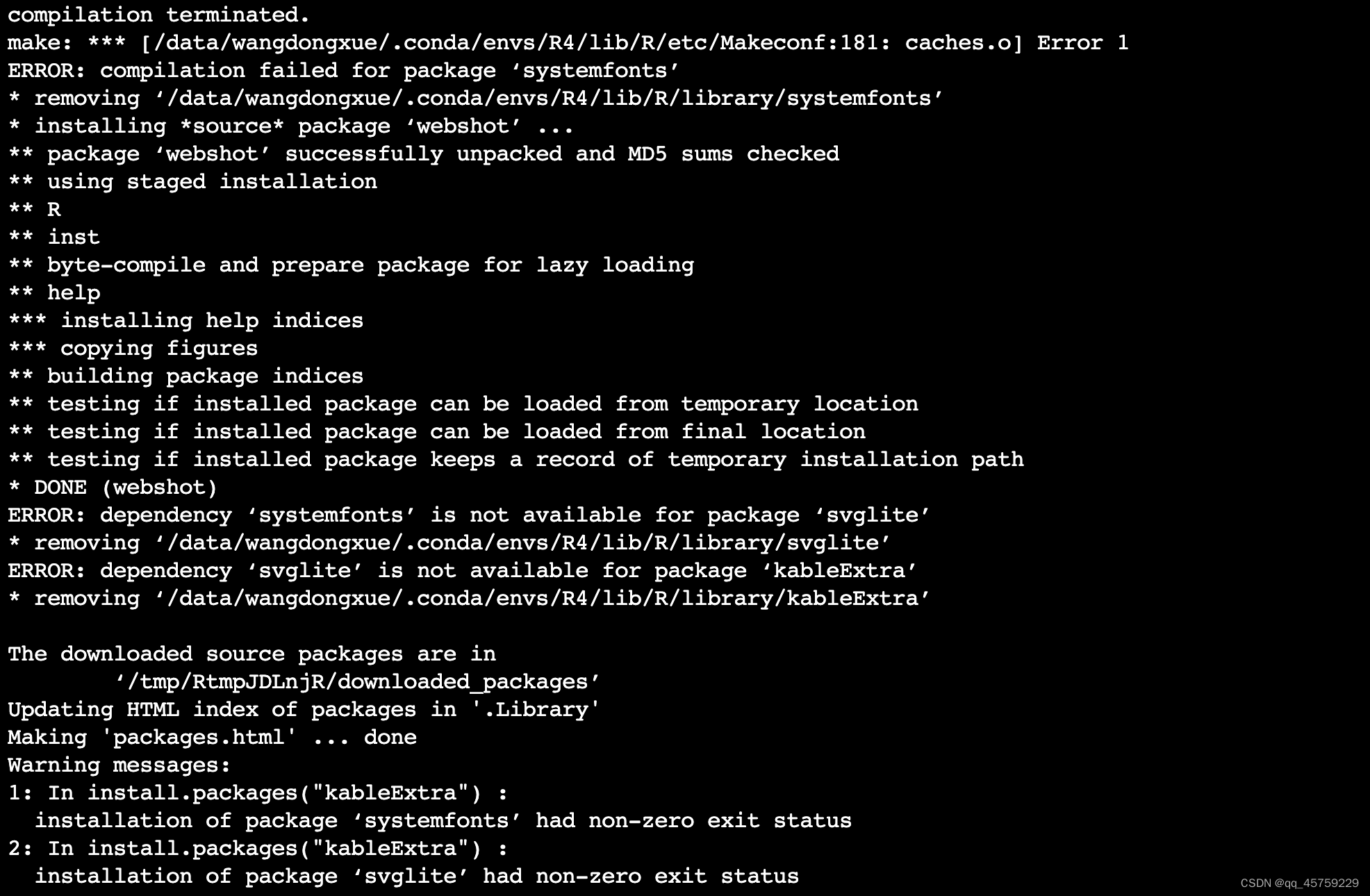 解决办法
解决办法
conda install -c conda-forge r-systemfonts






















 3906
3906











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








