现在基因测序的瓶颈主要在哪里?精度?速度?
做相关研究的朋友测一次序大概需要2周左右的时间( http://news.zhenduan.org/?p=3971.html ),我想问的是瓶颈主要在哪里?
精度:
现在测序的精度有没有问题?
速度:
测序的速度瓶颈在哪里?数据量大?算法复杂?
说白了,目前这个阶段还处于 读长 和 组装精度 的矛盾之中;
不过个人预计3年左右以后,这都不是事儿了;
随着越来越多的IT技术以及数理方法在测序数据分析中的应用,以及软硬件技术本身的发展,组装的精度会越来越高;
同时读长也在成本下降的同时逐步加长,整体上是很明朗的。
准确度(不是精度),读长对基因组的组装有重要影响:
理论上,如果生物基因组没有重复、杂合的影响,即使短序列(30~40bp)的也能够完美的组装出其基因组。但是考虑二者的影响(测序错误造成影响某种程度与杂合类似)则仅仅利用短reads来组装是不现实的,因此还需要大片段文库(bac, fosmid)来辅助组装,而大片段文库主要还是传统的方法,这就是主要的成本所在(1s M~10s M rmb),当然比10年前成本已经大为降低。
3代测序其中一个优势就是解决2代读长过短的缺点,因此理论上,如果reads长度能跨过repeat region的长度,则理论上就能够实现基因组的完美组装,之前有人(可能是Myers,不太记得)认为>10kb的片段足够跨越任意repeat region。除此之外,2代通常在GC含量高或低的区域存在bias(sanger测序也有这样的问题),这样一个后果是造成基因组某些区域无法检测到从而造成基因组的不完整,3代在这一点上也是号称无GC content bias. 当然,虽然3代目前吹的天花乱坠,但是实际应用中的也就PacBio(ion torrent算2.5代),其通量现在无法与illumina比,另外其reads的质量之差可以用丧心病狂来形容(> 15%),其矫正仍需要2代的reads,不过相应的软件太少,而且最要命的是慢的要死(我们自己的项目32 core已经跑了4个月还是没有结果),而直接利用raw reads拼接的软件几乎没有(需要重新设计实现高容错度),因此PacBio目前也只用在少数细菌基因组,老实说,没有人会对细菌的组装感兴趣吧。
最后,以发展的眼光,3代迟早会取代2代,如果真有这一天,会带来很多改变,对搞bioinformatics的人来说,研究基于De brujin组装算法的人会失业,主流拼接算法又会回到overlap-layout-consensus,后者实现起来更简单,速度更快,结果会更好。当然,如果某一天,长度达到基因组全长,搞拼接算法的人会集体失业。
- peter he2015-06-27
三代基于微机电信号测序的技术,基本不需要装配了,当然现在误差10%,离商业应用还有很多路要走
赞回复踩举报
- tao cui (作者) 回复peter he2015-06-27
目前三代读长最多不过几十k,怎么可能不要装配?最多rna-seq可能不用,基因组肯定要组装
赞回复踩举报
- 「已注销」2015-07-10
能否解释一下为什么“研究基于De brujin组装算法的人会失业,主流拼接算法又会回到overlap-layout-consensus”?De bruijn assembly的劣势在哪?
赞回复踩举报
- tao cui (作者) 回复「已注销」2015-07-10
DB的劣势
1. 内存消耗,如果增加kmer的长度,内存会指数增长。
2. DB虽然是不需要设计精巧的overlap算法,但是对read质量要求高,reads需要correction,但是由于二代数据量巨大,这一过程非常缓慢。
3. 2的一个结果导致DB无法使用杂合度高或者错误率高的reads组装成高质量的draft。本课题组之前454测序的项目比较,结果显示,对于高度杂合的物种,wgs assembler组装的质量要远好于一般的DB算法,虽然后者时间上有优势。赞回复踩举报
- 「已注销」回复tao cui (作者)2015-07-11
受教了!!
赞回复踩举报
- lpp2017-02-14
老实说,很多细菌的基因组复杂程度比人类高,人家重复序列密度非常大。现在玩算法的细菌组装都搞不定,大的基因组可想而知。
理想情况下是可以以高精度测足够长,如果有技术可以直接测人类基因组全长,就什么问题也没有了。现在流行的测序方式是二代测序,通量高,价格低,而且很准确(错误率在千分位),但是长度有限,目前最长应该只能到200,再长准确度就没法保证了。所以之后propose了第三代测序,比如Pacific Bio的单分子测序,ph测序,还有至今没有见到实物产品的nanopore测序,三代测序可以测很长,就避免了很多计算的问题,但是错误率很高(Pacific Bio好像是10%),所以还是没办法取代二代测序。
至于速度问题,二代测序基于边合成边测序,之前需要扩增,所以应该主要的限制是DNA的合成速度吧,如果想从根本上有显著提升,只能期待三代测序,使用完全不同的原理。至于后续分析根据测序要研究的问题就有很多情况了。
一代:通量太低,要拿到足够的数据就非常费钱
二代:通量大,特别是Hiseq,一个lane跑下去估计够一代的跑好几个月甚至一年,所以相对来说成本低很多;但是读段太短,要拼长很困难,所以瓶颈主要在拼接组装的算法上,因为拼接往往比测序要费时费力的多
三代现在貌似还不成熟,听说错误率比较高
从测序仪上讲:
第一代的测序方法,主要是sanger测序,准确性高,测序长度好(1K以上),但是测序通量低,而且价格昂贵。
第二代测序仪,主要有illumina的,solid和454几个常用的测序平台。第二代测序仪最大的有点就是价格便宜,通量非常高,准确性99%。
但是illumina主要的问题是测序长度短,100bp以上错误率就会大大提高。另外两个可以更长,但是成本也略比illumina高。短序列的reads在做基因组装的时候,遇到大的重复片段就会很麻烦。
第三代的测序仪,即所谓的单分子测序,可以测的长度很高,但是会引入第二代测序很少出现的indel(插入,缺失)的情况。
从数据分析上看,高通量,大数据的分析,在计算机的存储和计算资源的消耗上都是很高的。
从实验室提取DNA,但最后得到分析结果,中间需要经历 建库-测序-比对/组装-变异检测-注释等 一系列实验和数据分析过程,第二代测序的方法动辄单个样本就上T的数据量都会使得分析过程耗时耗资源。
作为学院测序第一人!( ̄□ ̄||开玩笑的,学院测序的牛人不是去华大就是去北大了。。。)我可以很负责任的告诉题主,目前基因测序的瓶颈不在精度,也不在速度,而是“钱!”。确切的说就是在保证一定精度和速度的条件下,想尽办法减少测序成本。这才是现在基因测序要做的。下面放张wiki的图:
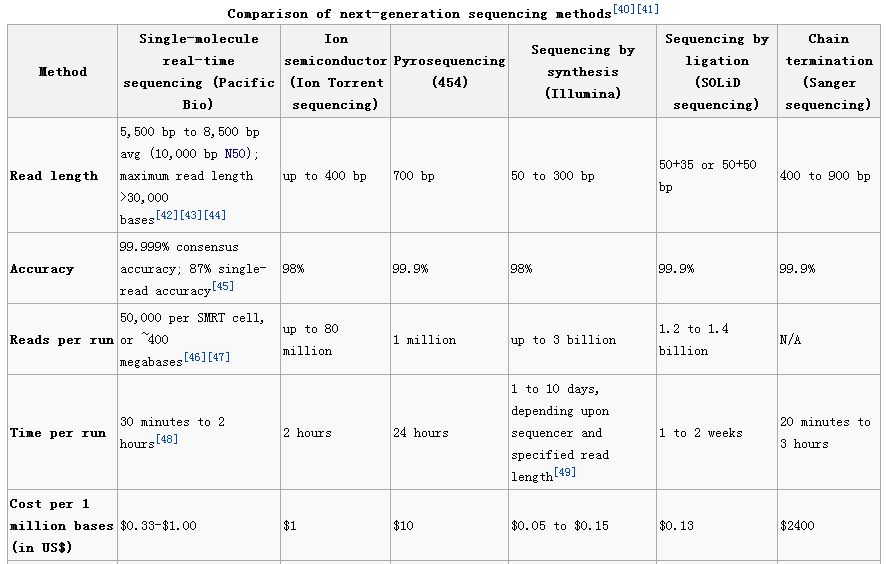
图中可以看到,现在大部分的测序方法的准确率已经达到99.9%,低的都能达到98%,准确率已经不是问题。而速度这个参数与钱比,根本不值一提。打个比方,你想要测测自己的全基因组,现在有两个公司,一个7万刀一小时给你做好,一个1万刀7小时给你弄好,你选哪个?为什么现在Illumina的东西卖的最好,你看上面的图就知道,因为便宜!($0.05-0.15 per 1 million bases)
那为什么还要开发新的方法?因为还不够便宜!现在你去给自己用Illumina的东西去测序,一个人是48000刀(当然团购有折扣的,低至一折哦)。
但是我们的目标是——1000刀一个人!希望在不久的将来,人人都能有自己的基因组数据,到时候人们互相吐槽的时候就会说,“我*,你怎么生的?比我多一个AT碱基对啊!”
目前来说,对于第一代测序,成本主要在于试剂,数据处理占小部分。对于第二代测序,成本主要在建库,PCR和引物,测序和数据分析也占大约30%。关键是每一步都要花不少的钱,加起来就贵了
第一代测序,成本在人力上,太耗人力了。
现在大部分的测序方法的准确率已经达到99.9%,低的都能达到98%,准确率已经不是问题。
不可以这么说的。这个0.1%的错误率其实说明不了什么问题。
到了第三代,成本下来了,但是误差也来了,10%,所以测序的路需要IT技术的帮助,才会普及。
作者:徐光启
链接:https://www.zhihu.com/question/20479004/answer/251678760
来源:知乎
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。
复制自己的专栏文章来回答基因测序中的关键问题。
本文主要阐述和对比基因芯片和下一代基因测序技术。
有监督基因芯片检测原理
❖原理与酶免ELISA类似:样品制备(核酸分离与复制)-核酸标记-杂交-洗片-图像处理
❖定性(或半定量分析)
❖灵敏度与特异性是基因芯片技术中最核心的问题之一

基因芯片制造技术简析
基因芯片根据制备技术不同,可以分为两大类:cDNA芯片和寡核苷酸芯片(Oligo)
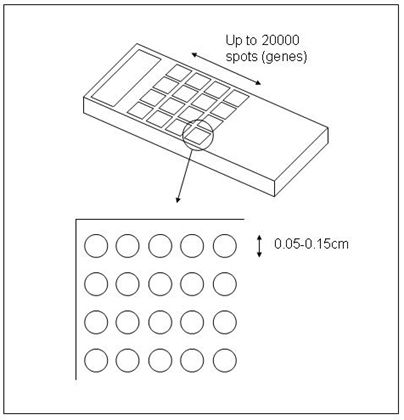
cDNA 芯片
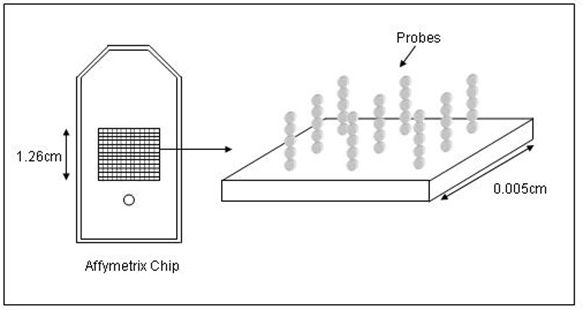
寡核苷酸芯片(Oligo)
cDNA芯片特点:
以玻片为载体,cDNA为探针;
成本低廉;
相对oligo有定量优势;
实验程序复杂;
可重复性差;
Oligo芯片特点:
以硅片为载体;
稳定,质量可控;
实验程序可标准化;
工业化程度高;
Oligo芯片是基因芯片制造技术的趋势。
基因芯片VS下一代基因测序(RNA-sq)

国际学术界人士对基因芯片VS基因测序(RNA-sq)看法
’The only wasted microarray data are those that are of poor
quality or where there is insufficient data (MIAME or clinical annotation) to
allow their appropriate use. There are certainly plenty of worthless microarray
data sets out there but these were poor before the advent of RNAseq.’
Prof.Robert Clarke, Georgetown university,2014
’Many people do profiling to get an idea which known genes and pathways might be
involved in a biological response (to a treatment, to a disease, to a cell development...).
Microarrays are perfectly fine here. ‘
Prof.Jochen,Justus-Liebig-Universität Gießen,2014
‘We have switched completely to RNA-Seq. It provides quantitative
and comprehensive expression data.’
Michael Iadorala, National Institutes of Health,2014
‘For all the results generating from each steps in the procedure of microarrays
technique, the possible standarderror of the means ranged from 5-10%. When you multiple these 5-10% 3 to 4 times (steps), the final possible standard error of the means will be completely unreliable. ‘
Pro. MH Zhou, University of HongKong, 2014
‘I would choose next generation sequencing for my next expression profiling study.
The cost is dropping fast and its unbiased nature are indeed preferable over microarrays’.
Prof. Oliver, Stanford University, 2014
学术界对基因芯片的未来持不乐观态度。但是,这些学术人士是从基因研究的量化和准确性的需求而做出的判断。
基因芯片技术面临的挑战
需要自身技术提高的关键点:
❖1. 提高基因芯片的特异性;
❖2. 简化样品制备和标记操作;
❖3. 增加信号检测的灵敏度;
❖4. 高度集成化样品制备、基因扩增、核酸标记及检测仪器的研制和开发。
来自基因测序技术的压力:
❖1. 基因测序技术价格几乎每六个月降价一半;
❖2. 基因测序相关操作和数据分析标准化进展很快,为以后大通量分析奠定基础;
❖3. 更广泛的全基因表达应用;
基因芯片的发展趋势:会不会被下一代基因测序(NGS)取代?
❖在学术研究领域,尤其以下分支,NGS因全基因测序,特异性灵敏性高和快速走低的价格会在10-15年取代基因芯片技术
染色质免疫沉淀(Chromatin immunoprecipitation)
基因表达(Gene Expression)
细胞遗传学(Cytogenetics)
❖但在以下应用领域,基因芯片技术10-15年因简单操作,标准化,通量大和暂时的价格优势而无法被取代(就像分子诊断暂时无法取代elisa一样)
基因分型(Genotyping)
临床诊断(Diagnostics)
测序只是最基础的一环,测完后的解读才是最艰难的点,因为你不能解读就找不到应用的出口。
目前的测序技术就是让你知道碱基序列是什么,但这些碱基序列如何影响疾病,影响表达,人类只搞懂了1%。
作为一个在海外从业10年的生信民工。算是见证了这个行业的发展。
速度肯定不是问题了现在,精度有待提高。感觉最大的瓶颈是测序长度。
目前assembly靠计算机算法,如果哪天测序长度增加的话,assembly的精准度会提高很多。
应该说发展到后期已经不是测序技术的突破了,应该是倚重大数据,算法这些的。
如何在数据里提取有效,且有生物学意义的数据,如何运用这些数据知道疾病诊断,药物开发等。
其实这个问题,我想简单的说一下:
一代测序,也就是sanger测序,是最普通的测序的方法,这个方法的原理很简单,最基本的应用就是获取一段基因的序列信息,其实题主说的测序应用很少应该不包括这个方法,至少在我们实验,这个方法还是得到了很大的应用的;
二代测序,Next-generation Sequencing,又被成为高通量测序,现在主要针对二代测序的平台主要是illumina测序平台,lifetech(现在被thermo收购了)的ion torrent平台,还有之前的Roche 454平台,这些平台都各有各的优势,454的读长长,illumina通量高,时间快,ion torrent成本较低,但是这三个平台都没有客服二代测序最深刻的短板——测序成本相当之高。这里说的成本高不只是花钱花的多,而且后期的测序数据出来个10个G都是少的,处理这些数据需要极强的生物信息学的背景,这方面的人才的成本真心的高;另外我见过几个实验室买过二代测序仪,主要就是Hiseq和ion torrent,但是真正用起来的实验室真心的少,大部分还是在那贡着吧!!所以说题主说的精度和速度只是其中的一个原因(这两个就先不吐槽了),想做二代测序,你先成就一个生物信息学的博士再说吧,否则免谈。。。要不就乖乖的送公司做——成本在10-20W之间!!
三代测序没怎么接触过,不过听说名字叫做单分子测序,先膜拜一下!!!
个人觉得,二代测序,难度主要在测序文库的构建和数据分析。
测序文库的构建,很耗人力。如果是重头测序,需要构建不同级别的文库。虽然有标准的实验步骤,不同人构建的文库质量还是有差异的。
数据分析中,有两个难题:重复序列和杂合性。
构建不同级别的文库,就是为了解决重复序列的问题。
生物大多数是多倍体,人是二倍体。测序组装的时候,我们假设生物不同染色体组差异不大,但是有些生物倍性复杂,给组装带来难度。
不知道题主测序精度指的是什么?
测序仪读取序列的精度还是序列组装的质量?
测序仪的精度已经很高了。序列组装需要一定的覆盖度。
前面说的准确度什么的99%我感觉都是说测序仪对碱基的识别,但是在后续信息分析阶段,有一些比对算法、变异识别软件等的问题,会导致最终测序得到的结果准确率下降很多,fusion,cnv什么的先不说,一些复杂的indel准确性会较低。还有有的区域二代测序就是测不准。所以种种原因导致最终的检测结果准确性降低。
测序序列短,不能拼接。第三代单分子测序仪移动速度快,错误多
作者:EDISF
链接:https://www.zhihu.com/question/20479004/answer/428125437
来源:知乎
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。
如果说要有什么新的测序方法,最近看到一篇文章就过来水一下。
这是一篇有关用量子隨穿效应做核酸序列解码的文章,总结起来就是简单,经济和高效,原理大概就是使序列通过一个窄缝,然后通过隨穿效应中能量对距离变化产生的不同信号组来辨别不同的碱基序列。(这大概是最简单的解释了)

量子力学的知识本人是渣渣怕是解释不清楚,各位大神有兴趣可以去扒扒原文。










 当前基因测序技术面临的主要挑战包括测序长度、组装精度、成本控制及数据分析难度。随着IT技术进步,预期这些问题将逐步解决。
当前基因测序技术面临的主要挑战包括测序长度、组装精度、成本控制及数据分析难度。随着IT技术进步,预期这些问题将逐步解决。


















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










