
github:地址
Rdkit|分子片段、R集团分解、片段指纹与指纹重要性分析
分子片段生成
分子片段(molecular fragments)是一组可能具有相关功能的相连的原子组成的。RDKit中包含了大量把分子分解成片段的方法和处理片段的工具。
- 获取官能团库:
RDConfig.RDDataDir目录下的FunctionalGroups.txt - 根据官能团库实例化一个参数器:
FragmentCatalog.FragCatParams()
import os
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem import RDConfig
from rdkit.Chem import FragmentCatalog

- lLen: 官能团最短
- uLen: 官能团最长
- fgroupFilename: 官能团路径
# fName = os.path.join(RDConfig.RDDataDir, 'FunctionalGroups.txt')
fName = "./data/FunctionalGroups.txt"
fparams = FragmentCatalog.FragCatParams(1, 6, fName)
- 查看库中包含的官能团数量:
GetNumFuncGroups() - 查看某个官能团对应的基团:
GetFuncGroup()
print(fparams.GetNumFuncGroups())
m = fparams.GetFuncGroup(2)
Draw.MolToImage(m)

- 传入参数器,创建一个片段存储器:
FragmentCatalog.FragCatalog()
产生的分子片段都会存储在该对象中 - 创建一个片段生成器:
FragmentCatalog.FragCatGenerator()
通过该对象生成片段 - 计算分子片段:
AddFragsFromMol() - 查看分子片段数量:
GetNumEntries()
m = Chem.MolFromSmiles('OCC=CC(=O)O')
fcat = FragmentCatalog.FragCatalog(fparams)
fcgen = FragmentCatalog.FragCatGenerator()
fcgen.AddFragsFromMol(m, fcat)
fcat.GetNumEntries()
通过存储器查看片段:fcat.GetEntryDescription()
尖括号中的内容:表示与片段相连的官能团,以下面的结果为例:
第0号片段中,对应着一个乙基片段,该乙基与一个羟基相连。
第1号片段中,对应着一个乙烯片段,该乙烯与一个羧基相连。
Draw.MolToImage(m)

print(fcat.GetEntryDescription(0))
print(fcat.GetEntryDescription(1))
关于官能团的详细信息,可以通过下述方法获取:
- 向存储器传入分子片段id,获取片段中所包含的官能团编号:
fcat.GetEntryFuncGroupIds() - 向参数器传入官能团编号,获取官能团对应的mol对象:
fparams.GetFuncGroup()
print(list(fcat.GetEntryFuncGroupIds(0)))

funcgroup = fparams.GetFuncGroup(34)
print(Chem.MolToSmarts(funcgroup))
print(funcgroup.GetProp('_Name'))

提取得到的片段是层级结构,小片段在最底层,逐渐合并形成大片段。可以查看一个小片段形成了哪些大片段。
- 根据id获取片段:
fcat.GetEntryDescription() - 获取上级片段id:
fcat.GetEntryDownIds()
print(fcat.GetEntryDescription(0))
list(fcat.GetEntryDownIds(0))
根据上级片段id,获取上级片段信息
fcat.GetEntryDescription(2)
片段指纹生成
先将多个分子的片段汇总到一个片段存储器中
ms = [Chem.MolFromSmiles('OCC(NC1CC1)CCC'), Chem.MolFromSmiles('OCC=CC(=O)O')]
fcat = FragmentCatalog.FragCatalog(fparams)
for m in ms:
fcgen.AddFragsFromMol(m, fcat)
print(fcat.GetNumEntries())

存储器收集完所有片段后,再用它来生成分子指纹
- 创建一个片段指纹生成器:
FragFPGenerator() - 传入分子和存储器来生成指纹:
GetFPForMol(mol, fcat) - 以字符串形式查看指纹:
ToBitString() - 查看指纹中哪些位是有效的:
GetOnBits()
fpgen = FragmentCatalog.FragFPGenerator()
fp1 = fpgen.GetFPForMol(ms[1], fcat)
print(fp1.ToBitString())
print(list(fp1.GetOnBits()))

可以用处理一般分子指纹的方法来处理片段分子指纹,例如寻找相同的片段
- 先对分子指纹做一步“&”位运算,两个指纹都为1时,结果为1,否则为0
- 获取两个指纹中都出现的片段:
GetOnBits() - 查看片段信息:
GetEntryDescription()
fp0 = fpgen.GetFPForMol(ms[0], fcat)
andfp = fp0 & fp1
onbit = list(andfp.GetOnBits())
print(onbit)
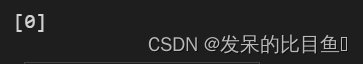
fcat.GetEntryDescription(onbit[0])

也可以按上述思路查看一下哪些片段导致了分子的不同
- 对分子指纹做“^”运算,两个指纹相同时,结果为0,否则为1。再做一步“&”运算
- 按上述方法查看相异片段
dis = fp0 ^ fp1
combinedfp = fp0 & dis
onbit = list(combinedfp.GetOnBits())
print(onbit)
fcat.GetEntryDescription(onbit[-1])

指纹重要性分析
这里主要介绍指纹对离散标签的重要性分析。在rdkit.ML.InfoTheory.rdInfoTheory.InfoBitRanker中提供了对指纹分析的功能。这个类可以根据分子指纹和离散标签,对特征进行计算和排序,看看哪些特征对活性比较重要。
- 先对163个分子生成片段指纹,完整代码如下
sdf_file = './data/bzr.sdf'
suppl = Chem.SDMolSupplier(sdf_file)
sdms = [x for x in suppl if x is not None]
print(len(sdms))
# 获取官能团库
fName = './data/FunctionalGroups.txt'
# 片段参数器
fparams = FragmentCatalog.FragCatParams(1, 6, fName)
# 片段存储器
fcat = FragmentCatalog.FragCatalog(fparams)
# 片段生成器
fcgen = FragmentCatalog.FragCatGenerator()
# 片段指纹生成器
fpgen = FragmentCatalog.FragFPGenerator()
# 汇总所有片段
for m in sdms:
fcgen.AddFragsFromMol(m, fcat)
for i in range(fcat.GetNumEntries()):
print(fcat.GetEntryDescription(i))
fps = [fpgen.GetFPForMol(x, fcat) for x in sdms]
print(len(fps), fps[0].GetNumBits())

dir(fps[0])

fps[0].ToBitString()

信息增益(infoGain)分析,实例化一个排序对象:InfoBitRanker(nBits, nClasses, infoType)
nBits:指纹长度nClasses:类别数量,需要和标签满足的关系:0 <= 标签 < 类别数量infoType:度量指标。默认使用rdInfoTheory.InfoType.ENTROPY,即信息增益作为比较标准,它反映了使用某个特征进行分类后,系统混乱程度降低的多少,数值越大表明特征越重要。
from rdkit.ML import InfoTheory
ranker = InfoTheory.InfoBitRanker(len(fps[0]), 2)
- 获取每个分子的活性信息:
GetDoubleProp(‘ACTIVITY’) - 以7作为标准对活性离散化,大于7为1,小于7为0
- 根据指纹和类别进行投票:
AccumulateVotes(fp, act) - 获取前5个重要特征:
GetTopN(5) - 依次输出特征id、信息增益、特征为1类别中的无活性分子数、特征为1类别中的有活性分子数。
acts = [x.GetDoubleProp('ACTIVITY') for x in sdms]
for i,fp in enumerate(fps):
act = int(acts[i]>7)
ranker.AccumulateVotes(fp,act)
top5 = ranker.GetTopN(5)
for id, gain, n0, n1 in top5:
print(int(id), '%.3f'%gain, int(n0), int(n1))

- 加入偏置,以信息增益为例,重新设置infoType
- 设置偏置类别:
SetBiasList()
在这种模式下,一个特征与所设置了偏置类别的相关性要高于所有非偏置类别,例如设置偏置类别为4,某位特征为1对应的标签中,类别为4的数量应该大于其他类别的数量。
ranker = InfoTheory.InfoBitRanker(len(fps[0]), 2, InfoTheory.InfoType.BIASENTROPY)
ranker.SetBiasList((0,))
acts = [x.GetDoubleProp('ACTIVITY') for x in sdms]
for i,fp in enumerate(fps):
act = 0 if acts[i]<7 else 1
ranker.AccumulateVotes(fp, act)
top5 = ranker.GetTopN(5)
for id, gain, n0, n1 in top5:
print(int(id), '%.3f'%gain, int(n0), int(n1))

使用卡方检验(chi squared test),将infoType设置为如下参数,其他相同
ranker = InfoTheory.InfoBitRanker(len(fps[0]), 2, InfoTheory.InfoType.CHISQUARE)
for i,fp in enumerate(fps):
act = int(acts[i]>7)
ranker.AccumulateVotes(fp, act)
top5 = ranker.GetTopN(5)
for id, gain, n0, n1 in top5:
print(int(id), '%.3f'%gain, int(n0), int(n1))























 1120
1120











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








