欢迎关注”生信修炼手册”!
SMART是蛋白结构域的数据库,该数据库最新版本为v8,收录了1300多个蛋白结构域信息,覆盖了来自uniprot, ensembl等多个数据库的蛋白。官网如下
http://smart.embl-heidelberg.de/
该数据库有以下两种模式
normal
genomic
normal模式下包含了所有uniprot, ensembl的蛋白质信息,这些蛋白序列是存在冗余的,genomic模式下只包含了拥有完整蛋白质组的物种的信息。
两种模式可以通过SETUP菜单进行切换,通过颜色可以辨别所处的模式,示意如下

通过右上角的Search SMART按钮,可以检索该数据库,支持以下蛋白名称和domain两种检索方式。
输入uniprot或者ensembl 数据库中的蛋白ID进行检索,示例如下,根据uniprot数据库中的蛋白IDC1S_HUMAN进行检索
http://smart.embl-heidelberg.de/smart/show_motifs.pl?ID=C1S_HUMAN
检索页面包含如下结果
1. domian 结构图

从图中,可以看出,该蛋白质包括以下5种domain
CUB
EGF_CA
CCP
Tryp_SPc
还提供了每个结构域的位置信息

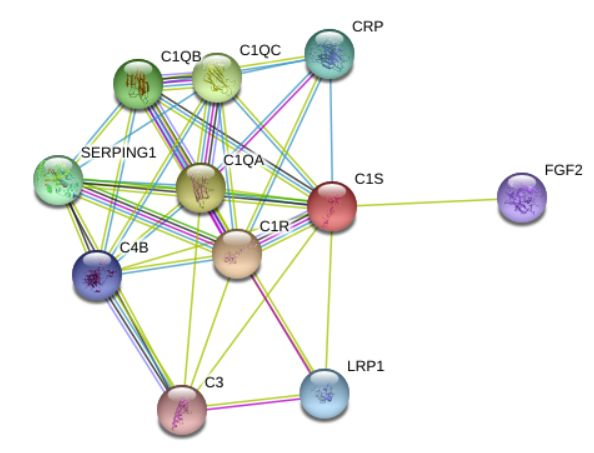
2. 蛋白质相互作用
提供了来自STRING数据库的蛋白相互作用信息,示意如下
3. pathway 信息
提供了Metabolic pathway 和 Kegg pathway 两个数据库的通路注释信息,示意如下

4. orthology group 注释
提供了来自eggNOG数据库的注释信息,示意如下

5. 转录后修饰
提供了来自PTM数据库的转录后修饰信息,示意如下

按照domain进行检索,示例如下,根据domian名称CUB进行检索。对于每个domain, 采用SM开头的编号唯一标识,同时提供了和其他数据库的关联信息,还支持下载多序列比对的结果。

·end·
—如果喜欢,快分享给你的朋友们吧—
扫描关注微信号,更多精彩内容等着你!





 SMART数据库,最新版v8,收录超1300蛋白结构域信息,覆盖uniprot、ensembl等数据库。提供正常及基因组模式,支持蛋白ID和结构域检索,展示结构域图、蛋白相互作用、通路信息、同源群注释及转录后修饰。
SMART数据库,最新版v8,收录超1300蛋白结构域信息,覆盖uniprot、ensembl等数据库。提供正常及基因组模式,支持蛋白ID和结构域检索,展示结构域图、蛋白相互作用、通路信息、同源群注释及转录后修饰。
















 1948
1948

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








