跟着 Cell 学作图 | 5.UMAP降维分析
“实践是检验真理的唯一标准。”
“复现是学习R语言的最好办法。”

DOI: 10.1016/j.cell.2020.05.032
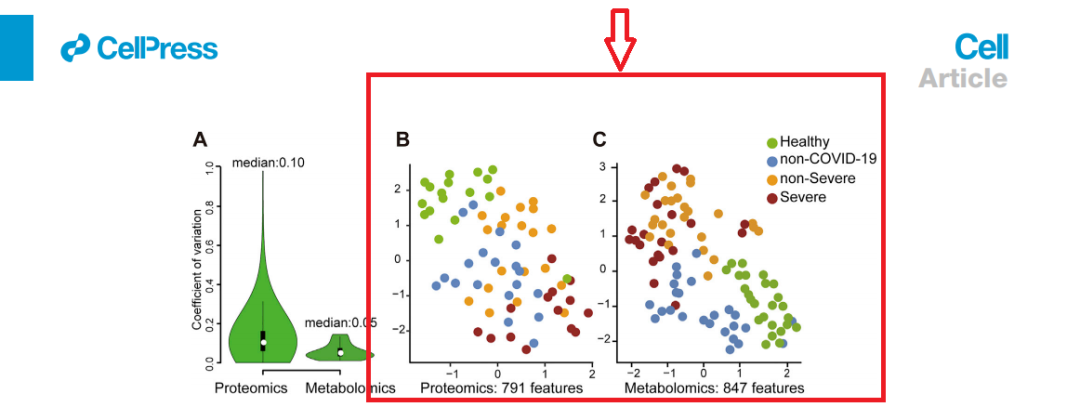
这篇2020年发表在cell上关于新冠的组学文章里面有大量的生信内容。今天带大家复现其中的一个Supplemental Figure:UMAP降维分析图。
本文代码及示例数据领取:后台回复“20210422”
UMAP
UMAP (Uniform Manifold Approximation and Projection) 算法是一种创新的降维流形学习算法。来自于拓扑数据分析。可以用于类似于t-SNE的可视化,但也可以用于一般的非线性降维。文章里的这个图可以看出,sars-cov-2感染患者的血清组学数据与健康个体的组学数据得到了很好的分辨,而其他组则表现出一定程度的分离。
数据格式

注:示例数据仅作展示用,无实际意义!
绘制
很显然,重点在于umap分析,可视化只是常见的散点图。
#------
title: "UMAP"
author: "MZBJ"
date: "2020/4/22"
#-----
# 初次使用需要安装
#install.packages("uamp")
rm(list = ls())
setwd("F:/~/mzbj/cell/2021.4.22")
# 载
library(umap)
library(ggplot2)
#导入数据,首列为行名
df <- read.csv(file = "df_umap.csv",row.names = 1)
#生成一个不含lable列的新数据集
df_umap <- df[,colnames(df)!='label']
#按行进行标准化
df_umap <- data.frame(t(apply(df_umap,1,function(v){(v-mean(v,na.rm=T))/sd(v,na.rm=T)})),stringsAsFactors=F)
#替换NA为0
df_umap[is.na(df_umap)] <- 0
#UMAP分析
umap <- umap(df_umap,method='naive',n_neighbors = 10)
#n_neighbors 用似的局部邻域的大小(根据邻域采样点的数量)。数值越大,流形的全局视图就越多,而数值越小,保留的局部数据就越多。通常值应该在2到100的范围内。
#method 可用的方法是“naive”(用纯R编写的实现)和“umap-learn”(需要python包“umap-learn”)
# 查看降维后的结果
head(umap$layout)
#提取umap值作图用
df1 <- data.frame(umap$layout)
df1$label <- df$label #加入lavle列
colnames(df1) <- c('X','Y','label') #改个列名 不改也行
p <- ggplot(df1, aes(x=X, y=Y, colour=label)) + geom_point(size=4)+
xlab(NULL)+ #不添加x y轴名称
ylab(NULL)
p <- p + theme( panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
legend.title=element_blank(), #图例标签隐藏
panel.border = element_blank(),
axis.line.x = element_line(color="black", size = 0.5),
axis.line.y = element_line(color="black", size = 0.5),
panel.background = element_blank())
#按文章的配色改个颜色
p <- p + scale_colour_manual(values=c("#D58F25","#882424","#7DA92E","#5A7EB3"))
p
#保存为svg格式图片
ggsave(p,filename = "umap.svg",width = 5,height = 4)
出图:

又是一模一样哈哈~
写在后面:
本系列重在复现,所以有些细节可能讲的不是很详细。大家有问题可以后台私信,或者在我的B站:
木舟笔记进行互动!制作不易,希望大家多多支持!
参考
https://github.com/lmcinnes/umap
https://cran.r-project.org/web/packages/umap/vignettes/umap.html
往期内容:
跟着Cell学作图 | 2.柱状图+误差棒+散点+差异显著性检验























 637
637

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








