R 实战 | 使用clusterProfiler进行多组基因富集分析
clusterProfiler这个包我就不再介绍了,网上关于用这个包做的基础的富集分析的教程已经非常多了,今天主要介绍一下使用compareCluster功能进行多组基因的富集分析。
示例数据
富集分析
多个基因集的富集分析
多个分组的富集分析
基本用法
参数
References
往期
示例数据
library(clusterProfiler)
data(gcSample) #载入
str(gcSample) #数据集格式
lapply(gcSample, head) #查看数据集> str(gcSample)
List of 8
$ X1: chr [1:216] "4597" "7111" "5266" "2175" ...
$ X2: chr [1:805] "23450" "5160" "7126" "26118" ...
$ X3: chr [1:392] "894" "7057" "22906" "3339" ...
$ X4: chr [1:838] "5573" "7453" "5245" "23450" ...
$ X5: chr [1:929] "5982" "7318" "6352" "2101" ...
$ X6: chr [1:585] "5337" "9295" "4035" "811" ...
$ X7: chr [1:582] "2621" "2665" "5690" "3608" ...
$ X8: chr [1:237] "2665" "4735" "1327" "3192" ...
> lapply(gcSample, head)
$X1
[1] "4597" "7111" "5266" "2175" "755" "23046"
$X2
[1] "23450" "5160" "7126" "26118" "8452" "3675"
$X3
[1] "894" "7057" "22906" "3339" "10449" "6566"
$X4
[1] "5573" "7453" "5245" "23450" "6500" "4926"
$X5
[1] "5982" "7318" "6352" "2101" "8882" "7803"
$X6
[1] "5337" "9295" "4035" "811" "23365" "4629"
$X7
[1] "2621" "2665" "5690" "3608" "3550" "533"
$X8
[1] "2665" "4735" "1327" "3192" "5573" "9528"富集分析
多个基因集的富集分析
xx <- compareCluster(gcSample, fun="enrichKEGG",
organism="hsa", pvalueCutoff=0.05)
table(xx@compareClusterResult$Cluster) #每个基因集富集个数
head(as.data.frame(xx)) #查看完整结果> table(xx@compareClusterResult$Cluster)
X1 X2 X3 X4 X5 X6 X7 X8
0 3 3 18 10 1 15 19
> head(as.data.frame(xx))
Cluster ID Description GeneRatio
1 X2 hsa04110 Cell cycle 18/384
2 X2 hsa05169 Epstein-Barr virus infection 23/384
3 X2 hsa05340 Primary immunodeficiency 8/384
4 X3 hsa04512 ECM-receptor interaction 9/187
5 X3 hsa04060 Cytokine-cytokine receptor interaction 17/187
6 X3 hsa04151 PI3K-Akt signaling pathway 19/187
BgRatio pvalue p.adjust qvalue
1 126/8105 2.441211e-05 0.007470105 0.006989572
2 202/8105 7.911793e-05 0.012105043 0.011326356
3 38/8105 3.297441e-04 0.033633898 0.031470314
4 88/8105 1.815667e-04 0.045098637 0.042158192
5 295/8105 4.490651e-04 0.045098637 0.042158192
6 354/8105 5.048355e-04 0.045098637 0.042158192
geneID
1 991/1869/890/1871/701/990/10926/9088/8317/9700/9134/1029/2810/699/11200/23594/8555/4173
2 4067/3383/7128/1869/890/1871/578/864/637/9641/6891/355/9134/5971/916/956/6850/7187/3551/919/4734/958/6772
3 100/6891/3932/973/916/925/958/64421
4 7057/3339/1299/3695/1101/3679/3910/3696/3693
5 2919/4982/3977/6375/8200/608/8792/3568/2057/1438/8718/655/652/10220/50615/51561/7042
6 894/7057/6794/2247/1299/3695/2252/2066/1101/8817/1021/5105/3679/3082/2057/3910/3551/3696/3693
Count
1 18
2 23
3 8
4 9
5 17
6 19dotplot(xx) #气泡图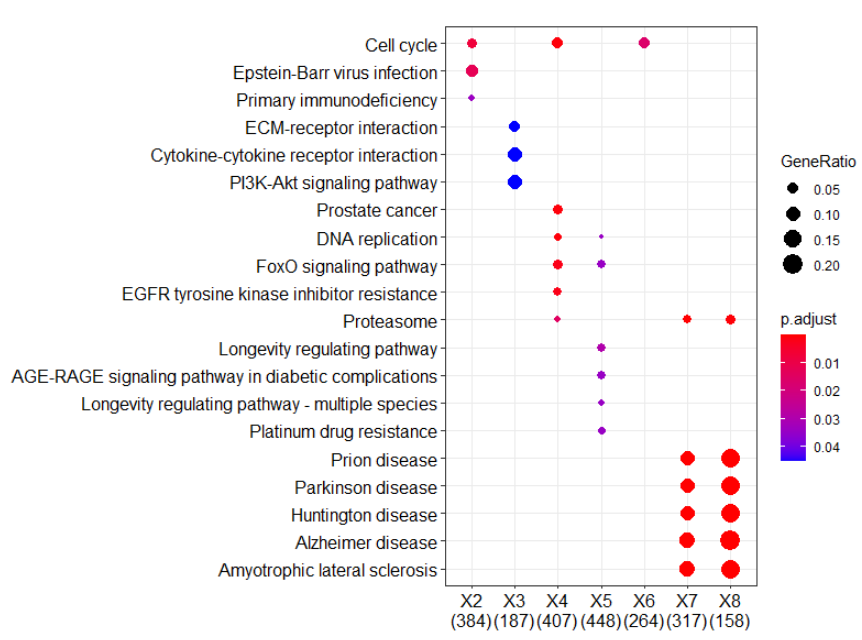
多个分组的富集分析
示例数据
data(geneList, package="DOSE") #载入DOSE包中的数据
head(geneList) #查看数据 包含基因名及其foldchange
mydf <- data.frame(Entrez=names(geneList), FC=geneList)
# 以下内容目的是构建分组 也可以用别的分组
# 将FC大于1的标注为上调 反之为下调
mydf <- mydf[abs(mydf$FC) > 1,]
mydf$group <- "upregulated"
mydf$group[mydf$FC < 0] <- "downregulated"
# 将FC绝对值大于2 的标注为B 反之为A
mydf$othergroup <- "A"
mydf$othergroup[abs(mydf$FC) > 2] <- "B"
head(mydf) # 查看示例数据(两个分组)> head(mydf)
Entrez FC group othergroup
4312 4312 4.572613 upregulated B
8318 8318 4.514594 upregulated B
10874 10874 4.418218 upregulated B
55143 55143 4.144075 upregulated B
55388 55388 3.876258 upregulated B
991 991 3.677857 upregulated B分析及其可视化
# 可以进行单组或多组分析,在 + 号后添加即可
formula_res <- compareCluster(Entrez~group+othergroup,
data=mydf,
fun='enrichKEGG')
dotplot(formula_res)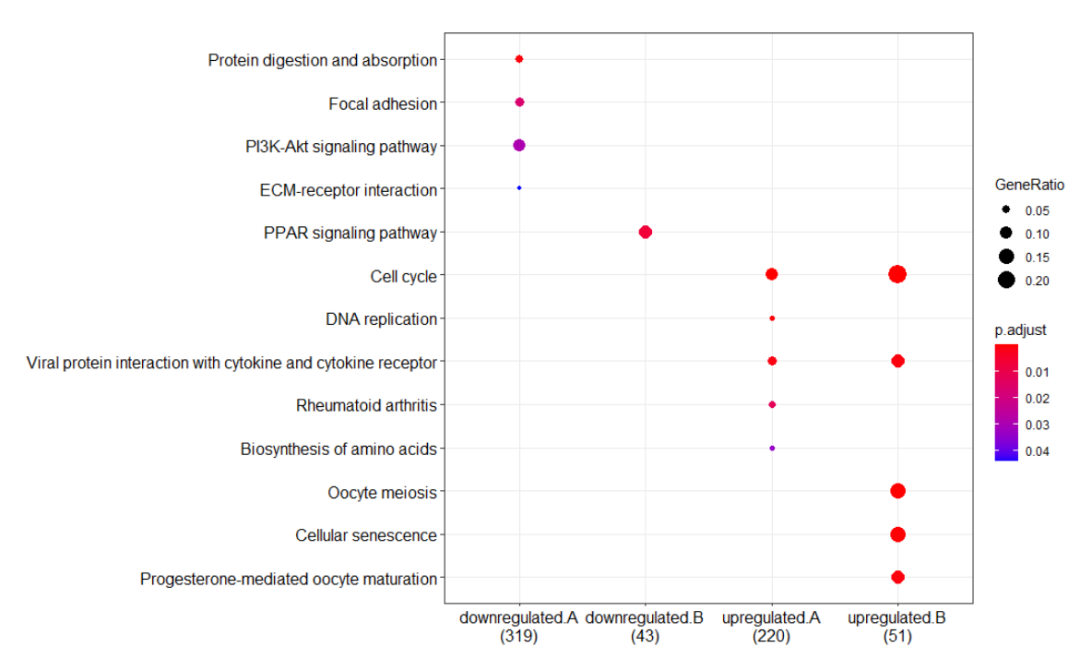
# 同样可以进行分面操作
dotplot(formula_res, x=~group) +
ggplot2::facet_grid(~othergroup) #对于只用一次该包的功能可以这么写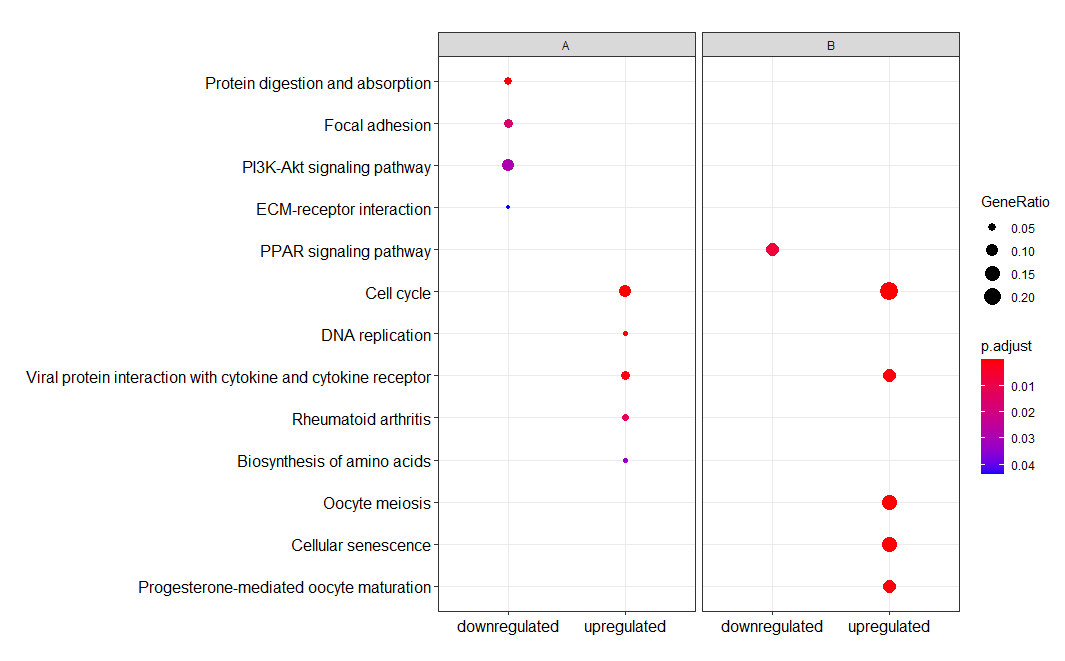
基本用法
最后附上用法参数。
compareCluster(geneClusters, fun = "enrichGO", data = "", ...)参数
geneClusters | entrez基因list. 或者, Entrez~分组 形式的列表 |
|---|---|
fun | "groupGO", "enrichGO", "enrichKEGG", "enrichDO" or "enrichPathway" . |
data | 数据集 |
References
Chapter 11 Biological theme comparison | clusterProfiler: universal enrichment tool for functional and comparative study (hiplot.com.cn)
往期






















 868
868











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








