本节用到的数据来源于25个欧洲国家的蛋白质摄入百分比,获取数据的链接是http://www.biz.uiowa.edu/faculty/jledolter/DataMining/protein.csv
#数据集
library(readr)
food <- read_csv("D:/a_DUFE/000master_gogogo/R/深入浅出R语言数据分析/protein.csv")
head(food,3)

数据集包括25条数据、10个特征。
K均值聚类
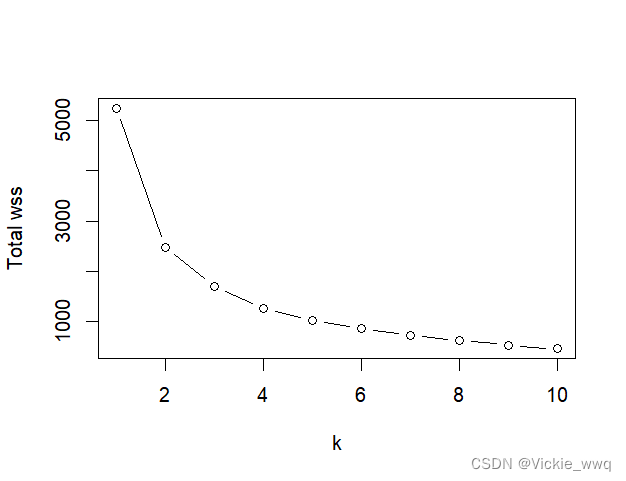
#确定聚类数目:Elbow方法
kmax <- 10
Elbow <- sapply(1:kmax, function(k) kmeans(food[,-1],centers=k,nstart=10)$tot.withinss)
plot(1:kmax,Elbow,type='b',xlab='k',ylab='Total wss')
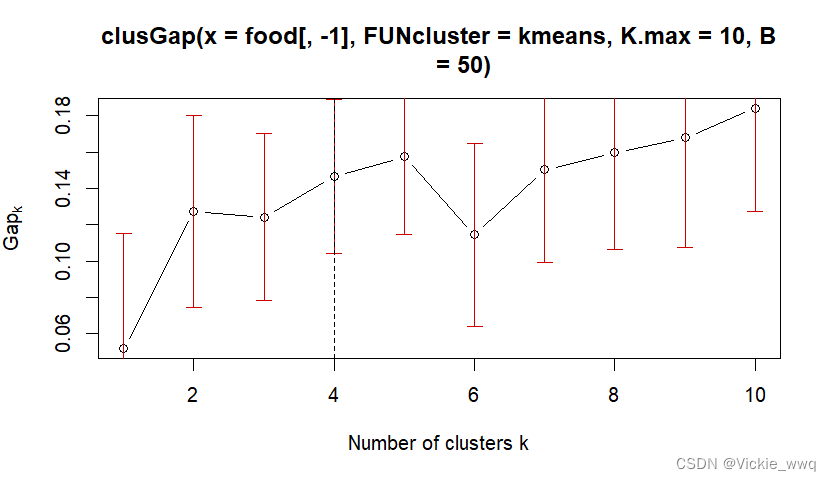
#确定聚类数目:Gap统计方法
library(cluster)
set.seed(123)
gap_stat <- clusGap(food[,-1],FUN=kmeans,K.max = 10,B = 50)
plot(gap_stat,xlab = "Number of clusters k")
abline(v=4,lty=2)
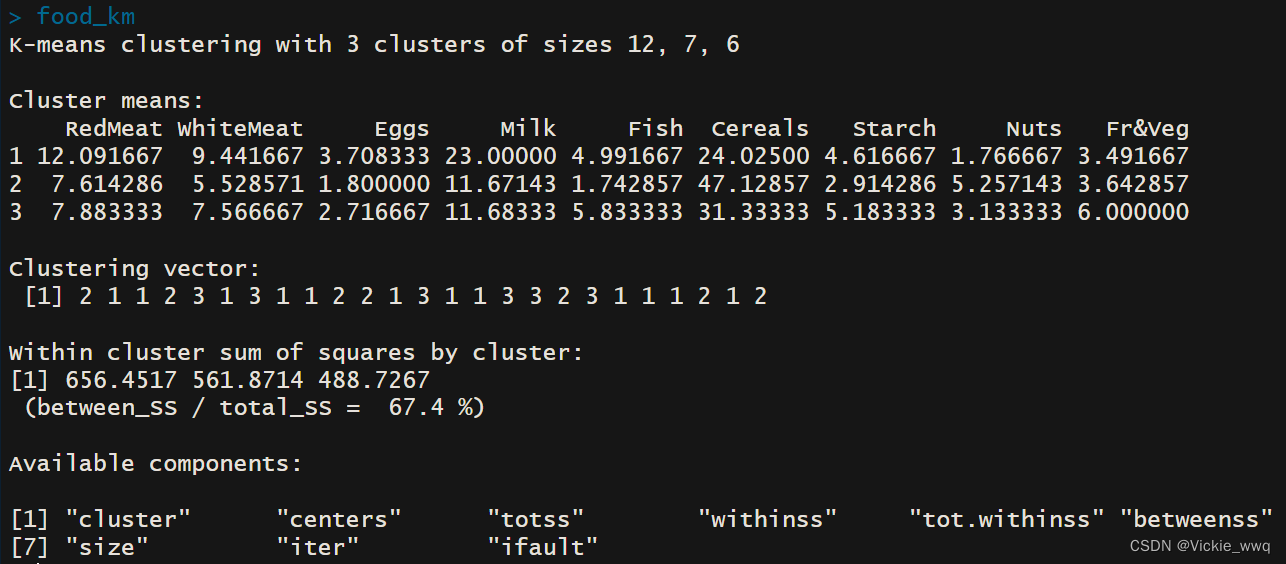
#构建聚类模型
set.seed(123)
food_km <- kmeans(food[,-1],centers = 3,nstart = 50)
food_km
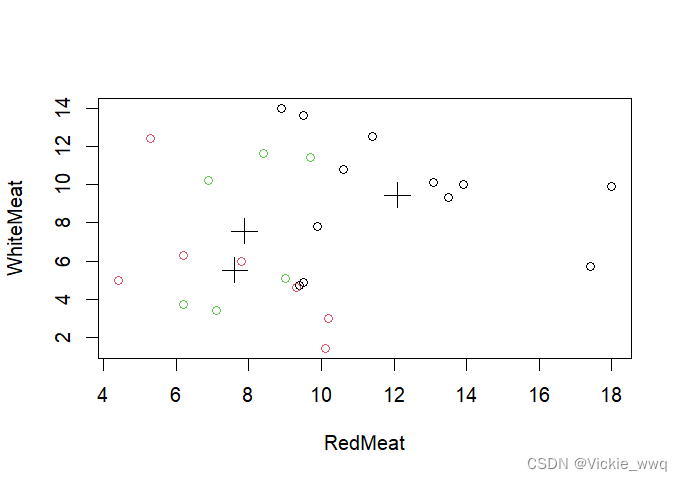
#结果可视化
plot(data.frame(food[,2], food[,3]), col=food_km$cluster)
points(food_km$centers,pch=3,cex=2)
clusplot(food[,-1], food_km$cluster, color = T, labels = 2, main = 'Cluster Plot')
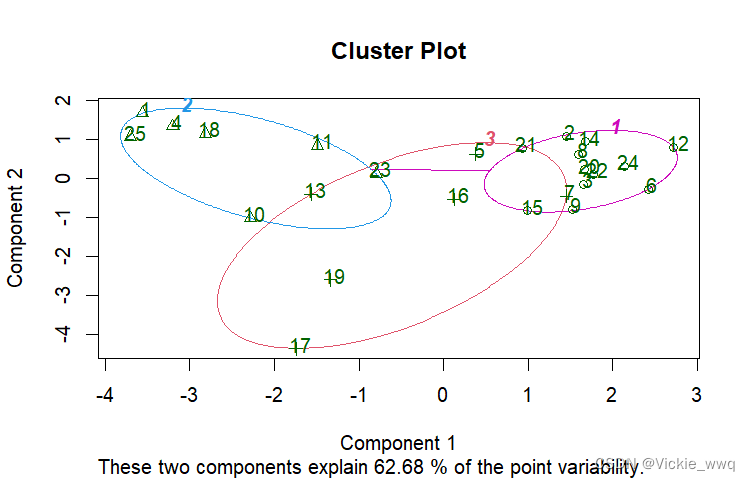
层次聚类
#计算距离
dist_data <- dist(food[,-1],method = "euclidean")
#进行层次聚类
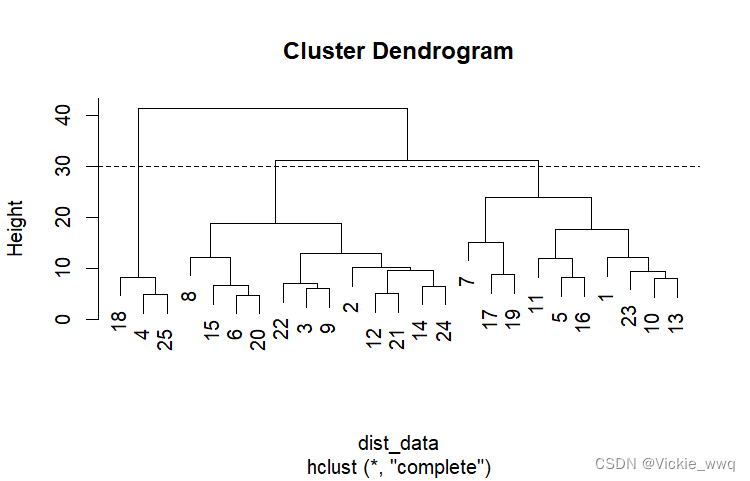
hdata <- hclust(dist_data)
hdata
# Call:
# hclust(d = dist_data)
#
# Cluster method : complete
# Distance : euclidean
# Number of objects: 25
plot(hdata)
abline(h=30,lty=2)
Medoids聚类(PAM)
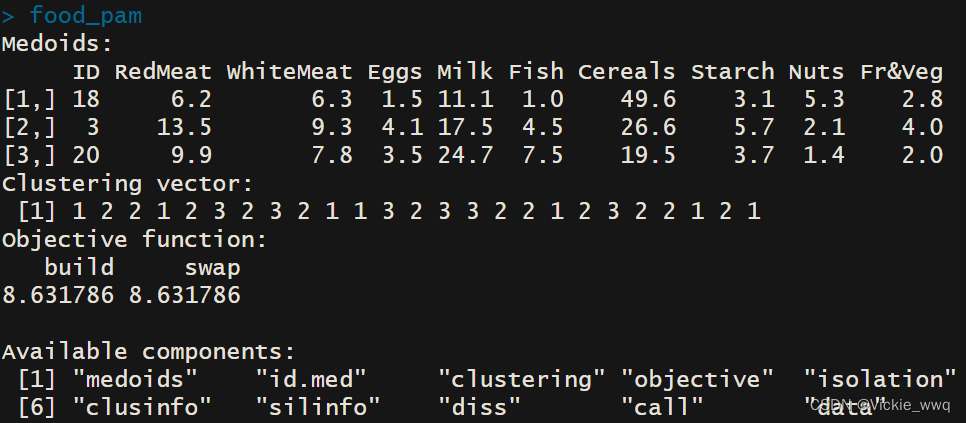
food_pam <- pam(food[,-1], 3)
food_pam
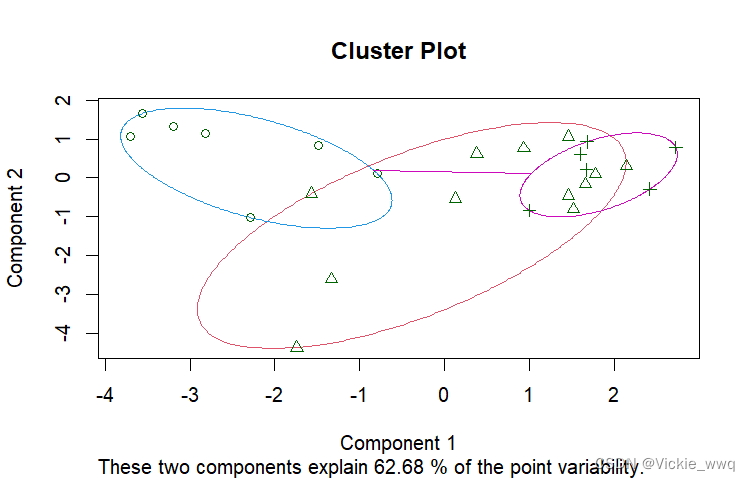
clusplot(food[,-1], food_pam$clustering, color = T, main = 'Cluster Plot')
clusplot(food[,-1], food_pam$clustering, color = T, main = 'Cluster Plot')






















 3021
3021











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








