作者,Evil Genius
关于OncoKB数据库
OncoKB(Precision Oncology Knowledge Base)网址:https://www.oncokb.org/,该数据库由Memorial Sloan Kettering Cancer Center开发并维护,以肿瘤患者基因突变为核心,收录突变对应的靶向药物使用、生物学与肿瘤学效应,以及突变在人群中分布频率和临床预后特征等信息。OncoKB数据来源包括FDA、NCCN、ASCO或ESMO会议论文、肿瘤领域专家共识和论文文献,以及cBioPortal,COSMI等公共数据库,共56种肿瘤相关的682个基因5425种改变以及对应的96种药物注释信息,每条信息经过临床基因组学注释委员会(Clinical Genomics Annotation Committee, CGAC)的定期审阅与修订。

2021年10月7日,OncoKB数据库获得了美国FDA的部分认可,它是第一个被FDA部分认可的癌症体细胞变异数据库(体系变异是生命后期发生的改变,而不是出生时遗传的胚系变异)。
FDA认可OncoKB数据库中一部分为2级(临床意义,46个基因)和3级(潜在临床意义,37个基因)共83个基因生物标志物(注意,FDA 2级和3级的基因有重叠部分,整合后为66个基因)的有效科学证据来源,可用于简化肿瘤NGS分析测试的开发和验证过程。以后肿瘤基因检测公司可以使用这些数据来支持上市前提交的肿瘤分析测试的临床有效性。使用DNA测序确定肿瘤的突变谱进而指导靶向治疗和研究性治疗方案。
由于OncoKB不包含针对特定治疗产品的任何CDx声明,因此根据定义,OncoKB中的任何基因变异都不会被视为FDA 1级。

▲ FDA认可的证据级别,其中1级为CDx;2级为具有临床意义的突变:使医疗保健专业人员根据临床证据(如专业指南中提出的临床证据)使用有关患者肿瘤的信息。这类信息得到了分析有效性(适当情况下针对突变本身或通过代表性的方法)和临床有效性(通常基于公开的临床证据,如专业指南和/或同行评审的出版物)的证据支持;3级为具有潜在临床意义的突变:不被认为是1级或2级生物标志物的突变可以被描述为具有潜在临床意义的癌症突变。这些突变可能可以提供有用的信息,或用于指导患者参加可能适合于他们的临床试验。此类信息得到了分析有效性(适当情况下通过代表性方法)和纳入专家组临床或基础机制(通常包括同行评审的出版物或体外临床前研究模型)的证据支持。
▲ OncoKB证据级别与FDA证据级别之间的映射:由于OncoKB不包括诊断公司要求处方针对特定治疗产品的任何CDx声明,因此根据定义,OncoKB中的任何基因变异都不会被视为FDA 1级;OncoKB的1类和2类证据,以及标准耐药性变异R1证据均映射到FDA 2级;OncoKB的2类新兴生物标志物(NCCN中的2A类)和3A,3B和4类证据,以及临床试验支持的耐药性变异R2证据均映射到FDA 3级。
更为直观的分类如下:FDA药物标签和部分NCCN指南为FDA 2级(临床意义),部分NCCN指南和同行评审的出版物以及OncoKB评估的基于生物标志物的研究或分析类型为FDA 3级(潜在临床意义)。

OncoKB数据库以基因为核心,提供了每个基因的变异、相关肿瘤类型,实验证据等信息,示意如下:
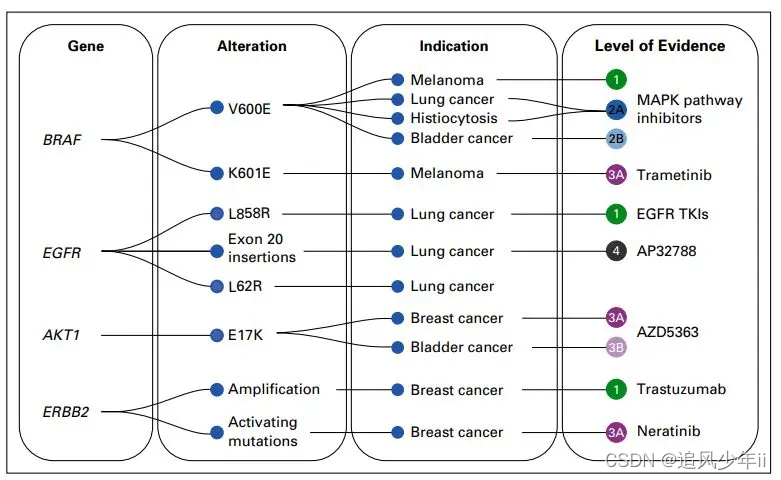
进入OncoKB官网主页,可以看到OncoKB根据不同证据水平将肿瘤中基因突变信息分为四个数据等级,其中:
1级数据为,被FDA推荐的,可以作为FDA批准的某药物治疗肿瘤疗效biomarkers的突变;
2级数据是,NCCN或其他专家共识的,在肿瘤标准治疗中可以作为经FDA批准的某药物疗效biomarkers的突变;
3A级数据为,令人信服的临床试验证明的,可以作为某药物治疗肿瘤疗效潜在靶标的突变;
3B级别数据为,经调查表明某FDA批准或未批准药物在肿瘤标准治疗中可以作为药物疗效biomarkers,但尚未被专家共识认可的突变;
4级数据为,高质量细胞或动物实验文献研究报道的,作为某药物治疗肿瘤疗效潜在靶标的突变。
另外有R1/R2级数据,为肿瘤耐药相关突变信息,R1为肿瘤标准治疗中可以作为FDA批准的药物耐药指标的突变信息,R2为某药物治疗肿瘤产生耐药的相关突变信息临床研究证据。
点击Levels of Evidence可见如下图所示的数据等级分级。

OncoKB数据库核心功能及操作演示
点击Cancer Genes可预览该数据库收录的全部肿瘤相关的基因突变信息:
第一列:基因名称
①列打“√”的基因表示被OncoKB收录有注释信息,标“信封”的表示可点击信封标志申请添加注释信息;
②列为肿瘤学功能,TSG为抑癌基因,Ocongene为癌基因;
③列为注释信息来源;
④列为数据来源的总数。各列标题右下角的小箭头均可点击进行排序,表格右上角提供检索功能。

Actionable Genes为OncoKB核心功能模块,点击进入功能页面,可以看到依次有Level 1~4和Level R1/R2级别的数据,提供以肿瘤类型、药物和突变基因名三种方式进行检索。
除了查询功能外,该数据库还提供了命令行工具,用于注释自己的基因组变异信息,支持SNV, CNV, 融合基因等信息的注释,该工具名为oncokb-annotator, 网址如下:
但是对于我们生信而言,不可能一个位点一个位点的查询,需要把数据库的所有信息全部抓取出来,以供临床诊断。
抓取数据库的时候需要我们提供基因列表,我们可以全部抓取,也可以按照我们自己分析的panel 基因进行抓取,准备一个excel表格就可以了,示例如下:

然后我们用python脚本进行数据抓取,全自动化脚本,
import os
import sys
import pandas as pd
import pandas
import requests
import json
from pandas import ExcelWriter
from openpyxl.utils import get_column_letter
project_path = '/share/work/zhangjianli/work/DATABASE/OncoKB/'
os.chdir(project_path)
def to_excel_auto_column_weight(df: pd.DataFrame, writer: ExcelWriter, sheet_name):
"""DataFrame保存为excel并自动设置列宽"""
df.to_excel(writer, sheet_name=sheet_name, index=False)
# 计算表头的字符宽度
column_widths = (
df.columns.to_series().apply(lambda x: len(x.encode('gbk'))).values
)
# 计算每列的最大字符宽度
#max_widths = (df.astype(str).applymap(lambda x: len(x.encode('gbk'))).agg(max).values)
# 计算整体最大宽度
# 设置列宽
worksheet = writer.sheets[sheet_name]
for i, width in enumerate(column_widths,start=1):
# openpyxl引擎设置字符宽度时会缩水0.5左右个字符,所以干脆+2使左右都空出一个字宽。
worksheet.column_dimensions[get_column_letter(i)].width = width + 6
t_on = 1
d_on = 1
p_on = 1
if(os.path.exists(project_path+'temp.xlsx')):
gene_list = pandas.read_excel(project_path+'temp.xlsx')
oncokbdatabase = pandas.read_excel(project_path+'Oncokb_db.xlsx',sheet_name='Therapeutic')
diagdatabase=pandas.read_excel(project_path+'Oncokb_db.xlsx',sheet_name='Diagnostic')
prodatabase=pandas.read_excel(project_path+'Oncokb_db.xlsx',sheet_name='Prognostic')
else:
gene_list = pandas.read_excel(project_path+'gene_685_oncokb.xlsx')
gene_list['is get']='F'
oncokbdatabase = pandas.DataFrame(
columns=['Gene', 'Alteration', 'Oncogenic', 'Mutation Effect', 'Citations', 'Level', 'Alterations',
'Level-associated cancer types', 'Tumor-MainType', 'Drugs', 'Tissue','Tumor-Form','Citations-2'])
prodatabase = pandas.DataFrame(
columns=['Gene', 'Alteration', 'Oncogenic', 'Mutation Effect', 'Citations', 'Level', 'Alterations',
'Level-associated cancer types', 'Tumor-MainType', 'Tissue','Tumor-Form','Citations-2'])
diagdatabase = pandas.DataFrame(
columns=['Gene', 'Alteration', 'Oncogenic', 'Mutation Effect', 'Citations', 'Level', 'Alterations',
'Level-associated cancer types', 'Tumor-MainType', 'Tissue','Tumor-Form','Citations-2'])
gene=[]
for g in range(len(gene_list)):
if(gene_list['is get'].iloc[g]=='F'):gene.append(gene_list['gene'].iloc[g])
#要search的gene列表已经在gene中
headers ={'Authorization': 'Bearer 84a75c6f-7daf-4fee-86ce-ae3f12f31413'}
count = 0
for index,g in enumerate(gene):
count = count+1
gene_url 







 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 2064
2064

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








