目录
0 参考文献
SLAT原论文:Spatial-linked alignment tool (SLAT) for aligning heterogenous slices
1 方法特点
空间组学技术的进步揭示了生物组织器官以及生物体本身结构的空间排布。尽管由于受到技术显示,空间组学技术相比单细胞组学技术降低了一些分辨率(空间组学每一个检测的spot包含多个细胞,相当于检测了这些细胞的综合值)。但是空间组学技术允许人们在关注细胞分子参数的同时,获得空间位置信息,对于帮助人们理解生命活动的调控原理和微环境对细胞的影响等方面具有更大的作用。
现在各种组学(转录组、表观组、蛋白质组等)的技术都已经实现了对于切片的分子参数进行原位测量。因此寻找一种方法合理地将多个切片对齐是至关重要的,这可以增强下游任务的表现。比如将处于不同发育阶段的胚胎切片进行对齐,可以帮助确定在发育过程中,关键的时空变化以及其分子基础。
然而目前大多数空间组学数据对齐方法,都是针对同质切片进行对齐,比如对同时期测量的同器官、同组学的多个相邻切片进行对齐,重建组织的3D结构。无法用于对齐异质切片,因为异质切片,通常会具有明显的非刚性形变、不同的空间分辨率以及复杂的批次效应。
这篇文章提出一种可以用于异质切片对齐的方法SLAT(Spatially-Linked Alignment Tool),当然,这种方法也可以进行同质切片对齐。
值得一提的是,这是首个发表的异质切片对齐方法。

2 算法细节
2.1 数据建模
空间位置信息和组学特征的联合建模是每一个空间切片对齐算法都需要进行的步骤。
在SLAT中,将数据集表示为,其中N是spot(或cell)的数量,
代表第i个spot原始的组学特征的向量(G为组学特征的数量,例如在空间转录组数据中就是所测量的基因的种类数量),
代表第i个spot的空间位置坐标。
将每个spot的特征和坐标组成矩阵为。对于要进行对齐的两组数据,使用
和
表示。
为了能够校正不同样本之间的批次效应,在数据预处理阶段使用基于SVD分解的跨数据集矩阵分解策略。
将进行了标准化后的数据表示为。随后进行如下SVD分解

根据分解得到的矩阵,可以通过如下形式得到经过批次校正的量数据集的嵌入结果(embeddings)

其中,是对应最大的M个特征值的矩阵。
对数据集中的数据构建邻接图
,其中
对应于根据上述分解获得的批次校正后的表示
,边连接节点与其k近邻。这个邻接图可以使用二值矩阵A表示,1对应节点之间有边连接,0则反之。
值得注意的是,在对齐来自不同技术的异质切片时,由于可能不同技术具有不同的空间分辨率,所以可以通过调整k的大小,来针对每个切片构建邻接图。
SLAT将两个数据集根据他们对应的两个空间邻接图,将该问题建模为最小成本二分匹配问题。

其中是分别来自两张图的节点
的嵌入表示,M是一组固定大小的匹配关系。类似地,使用
来表示两个数据集的嵌入表示的集合。
上述问题已经被证明等价于最小化图匹配对应的节点表示的Wasserstein距离。
2.2 整体表征的构建
准确的对齐应该使得对齐的spot具有相同的类型和空间位置情况。空间位置情况包括各种规模的空间情况,例如从微环境到其整体组织中的所在位置。
SLAT首先使用LGCN(lighweight graph-convolutional network,轻量级图卷积网络)来生成整体的细胞表示。

其中,
是
的度矩阵(degree matrix,是一个对角矩阵,其中每个节点的度数(连接边的数量)是对角线上的元素),L是最大步数。
最后得到的整体表征是每一步得到的矩阵的拼接。
根据上面的方法,可以对每一个数据集分别进行操作,从而获得每个数据集的整体表征

2.3 对抗图对齐
根据2.2的方法得到了两个数据集的整体表征之后,SLAT使用对抗对齐的方式学习两个数据集的spot的嵌入表示,使得图匹配的Wasserstein距离最小。
具体而言,使用一个多层感知器来减轻两种整体表征由于组学分部之间的差异或者空间拓扑引起的系统偏差。

引入Wasserstein判别器并构建损失函数,Wasserstein判别器
需要来最大化这个函数。
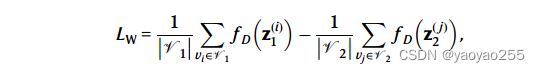
被训练以最小化上述损失函数。
由于不同数据集存在有差异的spot类别分布,或者数据集特有的空间区域,因此不应该假设不同数据集具有相同的分布。

在对抗训练中,使用Wasserstein判别器的输出作为动态剪切标准,从两个数据集选择具有最小 Wasserstein 距离的一部分spot,分别选出个spot,c是一个控制比例的超参数。

这些spot代表着指导对齐的最可靠的anchor。因此损失函数构建如下

这种方式保证了特有的区域不会被强制对齐。
结果表明SLAT在c=0.6时表现最好。
为了防止退化(所有嵌入表示坍缩至同一个值),SLAT采用了一个重建部分来确保嵌入表示保留了足够多的信息以重建输入。具体而言,使用一个多层感知器来实现重建输入,并构建对应的损失函数如下

通过消融实验,他们发现Wasserstein鉴别器在对齐同质切片时效果不明显,在对齐异质切片时明显提高了正确率。
2.4 总体优化目标
综合以上损失函数,得到SLAT的整体优化目标如下

其中α是一个用来权衡损失函数重要程度的超参数。使用随机梯度下降SGD进行优化。
2.5 坐标匹配
除了上面的模型,SLAT还提供了一种可选项,用于使用附加信息来匹配两个切片的spot的空间坐标。坐标匹配的目标是通过估计仿射变换矩阵M来大致对齐不同切片的总体方向,例如用于两个对称的切片如左右脑)。具体策略取决于可用信息的类型。
首先,遵循组织结构的知识,M可以通过结合放缩、旋转和平移来计算。其次,如果有H&E染色图像,可以通过SimpleElastix,一个具有良好效果的医学图像配准工具,来估计M。最后,如果没有其他可用信息,可以直接使用ICP进行进行图像对齐。
有了M矩阵之后,则可以通过一下方式改变其中一个数据集的坐标。
![]()
2.6 spot概率匹配
有了两个数据聚集(切片)的嵌入表示,坐标变换后的坐标
,就可以将两个数据集进行概率匹配。
对于第一个数据集中的第i个spot,现在第二个数据集根据坐标,找到其k近邻作为候选。
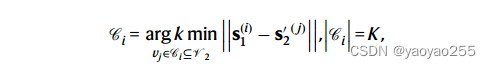
然后根据嵌入表示,计算这k个近邻与该spot的嵌入表示的余弦相似度。将计算得到的余弦相似度与一个由1000对随机抽样的细胞对形成的零分布进行比较。最后得到的匹配结果包含p-value小于0.05的匹配。
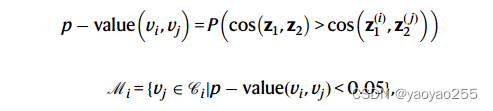
























 1496
1496

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








