「
点击蓝字 / 关注我们
」
近日,中山大学骆观正课题组与上海科技大学季泉江课题组合作,在Cell Reports杂志发表了题为“Targeted genetic screening in bacteria with a Cas12k-guided transposase”的研究论文,开发了名为Site-specific Transposon-Asisted Genome Engineering (STAGE)的新方法,该方法允许在多种微生物(例如铜绿假单胞菌和肺炎克雷伯菌)中进行高通量的Cas12k介导的基因敲除筛选。在这里附上文献详解以馈读者。

微生物基因组的多样性使微生物种群能够快速适应不断变化的环境。为了研究未探索的基因组功能,高通量基因组编辑和筛选是基因型-表型关系功能分析和了解细胞网络复杂性的主流方法。随着高通量的DNA合成技术和高通量测序(NGS)的进步,并行产生准确和特定突变的高通量方法已不是基因组规模分析的瓶颈。目前几种高通量基因工程方法,例如Tn-seq、MAGE、TRMR和CREATE已被开发用于细菌中的快速基因组大规模诱变。Tn-seq已被广泛应用于不同细菌物种中,是构建基因组规模的转座子诱变文库的经典策略。然而,靶向非特异性基因组位点使得该方法无法构建位点特异性诱变文库,通常需要后续大量的筛选来分离所需的突变。相比之下,由同源重组介导的高通量基因工程方法(例如MAGE和TRMR)使得在大肠杆菌中位点特异性的基因组整合成为可能。然而由于其他细菌物种可能缺乏有效的噬菌体重组蛋白,使得这些方法在其他菌种中不像在大肠杆菌中那样有效。最近,两个CRISPR相关转座子系统被报道称可在大肠杆菌中使用gRNA来介导高效和位点特异性基因组转座,而不依赖于同源重组。鉴于其可编程性、高效率和可操作性,该技术是用于位点特异性基因组规模诱变的理想高通量基因工程工具。
铜绿假单胞菌中ShCAST系统的构建
CRISPR-Cas12k引导的转座酶系统是一种由Cas12k、sgRNA、TniQ/TnsBC转座酶构成的定向转座系统。利用Cas12k/gRNA复合物的基因组识别和定位功能, TniQ/TnsBC能够实现蓝藻细菌基因组的定向转座。为了评估该系统是否适用于其他细菌物种,我们设计并构建了一个基于双质粒的ShCAST系统,以鉴定该系统在铜绿假单胞菌中的整合效率。其中pCASTPA质粒可以在L-阿拉伯糖诱导型启动子PBAD的控制下表达TnsB、TnsC、TniQ和Cas12k蛋白,而pGTPA质粒包含gRNA表达盒和转座子供体DNA(图1)。此外,反选择标记sacB也被组装到两个质粒中,在蔗糖存在下sacB会变成致死基因,可用于转座后的质粒筛选。

图1
我们将含有九个不同Spacer的pGTPA质粒分别电穿孔到携带pCASTPA质粒的铜绿假单胞菌PAO1菌株中。培养后通过使用与目标基因组位点相邻的引物和位于转座子供体DNA内的引物进行聚合酶链式反应(PCR)确认,在9个测试位点均发生了成功的转座事件(图2)。证明了双质粒的ShCAST系统可以应用于铜绿假单胞菌。

图2
之后为了仔细评估ShCAST系统在铜绿假单胞菌中的插入频率,我们将eGFP作为转座子供体DNA组装到pGTPA质粒中。在靶向rhlR和lasR基因时,分别在921个总菌落中观察到10个明亮菌落,在657个总菌落中观察到8个明亮的菌落。此外,定量PCR(qPCR)分析显示,9个测试位点的整合效率范围为10-5到10-2,与eGFP转座分析的结果一致(图3)。
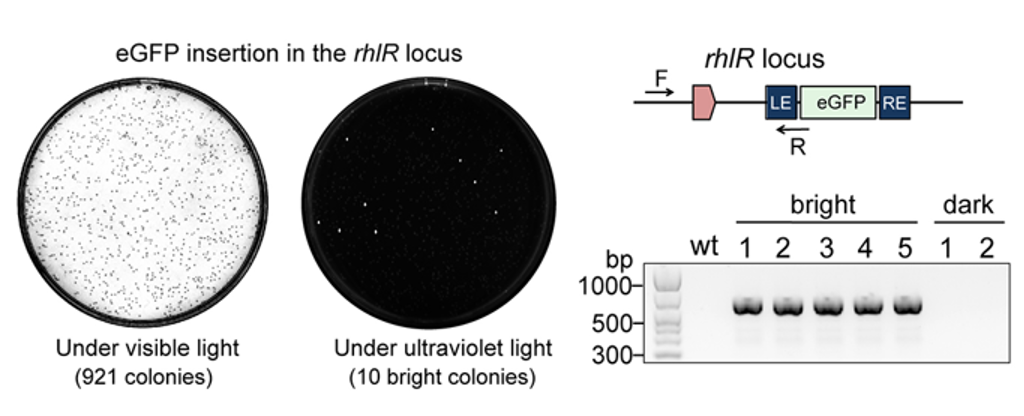
图3
此外为了进一步扩展ShCAST系统在不同细菌中的多功能性,我们将ShCAST系统应用于肺炎克雷伯氏菌,这是一种重要的工业微生物和主要的人类病原体。与铜绿假单胞菌的结果相似,肺炎克雷伯菌基因组中的10个测试位点都成功地被转座子DNA整合,但是整合的频率相对较低。
ShCAST系统在铜绿假单胞菌和肺炎克雷伯氏菌中的低整合效率阻碍了成功转座菌种的分离。因此我们试图使用抗生素选择来富集包含成功插入转座DNA的阳性菌种。为了实现这一目标,我们使用庆大霉素抗性基因作为转座子供体DNA。并在菌种发生编辑后,在含有蔗糖和庆大霉素的Luria-Bertani(LB)板上选择菌落。蔗糖用于去除含有游离质粒或整合到细菌基因组中的质粒的细胞,而庆大霉素用于筛选成功整合因此含有庆大霉素抗性基因的菌种(图4)。
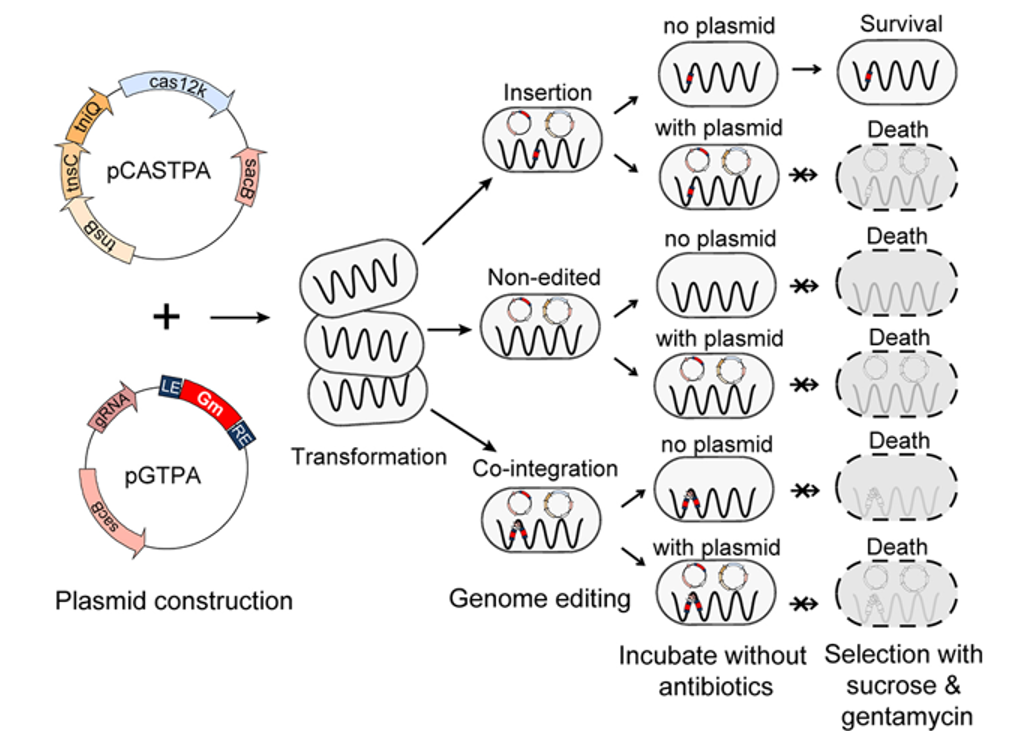
图4
在用庆大霉素和蔗糖选择后,铜绿假单胞菌中的所有测试基因都实现了高效率的富集。此外,随机挑选三个菌落进行纳米孔测序发现转座子供体DNA成功插入目标位点,同时没有检测到脱靶整合或剩余质粒。
STAGE突变文库的设计与构建
鉴于ShCAST系统的位点特异性转座的能力和选择后阳性转座事件的高富集效率,研究试图在铜绿假单胞菌中构建一个集中的TF库,以便对TF功能进行全面分析。我们根据NCBI和已有的基因注释,将铜绿假单胞菌PAO1菌株中的593个带注释的TF确定为靶基因,为每个靶基因设计并选择了15个sgRNA,一共8891个sgRNA靶向593个TF。除此之外我们还设计了100个靶向10个必须基因的sgRNA作为阴性对照。同时在sgRNA的3'和5'端添加与pGTPA的质粒骨架同源的25个核苷酸序列,以促进sgRNA文库的组装(图5)。

图5
在芯片合成sgRNA文库后,我们将sgRNA文库组装到pGTPA质粒中。对质粒的NGS分析显示100%的spacer序列以低偏差存在于质粒文库中。将质粒文库电穿孔到含有pCASTPA的PAO1菌株中生成突变菌种文库。在用蔗糖和庆大霉素筛选后,收集到大约2.6×106个菌落,达到了sgRNA质粒文库~290倍的覆盖率,保证了文库的完整性。
STAGE突变文库的高通量测序分析
为了评估构建的STAGE菌株库的质量,我们通过向每个插入片段引入唯一分子标识符(UMI)来设计定量扩增子测序策略。在对NGS文库进行深度测序后,计算UMI数以表示靶向插入事件的频率(图6)。

图6
首先我们确定了每个插入位点与设计的spacer的最近识别位点之间的距离,大多数插入事件发生在识别位点下游的60-70个碱基对(bp),与大肠杆菌中ShCAST系统的插入模式一致(图7A)。此外,我们还观察到5bp重复插入事件,这是Tn7转座产物的标志性特征。而在整个基因组中插入事件中,发现总reads的约93%位于目标位点附近。对NGS数据的进一步分析显示,92.9%的设计spacer序列下游存在插入事件,且覆盖了98.48%的靶基因,未检测到所有针对10个必需基因(阴性对照)的插入事件。大多数靶基因中均鉴定到了超过10个以上的sgRNA介导的插入事件,这些插入事件发生的频率很高(图7B)。值得注意的是,插入频率在目标靶基因之间差异很大(图7C)。为了评估插入频率是否反映了局部序列特征,我们计算了可能影响gRNA识别的多个因素,包括PAM序列、GC含量、相对于目标基因的相对位置和基因组命中分数。然而,插入频率与这些因素仅微弱相关。环境压力和不同转座子突变体的生长速度也可能会干扰插入频率的鉴定。通过将sgRNA质粒文库的丰度与相应靶基因的插入读数进行比较,我们注意到靶基因内部所有插入事件总体的插入读数分布与原始质粒丰度显著不同,表明STAGE文库的插入频率在培养过程中受到了影响。我们进一步分析了插入频率显著变化的目标基因,揭示了插入频率降低或增加的两个基因类别(图7D)。这一结果表明,这些基因的破坏会严重影响细菌的生长,导致这些突变体的快速生长或死亡。

图7
而在插入频率低的基因中,除了10个必需基因的阴性对照组外,我们在STAGE菌株库中还观察到14个不可编辑基因。其中ptrB、mucA、rsmA和glnK四个基因被Tn-seq识别为必需基因。而之前的研究表明,这四个基因都能够在铜绿假单胞菌中成功删除,因此这些基因的重要性存疑。总之,这些结果表明,ShCAST介导的转座子筛选是一种强大而可靠的必需基因鉴定方法。
STAGE文库抗生素耐药相关基因的鉴定
铜绿假单胞菌是ICU、老年患者和烧创伤患者局部感染和全身感染的常见病原菌。近年来,铜绿假单胞菌的感染率呈现上升趋势,同时耐药性也日益严峻,因此迫切需要阐明耐药机制并开发治疗方法。我们试图使用STAGE文库确定导致铜绿假单胞菌抗生素耐药性的基因和相关途径。我们用三种不同的临床相关抗生素(亚胺培南,头孢他啶和氧氟沙星)处理STAGE突变文库。暴露于亚致死水平的抗生素会导致细菌生长的分化,从而使耐药突变体在培养过程中富集,而抗生素敏感突变体逐渐被消除(图8)。

图8
我们对所有样品进行了NGS分析,在对照组和抗生素处理组之间没有明显的插入分布差异,这表明大多数TF突变体的相对生长没有受到显著影响抗生素治疗(图9A)。通过比较抗生素处理样品中每个靶基因的插入读数与未处理对照样品的插入读数,我们能够识别出与抗生素抗性有关的基因。首先对亚胺培南处理样品分析,确定ampG、mucB、ampC、ampR、hptB、dksA、rsmA和algU基因的破坏会增加细菌对亚胺培南的易感性,几乎所有的靶向这八个基因的插入读数同时下降,支持了在亚胺培南存在下这些基因的破坏确实会减少细菌生长的观点(图9B、9C)。类似地,在头孢他啶处理后,靶向mexR、nalC和nalD基因的插入读数上升,表明这些基因的破坏有助于头孢他啶耐药。相反,algU、ampDh2和dksA基因的破坏降低了细菌对头孢他啶的耐药性。此外,确定了mexR、mexZ、nfxB、nalC和nalD基因的突变可以提高细菌对氧氟沙星的耐药性。在三个处理组中确定的基因中,我们注意到一些基因涉及两个或更多的处理组,这表明可能存在某些多药耐药性的普遍机制(图9D)。
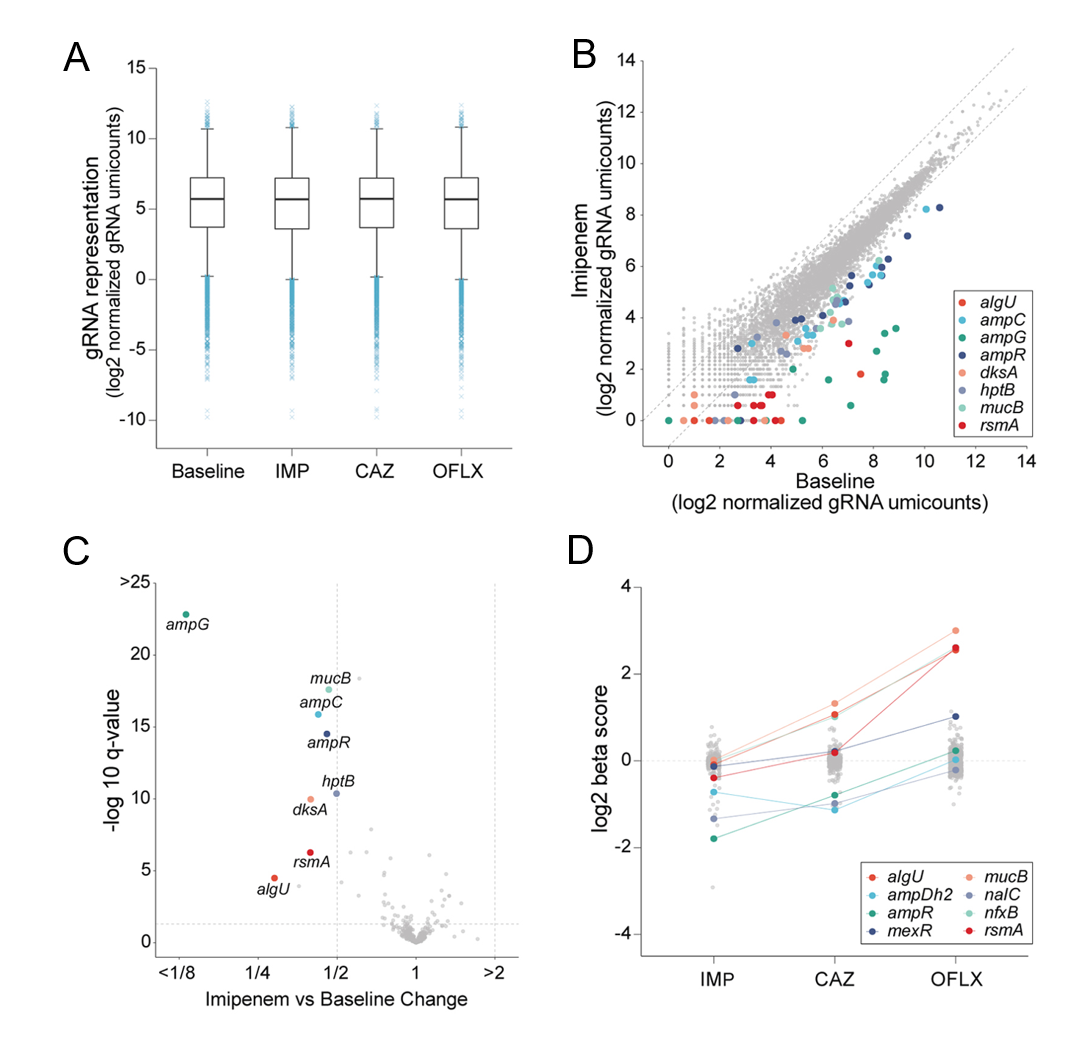
图9
突变体重建验证已识别的抗性/敏感基因
为了验证从STAGE文库中鉴定出的基因在抗生素耐药性中的作用,我们使用基于CRISPR-Cas9的基因组编辑技术重建了可靠的突变体。mucB、rsmA、hptB或dksA基因的失活显著降低了细菌对亚胺培南的耐药性。值得注意的是,当在亚胺培南存在下使algU失活时,没有观察到突变菌株的生长。在头孢他啶存在的情况下,mexR和nalD突变体的生长速度比野生型菌株快,而algU、dksA和ampDh2突变体的生长明显受到抑制。此外,mexR、mexZ或nalD基因的破坏增加了细菌对氧氟沙星的耐药性(图10)。所有这些表型都可以通过重新引入野生型基因来恢复。此外,我们检查了algU、mexZ和dksA基因在其他两种铜绿假单胞菌菌株PAK和PA14中的抗生素抗性作用,其结果与PAO1菌株的结果一致。总之,这些发现表明STAGE方法是一种有效且可靠的工具,可用于基因型-表型关系的高通量分析。
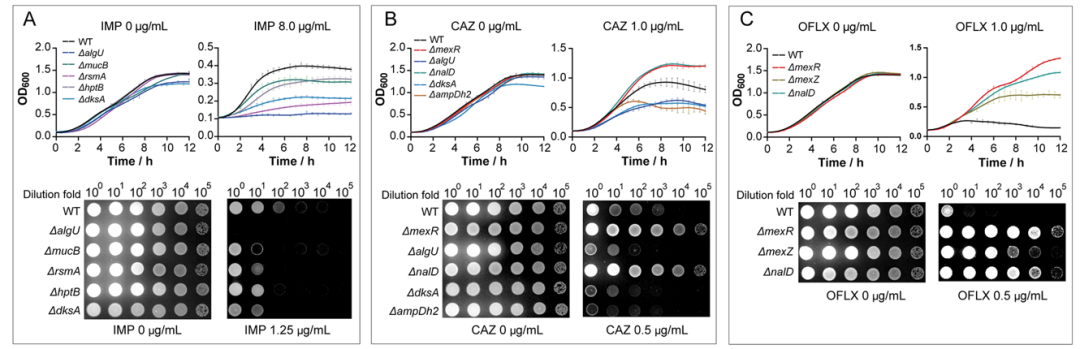
图10
在从STAGE文库鉴定的基因中,ampG、ampC、ampR和mucB先前已通过全基因组转座子筛选被鉴定为与亚胺培南抗性有关。MexR、NalC和NalD是MexAB-OprM多药外排泵的阻遏物。这些基因的失活会上调MexAB-OprM泵的表达,从而增加细菌对头孢他啶和氧氟沙星的耐药性。MexZ和NfxB分别是MexXY-OprD和MexCD-OprJ外排泵的阻遏物。mexZ和nfxB基因的失活导致MexXY-OprD和MexCD-OprJ外排泵的过度表达,从而导致氧氟沙星抗性。algU基因是参与藻酸盐生物合成的关键调节因子,它可以影响生物膜的形成,从而影响对抗菌剂的抗性,因此algU的突变导致菌株对亚胺培南和头孢他啶敏感。rsmA、dksA、hptB和ampDh2基因在细菌对亚胺培南或头孢他啶的耐药性中的作用仅在本研究中发现,其调控机制尚不清楚(图11)。

图11
综上所述,在这项研究中,我们报告了STAGE的方法,这是一种使用Cas12k引导的转座酶在不同微生物物种中进行高通量位点特异性基因组工程的方法。除基因破坏外,STAGE还可用于将功能基因组元件高通量整合到不同微生物物种的目标基因组位点中,这是以往依赖同源重组或随机转座子诱变的方法难以实现的。这种方法也可用于混合细菌群落中的多重基因破坏和物种特异性遗传操作。转座酶组件的未来工程和进化可以提高该系统的靶向特异性,并可能扩大其在真核细胞中的应用。
上海科技大学副研究员陈未中和实验室博士生任泽慧为本论文共同第一作者,实验室博士生柴国师参与了工作,上海科技大学季泉江教授和骆观正教授为共同通讯作者。本研究受到科技部重点研发计划、国家自然科学基金、上海市自然科学基金和广东省自然科学基金的支持。
本文链接(点击“阅读原文”跳转)
https://doi.org/10.1016/j.celrep.2021.109635
文 / 任泽慧
编 / 陈鸿萱
审 / Don
「
如果对这个方法感兴趣
欢迎联系我们更进一步交流
」
生物信息与表观组学
ID|luolab2017
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
文献阅读 热心肠 SemanticScholar Geenmedical
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文




















 1134
1134

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








