
对于培养未被培养的大多数微生物的创新
Innovations to culturing the uncultured microbial majority
Nature Reviews Microbiology [IF: 60.633]
DOI:https://doi.org/10.1038/s41579-020-00458-8
发表日期:2020-10-22
第一作者:William H. Lewis1
通讯作者:Thijs J. G. Ettema (thijs.ettema@wur.nl)1
合作作者:Guillaume Tahon, Patricia Geesink, Diana Z. Sousa
主要单位:1荷兰瓦赫宁根大学微生物实验室(Laboratory of Microbiology, Wageningen University and Research, Wageningen, The Netherlands)
写在前面
分享标题:Nature 综述:对于培养未被培养的大多数微生物的创新
文中名词解释
Enrichments富集:
在受控制的环境选择压力(如底物或温度)作用下,从分类学上不同的接种物演变而来的几种菌株的组合。
Pure cultures纯培养:
包含属于同一菌株的细胞的培养物,理想情况下起源于单个细胞或菌落,它们之间的遗传变异极小。
也称为无菌培养。
Co-cultures共培养:
确定的两个或多个菌株的组合,通常在实验室中人为地引入并一起生长,它们之间可能会建立种间代谢关系。
Isolation分离株:在同一样本或栖息地中发现的单个细胞,菌株或物种与其他的有物理上的分离。
Fluorescence in situ
hybridization(FiSH)荧光原位杂交:一种通过将荧光团偶联的寡核苷酸探针与生物样品中互补的靶分子(通常为16S rRNA)结合,用荧光信号标记细胞的方法。
探针可以被设计成具有高度的分类特异性,使它有可能在单细胞水平上分类鉴定微生物。
Dilution-to-extinction梯度稀释:一种连续稀释混合菌群培养物的方法,目的是分离将生长和分裂的单细胞,以建立单克隆和无菌培养物。
也可以称为有限稀释。
Growth factors生长因子:能被生物体用来促进生长的任何物质。
Symbiosis共生:两种或多种生物体的关联,通常是物理或代谢相互作用,通常会影响一个或多个参与伙伴的适应性。
Syntrophy互养共栖:
种间关系,一种物种产生的代谢产物被另一种物种用作生长底物。
Anaerobic厌氧:
在没有氧分子的情况下生长的生物。
Inocula接种物:
引入新鲜培养基中以启动新培养物生长的微生物样品。
Optical tweezers光钳:
一种通过显微镜和激光捕获从细胞悬液中分离单细胞的方法。
现在,许多光学镊子设置都是自动化的,并以微流控芯片运行。
使细胞通过悬浮液中的这些芯片,并捕获具有可检测表型的细胞,将其从主流移至无菌出口并收集。
Phenotypes表型:
受其基因(基因型)和环境因素影响的生物体的可观察或可检测特征。
Fluorescence-activated cell
sorting(FaCS)荧光激活细胞分选:根据自然或人工诱导的荧光特性(例如,通过荧光染色或标记技术)将细胞分散到单独的容器(例如试管或孔)中。Genome-resolved metagenomics基因组解析的宏基因组学:从宏基因组学数据重建基因组序列,通常通过生物信息学方法获得,其中将单个微生物的重叠群分组在一起(“结合”)。
Anoxic缺氧:一种完全没有分子O2的状态,例如在环境或培养物中。
Optical density光密度:
一种通常用于评估液体悬浮液细胞密度的分光光度法,通常是通过测量600 nm波长的光在穿过样品时其被细胞散射的程度。
Flow cytometry流式细胞仪:
一种基于物理或化学特性用于检测和计数细胞的技术。
MALDI-TOF mass spectrometry MALDI-TOF质谱:
MalDi是一种用于质谱分析的电离技术,其原理是将样品嵌入特殊的基质中,然后用激光将其解析。
该技术可以分析生物分子和有机分子。
摘要
尽管微生物基因组数据激增,但实验测试对于确认有关细胞生物学,生态作用和微生物进化的推论仍然很重要。由于大多数古细菌和细菌的多样性仍未被培养且特性描述欠缺,因此培养仍是当务之急。人们对有效的培养策略的兴趣和需求日益增长,这导致了许多方法学和技术的快速进步。在这篇综述中,我们讨论了可能阻碍分离和培养新微生物的常见困难,并综述了新兴的、创新的靶向或高通量培养方法。我们还重点介绍了成功培养新型古细菌和细菌的最新实例,并提出了未来培养尝试的关键微生物。
方框1. 经典的培养策略和方法
Box 1 Classical cultivation strategies and methods
生态学理论表明,与植物相关的微生物组是由宿主,微生物和环境之间复杂的相互作用所形成的。全基因组关联研究表明,宿主基因组确实会影响微生物组。
微生物培养的起源可以追溯到19世纪中叶,许多现代的培养工作都依赖于一个多世纪以前引入的一些相同的早期原则。可以采用几种策略来富集并分离出特定的微生物,其中许多依赖于直接观察培养物的生理行为以及所含微生物的表型和基因型特征(见图)。在选择最合适的分离措施时,微生物分离研究人员的经验也很重要。
富集特定物种分类群的技术示例包括:设计选择性营养培养基(例如,使用特定的底物),应用选择性的物理化学条件(例如温度,pH,盐度和气相组成),添加选择性抑制剂(例如,抗生素,有毒化合物和代谢抑制剂)以及增加或减少特定生长因子(例如,氨基酸、维生素和金属)。这些策略中的每一个对特定微生物种群的生长和数量的影响都可以被监测,并用来确定进一步的分离方法。
在显微镜下观察培养物可能是确定分离策略的有效的方法。例如,在培养过程中,当目标微生物与其他微生物在大小或形状上有实质差异时,可以使用通过具有各种孔径的过滤器进行大小分级和通过梯度离心进行基于质量的分离来分离它们。随着时间的推移,显微镜观察有时可以检测微生物的不同生长速度,这可以用来告知继代培养周期,以便选择生长更快的微生物(通过在较早的培养阶段转接培养物)。
在固体培养基(通常是琼脂)的表面上生长培养物和菌落挑选是分离微生物的常用方法,使用替代的固化剂(例如结冷胶和琼脂糖)可以针对不同的微生物。还可以通过梯度稀释和设计实验来选择运动性表型(例如趋光性,气动力性,趋化性,趋电性或趋磁性)来分离液体培养基中的微生物。
另一个考虑因素是用于对生长培养基进行灭菌的方法,最常见的方法是高压灭菌。但是,除了存在某些组分降解的风险外,高压灭菌过程中某些组分的存在还可能导致形成有毒副产物,例如过氧化氢,从而抑制生长。单独高压灭菌培养基成分或使用过滤灭菌法可以避免这些问题。
扩展阅读:再这么配培养基,你的细菌都被毒死了!
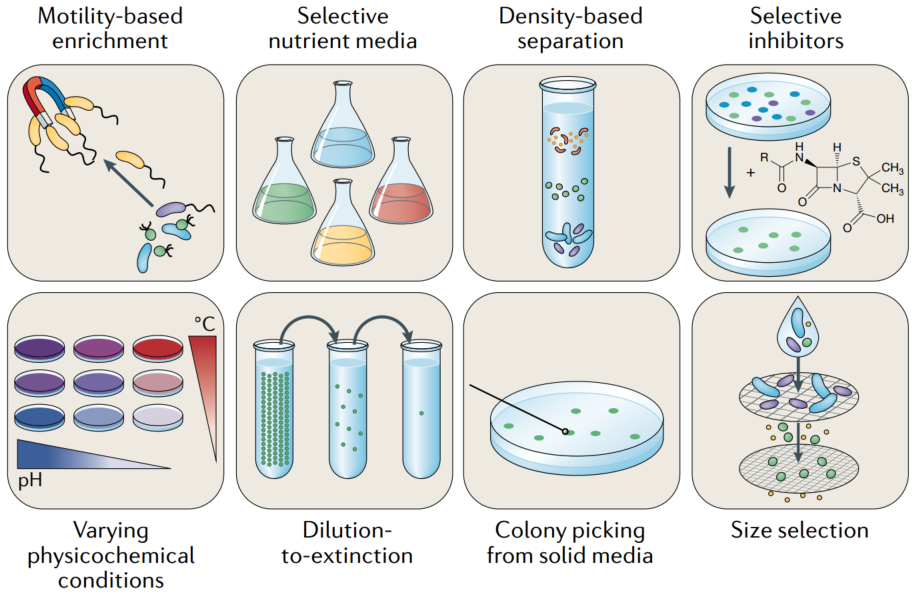
图1. 培养的细菌偏向于拟杆菌门、变形菌门、厚壁菌门和放线菌门
Cultured bacteria are biased towards Bacteroidetes, Proteobacteria, Firmicutes and Actinobacteria
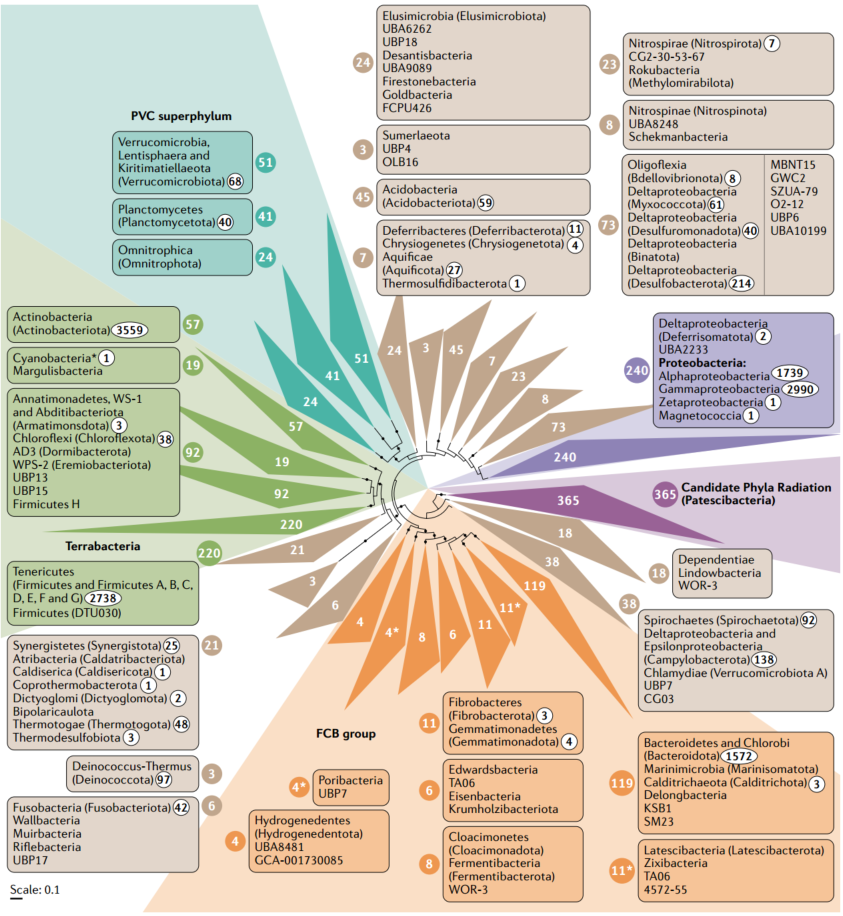
细菌的系统发生树,由每个物种总共15种核糖体蛋白中至少5种的连接比对推断,由从基因组分类数据库中获得的1,541个细菌基因组。彩色圆圈中以白色字体表示的数字是每个折叠的进化枝中各个分类单元的数目,还用于将相应的分类单元名称连接到进化枝。分类名称旁边的白色椭圆中的黑色数字表示根据BacDive数据库中分配给每个进化枝的物种类型菌株的数量(最后访问日期为2020年4月6日),针对这些分类描述的物种级培养分离株总数。没有编号的分类单元在BacDive中没有培养的分离记录。科学文献中已经报道了许多可培养的代表,但在该图中没有用数字表示出来,因为培养物还没有被官方描述和/或保存在培养保藏中,因此不包括在BacDive中(目前缺乏一个全面的数据库来记录所有培养细菌,包括未被官方描述或未保存在培养库中的细菌)。该树是从包含来自不同物种的同源蛋白质的数据集中生成的,使用MAFFT(L-INS-i)分别进行比对,然后将每种蛋白质的比对进行联结,从而将属于同一物种的那些蛋白质合并以形成一个序列。使用带有选项-gt 0.5的trimA1去除了串联比对中保守性较差的位点。使用IQ-TREE中LG + C60 + F + R10模型,通过1,000个超快速自展支持,从这种修剪对齐生成一个系统发育。用黑点标记的分支的支持值≥95%。考虑到用于推断这种系统发育的有限的蛋白质数据集,在某些情况下,某些物种或群体之间更深层次的关系可能不能反映基于更深入且得到更好支持的分析而被更广泛接受的关系。特别是,therinococcus - thermus (Deinococcota)和Chlamydiae (Verrucomicrobiota A)分别不与Terrabacteria和PVC superphylum的其他谱系分组。*尽管存在着众多蓝藻谱系的众多培养代表,但在BacDive中其代表性不足。与大多数细菌不同,由于历史原因,蓝细菌的分类大多采用植物法规(即藻类,真菌和植物的国际命名法)。结果,蓝藻缺乏明确的类型菌株,因此没有在BacDive中广泛列出,并且缺乏现有蓝藻培养物的综合数据库。
图2. 古细菌的多样性主要由未被培养的群体所主导
Archaeal diversity is dominated by uncultured groups
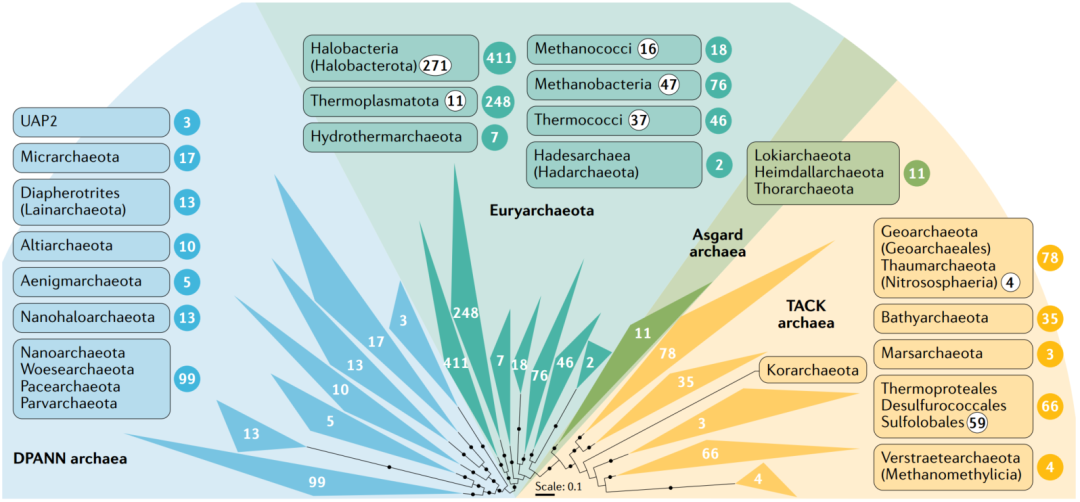
一种古细菌的系统发育树,由每个物种总共15种核糖体蛋白中至少5种的串联比对推导,由从基因组分类数据库中获得的1,166种古细菌基因组编码。彩色圆圈中以白色字体表示的数字是每个折叠的进化枝中各个分类单元的数目,还用于将相应的分类单元名称连接到进化枝。基于BacDive数据库中分配给每个进化枝的物种类型菌株的数量,分类名称旁边的白色椭圆中黑色字体的数字表示针对这些分类描述的物种水平培养分离株的总数(最后访问时间为2020年4月6日)。没有编号的分类单元则没有在BacDive中有记录的可培养分离株。在科学文献中已经报道了许多可培养的代表,但在该图中没有用数字表示,因为在培养物的收集中没有正式描述和/或保存这些培养物,因此没有将其包含在BacDive中。当前缺乏一个全面的数据库来记录所有可培养的古细菌,包括那些没有被正式描述或存放在培养物收集中的古细菌。该树是从含有来自包含不同物种的同源蛋白质的数据集中生成的,使用MAFFT(L-INS-i)分别对这些蛋白进行,然后将每种蛋白质的比对合并在一起,从而将属于同一物种的那些蛋白质合并在一起形成单个序列。使用trimAl(带有选项-gt 0.5)去除串联对齐中保守性差的位点。使用IQ-TREE152中LG + C60 + F + R10模型,通过1000个超快自展支持,从这种修剪比对中生成一个系统发育。用黑点标记的分支的支持值≥95%。考虑到用于推断这种系统发育的有限的蛋白质数据集,在某些情况下,某些物种或群体之间更深层次的关系可能不能反映基于更深入且得到更好支持的分析而被更广泛接受的关系。
图3. 使用高通量或靶向方法分离用于培养的新型微生物的工作流程
Workflows for isolating novel microorganisms for cultivation using high-throughput or targeted approaches
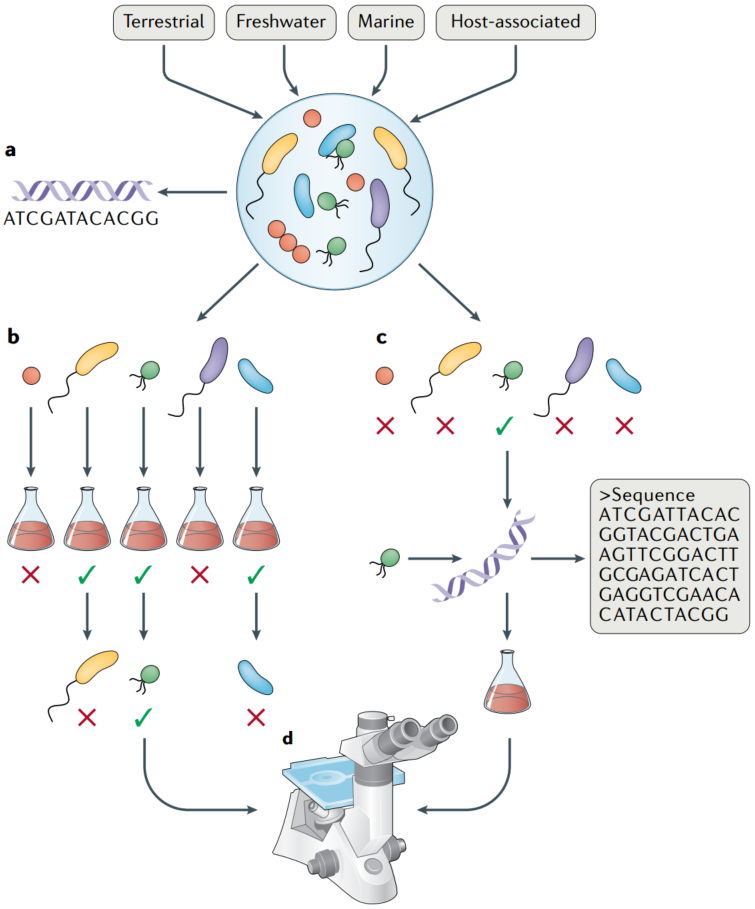
a|基于序列的栖息地筛选可用于识别具有高的目标生物相对丰度的位置,然后从这些位置收集细胞样品。
b|高通量方法可以通过用单细胞接种培养基来建立大量单培养物,孵育培养物,然后是生长筛选,然后筛选含有感兴趣的物种的活菌来实现。
c|靶向方法依赖于分离属于特定分类或功能组的细胞。
d|培养的分离株可用于下游表征和实验,以研究其生物学特性。
图4. 分离和培养新型微生物的创新方法
innovative methods for the isolation and cultivation of novel microorganisms
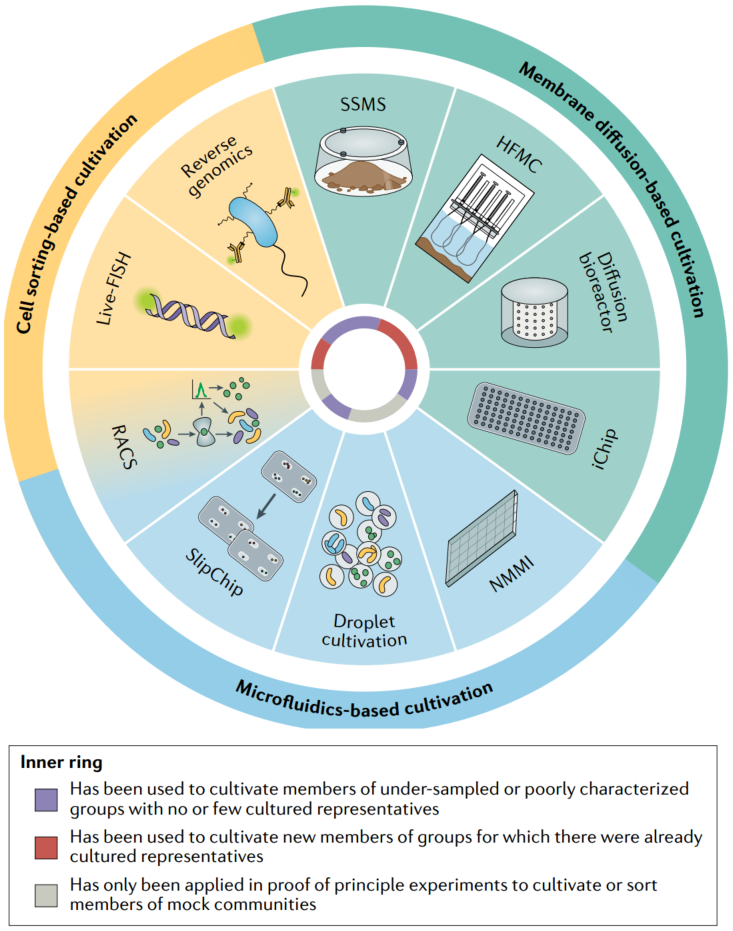
内环图例:
紫色:已被用来培养样本不足或特征欠佳的群体的成员,没有或只有很少的可培养的代表
红色:已被用来培养已经有可培养代表的团体的新成员
灰色:仅在被用于证明原理实验以用于培养或分类模拟的成员
基于膜扩散的培养方法(绿色),例如(分离)芯片,中空纤维膜室(HFMC),扩散生物反应器或土壤基质膜系统(SSMS),使用可渗透的膜,使营养物质和代谢产物扩散到培养基中,从而模拟培养过程中的更多自然条件。基于微流体的培养方法(蓝色),如纳米孔微尺度微生物培养箱(NMMI)或SlipChip,能够操纵小批量和大量重复的细胞,并且还可以与各种液滴培养方法结合使用。基于细胞分选的技术(黄色),例如拉曼激活细胞分选(RACS),活细胞的荧光原位杂交(live-FISH)或反向基因组学,提供了一种以细胞的功能或分类学子集为靶标进行分离的方法 。
图5. 用于新型微生物的靶向分离和培养的反向基因组学研究
reverse genomics for targeted isolation and cultivation of novel microorganisms
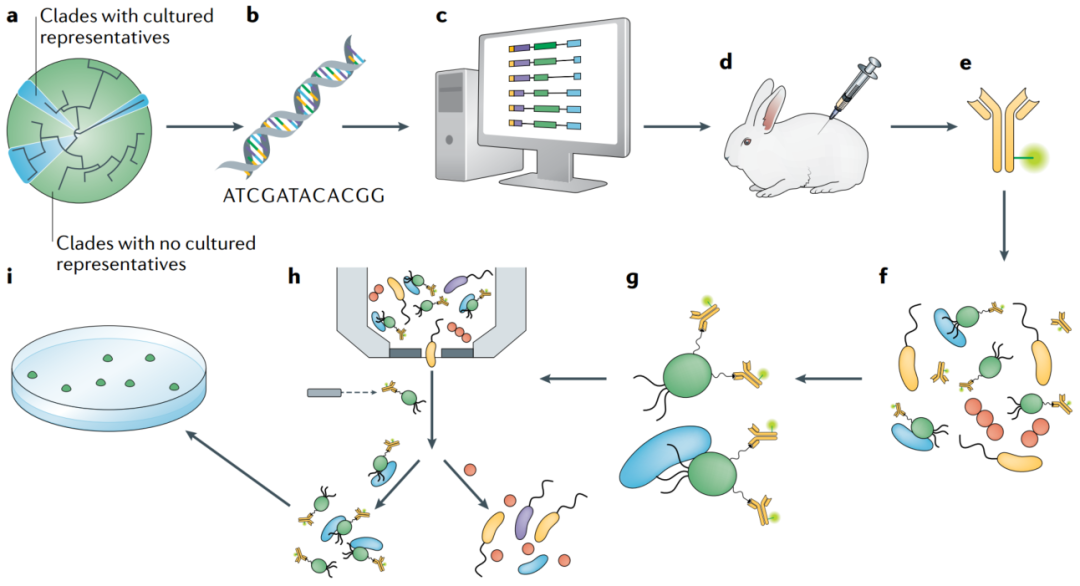
反向基因组学可用于新型谱系的靶向培养。
a|首先,鉴定属于新型或重要进化枝的目标微生物。
b|目标微生物的基因组可以从宏基因组数据中重建。
c|基于这些数据,可以预测蛋白质,并可以鉴定具有胞外结构域的高表达膜蛋白。
d|然后合成靶蛋白结构域抗原,并接种到合适的动物体内以生产抗体。
e|然后将产生的抗体纯化并偶联至荧光染料。
f|将抗体添加到环境细胞样品中。
g|抗体标记靶细胞。
h|然后可以基于赋予抗体的信号通过荧光激活的细胞分选对细胞进行分选。
i|将细胞分选到液体或固体生长培养基上。
如果靶标是彼此物理缔合的共生体,并且如果标记了一个细胞,则可以将两种微生物共同分选并用于接种一种互养共培养。
Reference
William H. Lewis,Guillaume Tahon, Patricia Geesink, Diana Z. Sousa,Thijs J. G. Ettema.Innovations to culturing the uncultured microbial majority. Nature Reviews Microbiology 19, 225–240 (2021). https://doi.org/10.1038/s41579-020-00458-8
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
文献阅读 热心肠 SemanticScholar Geenmedical
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文,跳转最新文章目录阅读




















 8873
8873











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








