点击蓝字 关注我们
基质尺寸和细菌运动性共同决定了早期阶段的海洋微塑料表面生物被膜组装

iMeta主页:http://www.imeta.science
方法论文
● 原文链接DOI: https://doi.org/10.1002/imt2.121
● 2022年6月7日, 中国海洋大学张伟鹏组在 iMeta 在线发表了题为 “Early stage of biofilm assembly on microplastics is structured by substrate size and bacterial motility ” 的文章。
● 本研究结合宏基因组学、16S rRNA基因扩增子分析、宏转录组学、qRT-PCR和可培养菌株的微生物学和遗传学实验,以玻璃颗粒表面的生物被膜为参考,解析了不同尺寸(3 mm和0.3 mm)的微塑料表面生物被膜群落的结构组成和功能特性。
● 第一作者:秦澎、崔涵
● 通讯作者:丁维 (dingwei@ouc.edu.cn) 、张伟鹏(zhangweipeng@ouc.edu.cn)
● 合作作者:李盼欣、王帅涛、范燊、卢洁、孙萌、张恒、王首刚、宿晓燕、付慧慧、胡晓丽、林金水、张玉忠
● 主要单位:中国海洋大学海洋生物多样性与进化研究所、海洋生命科学学院、海洋生物遗传与育种教育部重点实验室、深海圈层与地球系统前沿科学中心;延安大学生命科学学院;山东大学微生物技术国家重点实验室
亮 点

● 直径0.3 mm的微塑料表面生物被膜群落结构与直径3 mm的微塑料表面生物被膜群落结构存在显著差异。
● 相比于直径3 mm微塑料上的微生物群落,0.3 mm微塑料表面生物被膜中含有更丰富的运动和趋化性相关的基因,这表明该群落更具“运动性”。
● 通过细菌的分离纯化与培养实验,证实细菌在0.3 mm微塑料上的定植与其运动能力存在强相关性。
摘 要
海洋微塑料表面生物被膜群落的组装过程与微塑料在海洋环境中的迁移和生态学影响密切相关,因此理解微塑料生物被膜群落的组装机制至关重要。然而,微塑料的尺寸对于海洋原位环境中的微生物定植和生物被膜形成的影响尚不清楚。在本研究中,我们结合宏基因组学、16S rRNA基因扩增子分析、宏转录组学、qRT-PCR和可培养菌株的微生物学和遗传学实验,以玻璃颗粒表面的生物被膜为参考,解析了不同尺寸(3 mm和0.3 mm)的微塑料表面生物被膜群落的结构组成和功能特性。首先在潮下带区域放置微塑料和玻璃颗粒,进行为期10天和20天的生物被膜发育。16S miTags和16S rRNA扩增子分析表明多个细菌属,如Oleiphilus、Oleibacter和Alteromonas,在直径0.3 mm微塑料表面的10天生物被膜群落中特异性存在。宏基因组功能分析显示10天的0.3 mm塑料表面生物被膜富集了与运动性相关的基因,包括鞭毛组装和趋化基因。宏转录组和qRT-PCR的结果在原位水平证实了生物被膜群落中运动相关基因的高表达。对0.3 mm塑料表面生物被膜群落细菌进行单菌分离纯化和培养,并通过分离菌株的微生物学实验,发现细菌在直径0.3 mm塑料上的定植程度与质子动势驱动的细菌运动能力存在强关联。最后通过基因敲除和定植实验,发现与野生型菌株相比,ΔmotAB突变株在0.3 mm微塑料上的定植能力显著降低。因此,我们提出细菌运动性和基质尺寸共同决定了海洋微塑料生物被膜形成早期阶段的群落组装。
视频解读
Bilibili:https://www.bilibili.com/video/BV19c411M7AG/
Youtube:https://youtu.be/Ri62DrGxTgI
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
微塑料颗粒(MPs)被定义为直径小于5 mm的塑料碎片,是海洋中塑料的主要存在形式。当前微塑料污染已经成为了海洋生态系统中最严重的威胁之一,这主要是由于其生物降解性差并且携带有害化合物,其中很多是持久性有机污染物。最丰富的海洋MPs类型是聚乙烯,其次是聚丙烯、聚氯乙烯和聚苯乙烯。MPs表面的疏水性促进了细菌的定植和生物被膜的形成。MPs表面的这种生物被膜被定义为“塑料际”,是在一项对北大西洋的MPs研究中首次提出。海洋生物被膜几乎存在于浸泡在海水中的所有基质表面,包括动物体表、岩石表面、有机颗粒和沉积物-水界面。因此,“塑料际”代表了一个特定的生物被膜群落。MPs表面形成的生物被膜反过来可以促进MPs的扩散。首先,生物被膜的形成会导致MPs密度增加,浮力降低。其次,生物被膜基质是粘性的,可促进异质聚集体的形成。MPs生物被膜的形成也会影响海洋生态系统中的元素循环,例如,MPs的存在已被证明会改变氮循环过程。上述这些研究揭示了MPs相关的微生物群落的重要性及MPs的环境影响,表明了MPs生物被膜的群落组装机制研究具有重要意义。
目前已有的一些研究发现多种因素会影响MPs生物被膜的群落结构。例如,一项研究指出,MPs相关生物被膜中的微生物多样性和均一性高于海水中的微生物,并且它们受到聚合物类型和暴露时间的共同影响。另一项关于影响塑料碎片上细菌定植环境因素的研究表明,海水盐度是影响MPs生物被膜中微生物多样性的主要因素。有趣的是,塑料的颜色也被发现会影响MPs相关生物被膜的群落结构和功能组成。此外,一些研究也关注了MPs尺寸(直径)与MPs生物被膜功能特性之间的关系。例如,在废水处理系统中,MPs的尺寸会影响抗生素抗性基因的环境行为。然而,MPs的尺寸对原位海洋环境中的细菌定植的影响机制仍不清楚。
在本研究中,我们在沿海海域进行了生物被膜形成实验,具体是将两种尺寸(3 mm和0.3 mm)的MPs和玻璃颗粒(GPs)放置于潮下带海水中,在生物被膜发育10天和20天后将其分别回收。我们通过宏基因组学分析、16S rRNA基因扩增子分析、宏转录组学分析和qRT-PCR,阐明MPs表面形成的生物被膜的群落组成结构和功能特点。随后,我们进行了大量基于可培养菌株的实验来支持和验证组学分析得出的结论。
结 果
MPs和GPs生物被膜中及海水中的群落分类结构
实验设计如图1A所示。MPs在被放置入海水中前,使用扫描电子显微镜(SEM)观察其表面特征,没有发现明显的差异(图S1)。总共收集了27个生物被膜和海水样本,从这些宏基因组中提取的miTags以97%的序列相似性进行OTU聚类,共生成18,356个OTUs。
将OTU进一步划分门水平,共生成78个门(变形菌门被进一步分类到纲水平),门水平的群落结构如图1B所示。在海水微生物群落中α变形菌纲丰度最高,其次β变形菌纲、放线菌门和泉古菌门在海水微生物群落中比在生物被膜群落中的丰度更高(图1B)。而γ变形菌纲和拟杆菌门是所有生物被膜群落中丰度最高。与其它生物被膜群落结构不一样的是,0.3-MPs-10的生物被膜群落较为独特,其γ变形菌纲占比61.57%,且在门水平上的物种多样性更低(图1B)。此外,从0.3-MPs-10发育到0.3-MPs-20的生物被膜群落组成存在明显的结构转变,具体表现:相较于0.3-MPs-10生物被膜,0.3-MPs-20生物被膜群落中γ变形菌纲的相对丰度下降了3.0倍,而拟杆菌门的相对丰度上升了3.6倍(图1B)。在属水平上,0.3-MP生物被膜与其他生物被膜群落结构明显不同(图1C)。在0.3-MPs-10生物被膜中,优势属为Oleiphilus(相对丰度14.71%)和Oleibacter(相对丰度14.35%),而它们在其他生物被膜群落中表现出较低的丰度(图1C)。与其他生物被膜和海水微生物群相比,Alteromonas在0.3-MPs-10生物被膜群落中大量富集。此外,0.3-MP不同发育时间生物被膜群落在属水平的物种组成也不一样,如在0.3-MPs-20中并没有检测到0.3-MPs-10生物被膜群落的优势属Oleiphilus(图1C)。
随后利用主坐标分析(PCoA)研究了不同时期不同介质材料及尺寸大小的生物被膜群落与海水微生物群落的异质性。总的来说生物被膜和海水微生物群落之间存在明显的界限(图S2)。分别对发育10天、20天生物被膜和海水微生物群落物种组成的差异分析发现,与其他聚集在一起的生物被膜样品不同,0.3-MP生物被膜(10天、20天)与其它所有样品均呈现明显的分隔(图S2)。总之,PCoA分析揭示了0.3-MP生物被膜,特别是0.3-MPs-10生物被膜群落的独特性,这与群落物种组成结果一致。由于上述分析是基于miTags,因此我们进一步使用了16S rRNA扩增子(V3-V4区域)测序来验证上述结果。研究发现,结果与miTags的方法一致(图1B)。

图1. 实验设计与生物被膜和海水群落的物种组成结构
(A)实验设计。在潮下带区域,在两种尺寸(3和0.3mm)的微塑料和玻璃颗粒上进行生物被膜发育。发育10天和20天后,将生物被膜样品转移回实验室,进行宏基因组(三个生物重复)和宏转录组测序和分析。用分离的单菌株进行实验并验证组学分析的结果。(B-C) 来自27个宏基因组的miTags的物种分类组成分析。丰度最高的30个门(B)或属(C)被展示,其余的合并为“其它”。变形菌门被分类到纲。3 mm和0.3 mm的玻璃颗粒、3mm和0.3mm微塑料颗粒上10天生物被膜分别称为3-GP-10、0.3-GP-10、3-MPs-10和0.3-MPs-10。20天生物被膜被称为3-GP-20、0.3-GP-20、3-MPs-20和0.3-MPs-20。
运动性和趋化性基因在0.3-MPs-10生物被膜中的富集及其原位高表达
由于0.3-MPs-10生物被膜群落与其他不同生物被膜、海水微生物群落相比,存在着独特的物种组成结构,我们使用单因素方差分析鉴定0.3-MPs-10生物被膜中富集的功能基因。通过KEGG数据库注释,发现749个功能基因在0.3-MPs-10生物被膜群落中大量富集(p-value < 0.001)。这749个功能基因主要参与了20个不同的基因通路,其中较多的基因(接近40个)与细胞运动性有关(图S7)。值得注意的是,有7个基因与鞭毛的生物合成、调节有关,如flgK:(编码鞭毛钩相关蛋白1,K02396)和fliK(编码鞭钩长度控制蛋白,K02414),且都在0.3-MPs-10生物被膜中大量富集(图2A)。
上述结果表明在0.3-MPs生物被膜发育的早期阶段,可能存在一个更具“运动性”的群落。为了证实这一结论,我们进一步使用DESeq2分析不同介质尺寸(0.3-MPs-10 VS 3-MPs-10)早期生物被膜功能基因的特点。结果发现1460个功能基因在MPs-10生物被膜群落中发生了显著变化(p-value < 0.001)。几乎所有与鞭毛相关的基因在0.3-MPs-10生物被膜中都表现出显著富集(fold change > 2; p-value < 0.001)(图S9)。例如,参与鞭毛组装的fliH基因表现出最大的倍数变化(图S9)。相较于3-MPs-10,其他高丰度的基因(如fliC、flgE和motA)都在0.3-MPs-10中被富集,其相对丰度增加了两倍以上(图S9)。此外,进一步探究fliC和flgE在0.3-MPs-10宏基因组中的物种组成,发现这两个功能基因主要在Alteromonas属、Cognatishimia属和某些玫瑰杆菌组成员(如Sulfitobacter、Roseovarius和Ruereria)细菌中存在(图S10)。
在原位水平上,通过宏转录组分析继续探究不同介质尺寸(0.3-MPs-10 VS 3-MPs-10)早期生物被膜中功能基因的表达情况。结果发现,运动相关基因和其他功能基因之间存在不同的分布模式,运动相关基因趋向分布在0.3-MPs-10生物被膜群落中,而其他基因趋向于分布在3-MPs-10生物被膜群落中。0.3-MPs-10中运动相关基因的表达水平也远高于3-MPs-10中的表达水平(图2C)。例如fliK(K02414)、flgF(K02391)、fliC(K02406)和mcp(K03406)(图2D)。此外,我们探究了0.3-MPs-10中fliC和flgE基因的活跃物种组成,结果显示属于Alteromonas、Pseudoalteronomas、Sulfitobacter、Thalassolitus和Oleiphilus的细菌在0.3-MPs-10生物被膜群落中存在高水平的原位表达,因而被认为是0.3-MPs-10生物被膜群落中最具运动性的微生物种类(图S11)。
随后qRT-PCR被进一步用于检测flgE基因在3-MP和0.3-MP生物被膜群落中的原位表达情况。通过对flgE基因在不同种细菌类群(Alteromonas、Leisingera、Phaeobacter、Thalassolitus和Vibrio)和不同发育阶段(5、10和15天)的生物被膜群落中的表达量进行分析,发现flgE在0.3-MP生物被膜群落中的所有五个属中都存在高水平的表达量,但在3-MP生物被膜群落中仅在两个属(Alteromonas和Vibrio)中存在极低的表达量(图S12)。此外,flgE基因存在阶段特异性的表达模式,如在0.3-MP生物被膜群落中,仅在早期生物被膜群落中(5或10天)检测到flgE的高表达量,而在晚期生物被膜群落中(15天)没有检测到表达(图S12)。

图2. 宏基因组学揭示10天生物被膜群落的差异表达基因,以及3-MPs-10和0.3-MP10生物被膜中细胞运动相关基因的表达水平
(A)前7个显著差异(p-value<0.001)的运动相关基因的功能注释和丰度分布(用单因素方差分析中的F值表示)。(B)基于宏转录组的0.3-MPs-10和3-MPs-10生物被膜中全部基因和细胞运动相关基因的相对丰度。基因的相对丰度显示为指数形式。(C)所有运动相关基因表达水平的排序。(D)基于宏转录组学揭示的两个生物被膜之间表达水平的倍数变化(FC=0.3-MPs-10/3-MPs-10)。
细菌的运动性与其在0.3-MPs表面定植之间的相关性
上述结果表明活跃细菌的运动性与其在0.3-MPs的定植存在一定关联。因此,通过从浸泡在海水中一个月的0.3-MPs生物被膜群落中分离细菌,以便后续进行单菌的实验验证。由于未分离到0.3-MPs生物被膜群落物种组成中相对丰度前两位的Oleiphilus属或Oleibacter属细菌,因此我们选择了一株属于Alteromonas的菌株作为候选菌株,该菌所在属的相对丰度在0.3-MPs生物被膜群落物种组成中排名前三。对Alteromonas菌株进行全基因组测序与分析,将其命名为A.sp009811496 PMMA93(以下简称PMMA93)。对PMMA93的代谢途径进行分析,发现PMMA93的鞭毛生物合成途径相对完整,包括约34个编码鞭毛马达、定子、基体、近端杆、环、钩、丝和帽等相关蛋白质的基因(图S14)。PMMA93基因组具有完整的Embden-Meyerhof-Parnas途径和三羧酸循环的所有基因(图S14),表明该细菌是一株异养细菌。PMMA93存在多个硝酸盐和亚硝酸盐的代谢基因,表明其可能具有无氧呼吸的潜力。同时,还发现PMMA93具有多个参与生物被膜形成、趋化相关的功能和调控基因(图S14)。此外,PMMA93拥有完整的呼吸链(图S14),可为细菌的游动和涌动提供能量。为了证实PMMA93的运动能力,以Polaribacter dokdonensis PC73被作为阴性对照(在0.3-MPs-10生物被膜宏基因组中未检测到Polaribacter属,以下称PC73),研究了两株菌的游动和涌动能力。结果表明,在0.3%(w/v)琼脂浓度的平板上,经过25℃ 5天的培养,PMMA93具有较强的游动能力,几乎占据了整个平板面积,而PC73没有显示出运动迹象(图S15)。在0.5%(w/v)琼脂浓度的平板上,经过5天的培养,PMMA93形成了直径为2.7cm的松散菌落(图S15),表明其发生了涌动。上述结果表明,PMMA93具有较强的游动和涌动能力,而PC73不具备运动能力。
为了验证细菌的运动性对微塑料生物被膜形成的影响,在PMMA93与3-MP或0.3-MP共培养的体系中加入不同浓度的羰基氰化物3-氯苯基腙(CCCP,一种可以消除质子动势PMF的化合物)以抑制细菌的鞭毛运动。孵育12小时后,对两种MPs上的生物被膜群落进行SEM观察(图3A),随后两种MPs上的生物被膜进行OD600的生物量测定(图3B)和菌落计数(图3C)。结果表明,不同浓度的CCCP处理对0.3-MPs上的细菌定植产生了不同程度的影响,12.5 µΜ和25 µΜ的CCCP分别抑制了50%以上和75%左右的细菌生物被膜生物量。相反,CCCP对3-MPs上的细菌定植没有显著影响。
为了进一步探究细菌在MPs上的定植,我们将运动菌株PMMA93和非运动菌株PC73进行了混合(1:1),并与0.3-MP或3-MPs进行共培养实验。通过16S rRNA基因扩增子测序,探究混合细菌在生物被膜状态(细菌定植在MPs表面)和在浮游状态下的丰度比例。结果显示,无论MPs尺寸如何,非运动菌株PC73在浮游状态下约占比50%(图S16A)。然而,在3-MPs表面的定植细菌中,非运动菌株PC73的占比下降至29-37%,在0.3-MPs表面的定植细菌中下降至10-15%(图S16A)。统计分析显示,3-MPs生物被膜中的非运动菌株PC73占比显著高于0.3-MPs(图S16B)。相反,在0.3-MPs的生物被膜中运动菌株PMMA93的丰度占比最高(图S16A和S16B)。也就是说运动菌株 PMMA93能更好地定植在0.3-MPs表面。
为了证实这一发现的普遍性,我们探究了CCCP对其他运动菌株在MPs表面定植的影响。我们选用了从海洋MPs中分离的其它三株菌株,包括Vibrio alginolyticus 2-8、Stentrophomonas pavanii P5G5和Tritonibacter mobilis 2-12。与PMMA93的结果类似,CCCP在很大程度上抑制了这三株细菌在0.3-MPs上的定植,而CCCP对其在3-MPs上定植的影响较小(图S17)。最后,为了进一步证实运动性和细菌定植之间的相关性,构建了Vibrio alginolyticus 2-8的motAB基因(侧鞭毛基因簇)突变菌株,结果发现ΔmotAB突变株在0.3-MPs表面的细菌数量与野生型相比表现出显著降低,但其在定植3-MPs上与野生型没有显著差异。
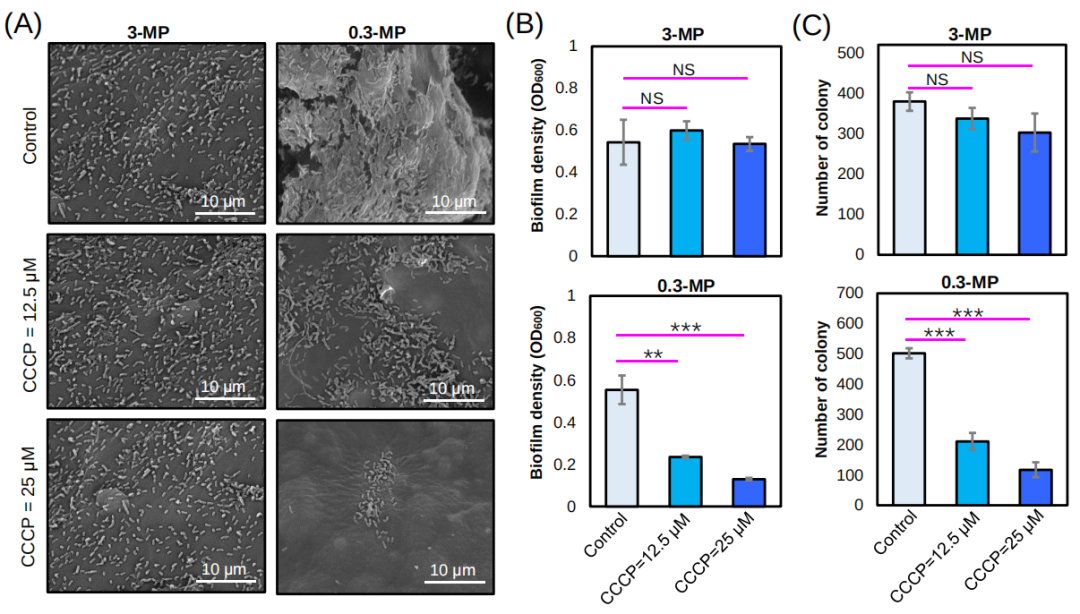
图3. 用不同浓度的羰基氰化物3-氯苯基腙(CCCP)处理的Alteromonas sp009811495 PMMA93的生物被膜
将PMMA93与3-MP或0.3-MP及添加0、12.5和25 μm CCCP共孵育12小时,然后进行扫描电镜成像(A)、细胞密度测量(B)和菌落计数(C)。发现不同浓度的CCCP处理对PMMA93在0.3-MPs上的定植的影响有显著差异,而对其在3-MPs上的定植没有显著影响。误差线代表三个生物重复的标准偏差。**表示p值<0.01,***表示p值<0.001,而NS表示不显著。
讨 论
在本研究中,我们通过将不同尺寸的微塑料和玻璃颗粒放置于潮下带原位海水中,探究了微塑料尺寸对微生物在微塑料表面定植的影响。基于miTags和16S rRNA基因扩增子分析,我们发现在0.3-MPs上形成的生物被膜(特别是在早期(发育10天))具有独特的物种组成结构和功能途径。运动相关基因的富集促使我们采用单菌株进行实验验证,以证明细菌的运动性与其在小尺寸微塑料上定植之间的关系。
有人认为,塑料际“只是”一种普通的生物被膜,与其他表面(如潮下带岩石表面)上的生物膜相比并不特殊。已有的一些研究发现的确支持这一观点,比如MPs和其他材料上微生物群落之间的差异通常是由稀有细菌类群所决定的,而高丰度的细菌类群没差异,并且环境变量对群落结构的影响通常比介质材料的影响大得多。不同地是,本研究证明了0.3-MPs上生物被膜群落物种结构独特性,特别是在其早期阶段,Oleiphilus和Oleibacter等高丰度为主要优势属。已有研究发现Oleiphilus能在油滴表面形成生物被膜,而Oleibacter能够形成厚鞭毛丝的巨大鞭毛蛋白,据推测这可能有助于它们适应特定的生态位。而这两个属细菌的存在与功能分析结果一致,即0.3-MPs-10生物被膜中存在大量运动相关的基因。此外,与0.3-MPs-10相比,0.3-MPs-20生物被膜群落在物种结构上有所不同,这可能是随着生物被膜的发育,群落内作用力的影响越来越大造成的。这与先前的一些关于生物被膜发育的研究具有类似的结果。例如,在潮下带区域,相比于基质类型,发育时长对于生物被膜群落的物种组成影响更强。这些研究结果表明,这种群落内作用力可能涉及物种间的相互作用,进而减少了生物被膜基质对微生物类群的影响,并且这种相互作用可能导致某些先驱物种(例如Oleiphilus)被取代。
宏基因组学和宏转录组学的结果都表明,0.3-MPs-10生物被膜群落上存在更为“运动”的群落。从机制上讲,0.3-MPs表面的物理特征会使得附着细菌遇到更强的水动力,因而其生物被膜群落可富集运动相关的基因。由水流和水中颗粒物质的布朗运动所产生的水动力可以被细菌游动的“动能”部分抵消,同时也防止了非运动细菌的附着。这解释了Oleiphilus和Oleibacter之所以具有广泛的降解石油的能力,因为它们需要增加动力来克服油-海水界面的静水压力。同样地,有研究发现运动增强了Halomonas菌株在表面活性剂涂层油滴上的粘附。此外,与许多颗粒不同,MPs可以带静电,这可能会增加细菌定植的难度,并导致MPs和GPs之间物种组成的差异,因为与GPs-10生物被膜相比,MPs-10中的一些运动相关基因表现出更高的丰度。此外,0.3-MPs较大的比表面积可能有助于运动细菌的附着,本研究发现运动细菌在密闭空间(如装颗粒的网袋)中接触到这些表面的机会更大。除此之外,趋化性相关基因的普遍存在意味着MPs释放的化合物可能在招募特定细菌物种中发挥着作用。例如,塑料添加剂和塑料寡聚物可以从MPs中释放到水中,并可能被某些微生物类群所利用。这一观点与之前的研究发现化学属性明确的颗粒被浸泡在沿海海水中细菌的运动性和颗粒的消耗能力都有助于微生物群落的组装这一结果一致。这些微生物群落,在140小时内经历了快速的演替,从一个能够降解碳颗粒的群落发展成一个丧失降解能力的群落,本研究中的情况可能也是如此。然而,MPs释放的化学物质的作用及其对生物被膜群落组装的贡献还有待进一步研究。
为了验证原位实验结果,我们对四种分离的代表菌株进行了微生物学实验。与原位实验结果一致,微生物学实验结果表明了运动性在细菌定植0.3-MP过程中的重要性。由于细菌运动是由PMFs驱动的,我们的实验也证明了细菌定植可能是一个耗能过程,这与之前的观察结果一致,即鞭毛运动是一个复杂的细胞过程,需要对宿主定植进行高能量投入。此外,运动菌株和非移动菌株的共培养实验表明,在0.3-MPs的细菌定植过程中,运动性可能有助于提高细菌的竞争力,这一发现也符合之前研究的结论,即细菌运动性在定植养分碎片时会带来更大的竞争优势。最后,采用敲除了侧鞭毛(弧菌侧鞭毛特别作用于弧菌在表面涌动而非在液体中游动)基因簇中motAB基因的弧菌突变株的定植实验,进一步证实了运动性在0.3-MPs定植中的作用。
本研究存在几个局限点。首先,原位生物被膜的发育只选择了一个位置,没有考虑环境波动的影响。尽管在之前的几项研究中已经记录了环境参数对塑料际微生物群落组成的影响,但其潜在的功能基础尚不清楚,这将是我们未来努力的方向之一。此外,仅选择生物被膜发育的两个阶段进行宏基因组和宏转录组分析。为了弥补这一限制,我们进行了qRT-PCR,结果表明,早期发育阶段( 5天或10天)0.3-MP生物被膜中运动相关基因的表达高于3-MPs生物膜中的表达。此外,仅选择了四个分离菌株来验证原位实验的结果。为了克服这一短板并进一步理解海洋生物被膜的发育,我们目前正在尝试构建一个由数百个分离菌株组成的合成群落,该群落将被用作模型群落,用以探究菌株水平和单个细菌水平的海洋生物被膜的组装机制。
结 论
结合组学分析和微生物实验的结果,我们提出基质尺寸和细菌运动性共同介导了海洋微塑料生物被膜形成早期阶段的群落组装,并形成了独特的微生物群落。这些发现进一步刷新了我们对生物被膜组装和微生物表面相互作用的理解,而这些可能正是微塑料在海洋环境中的生态作用和迁移的基础。
主要方法
材料与生物被膜发育
介质尺寸为 3 mm和0.3 mm,MPs密度为0.94-0.96 g/cm3,GP密度为2.5 g/cm3。尼龙网袋用于装载颗粒,颗粒约占袋体积的五分之一。这种设置允许海水自由交换,同时防止颗粒逃逸。颗粒和尼龙网袋用无菌海水清洗三次,然后进行高压蒸汽灭菌。
考虑到生物被膜在海洋环境中的发育存在不同的阶段,我们收集了不同尺寸MPs和GPs上形成的10天和20天生物被膜。每种类型的基质都有三个重复,并使用浮标将网袋悬浮在2米深的海水中。此外,在实验期间,在2m的深度收集了三个海水样本(5L/样品),因此总共有27个样本。生物被膜样品送回实验室后,用无菌海水冲洗MP和GP三次以去除松散附着的细胞后收集样品以便后续分析。
高通量测序和分析
使用DNeasy®PowerWater®试剂盒(Qiagen,Hilden,Germany)提取总群落DNA。总DNA的宏基因组和16S rRNA扩增子测序采用诺和致源的Illumina NovaSeq 6000测序平台,进行PE250双端测序。对3-MPs-10和0.3-MPs-10生物被膜进行宏转录组提取总RNA,并采用Novogene的HiSeq X Ten平台进行测PE250双端测序。所有数据数据下机后,采用本地服务器上的NGS QC Toolkit软件(2.3.3版)进行Illumina序列的质量控制。然后,对含有接头的reads、低质量的(质量分数<20)reads或不能匹配的高质量reads进行去除。具体的分析方法参见引文。
细菌运动性测定
对两株分离菌株进行运动性测定。用0.3%和0.5%浓度的Noble琼脂(Sangon Biotech,Shanghai,China)固化海洋肉汤2216E培养基。将在2216E液体培养基中生长到对数生长期(OD600=0.7)的细菌接种在琼脂平板上(表面下0.3cm)。接种的平板在恒定湿度(10%)和温度(25℃)下孵育,并记录菌落大小。每个处理做三个重复。
细菌定植实验与细胞计数
在2216E液体培养基中生长到对数期的细菌用于定植实验。将细胞与3-MPs或0.3-MPs以及不同浓度的CCCP在6孔板(Sangon Biotech,Shanghai,China)中于25℃孵育12小时。在这些浓度下,CCCP对受试菌株的生长没有影响。使用无菌的2216E培养基洗去定植在MPs上的细菌,使用Biotek Cytation5分光光度计(Biotek Instruments,Winooski,USA)以OD600对定植的细菌进行定量。每个处理做三个重复。同时,将附着在MPs上的细菌冲洗下来,用无菌海水稀释105倍,涂布在2216E琼脂平板上。在25℃下培养24小时后,对菌落计数。每个处理做三个重复。
细菌的共培养和定植
两个菌株在2216E液体培养基中生长到对数期,以1:1的比例混合。将混合细菌与3-MPs或0.3-MPs在6孔板中于25℃下孵育12小时。收集MPs生物被膜和培养基中的浮游细菌,使用V3V4通用引物(16S-341F和-805R,如上所列)提取DNA和扩增16S rRNA基因。在Novogene(中国北京)的Illumina PE250平台构建文库进行测序,测序深度为30,000条paired-end reads。使用Bowtie2将clean reads映射到从细菌基因组预测的16S rRNA基因序列。映射的reads数量用于计算基于两个目标菌株的16S rRNA基因拷贝数标准化后的细菌占比。每种处理及其测序做三个重复。
基因敲除
按照我们之前研究中描述同源重组方法,对V. alginolyticus 2-8中的motAB基因进行敲除。引物motAB-up-F/motAB-up-R和motAB-down-F/motAB-down-R,见表S7。使用重叠PCR将上游和下游片段连接后得到重叠PCR产物后,插入到自杀质粒pDM4的SphⅠ/SalⅠ位点上,构建敲除质粒pDM4-ΔmotAB。将构建好的质粒pDM4-ΔmotAB转化到大肠杆菌S17-1中。在TSBNa(含有3% NaCl的TSB培养基)琼脂培养基上,将E. coli S17-1(pDM4-ΔmotAB)与V. alginolyticus 2-8进行质粒接合转移,28℃孵育48 h。将细胞用PBS悬浮后接种到含有30 μg/ml氯霉素和100 μg/ml卡那霉素的TSBNa琼脂平板上,以筛选V. alginolyticus的一次重组子。从筛选得到的20个候选菌落中,挑选出来转移到TSBNa培养基中,并在28℃下孵育过夜,然后在含有12%蔗糖的TSBNa琼脂平板上进行筛选。使用引物对(motAB-up-F/motAB-down-R)进行PCR验证二次重组突变体,以确认motAB基因的敲除,并进行Sanger DNA测序验证。
数据可用性声明
本研究的宏基因组、16S rRNA基因扩增子和宏转录组数据集以登录号PRJNA902427上传到NCBI (https://www.ncbi.nlm.nih.gov/sra/PRJNA902427)。Alteromonas sp009811495 PMMA93的全基因组以登录号SAMN32034847上传到NCBI(https://www.ncbi.nlm.nih.gov/nuccore/CP113972)。补充材料(方法、图表、表、图形摘要、中文翻译版和更新材料)可通过在线DOI或iMeta Science(http://www.imeta.science/)获取。
引文格式:
Qin, Peng, Han Cui, Panxin Li, Shuaitao Wang, Shen Fan, Jie Lu, Meng Sun, et al. 2023. Early stage of biofilm assembly on microplastics is structured by substrate size and bacterial motility. iMeta e121. http://doi.org/10.1002/imt2.121
作者简介

秦澎(第一作者)
● 中国海洋大学海洋生物多样性与进化研究所与海洋生命学院博士研究生。
● 研究方向为海洋生物被膜群落组装与资源开发。曾获优秀研究生干部等荣誉。

崔涵(第一作者)
● 中国海洋大学海洋生物多样性与进化研究所与海洋生命学院博士研究生。
● 研究方向为海洋生物被膜群落组装机制。

丁维(通讯作者)
● 中国海洋大学海洋生命学院海洋生物遗传与育种教育部重点实验室副教授,硕士生导师。
● 研究方向为海洋泛生物被膜群落的物种与功能连通性。近年来围绕虾贝共附生微生物和近海生物被膜展开研究工作,指示了以生物体和非生物体为介质的泛生物被膜群落的存在。通过揭示不同群落之间的连通性从而交叉性地从生态学角度出发解决水产养殖的实际问题。以第一或通讯在iMeta、Nature Communications、mLife、Applied and Environmental Microbiology、Marine Life Science & Technology、Microbial Ecology等期刊发表论文10余篇。

张伟鹏(通讯作者)
● 中国海洋大学海洋生物多样性与进化研究所、海洋生命学院教授,博导,独立PI。
● 担任山东微生物学会海洋微生物专业委员会委员,山东微生物学会科普工作委员会委员,The Innovation和iMeta青年编委。研究方向聚焦海洋生物被膜(Marine Biofilms)的物种和功能多样性,内容主要包括生物被膜群落组装机制、物质代谢特点以及基因资源开发。以第一或通讯作者身份在iMeta、Nature Communications(3篇)、Microbiome(2篇)、Environmental Science & Technology 、mSystems (2篇)、Environmental Microbiology(2篇)、Applied and Environmental Microbiology(3篇)、Marine Life Science & Technology(2篇)等主流期刊发表论文30篇;共发表论文88篇,H-index=34。
更多推荐
(▼ 点击跳转)
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
iMeta | 浙大倪艳组MetOrigin实现代谢物溯源和肠道微生物组与代谢组整合分析
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science































 1870
1870

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








