点击蓝字 关注我们
非模式肠道菌基因编辑工具

方法论文
● 期刊: iMeta (IF 23.7)
● 原文链接DOI: https://doi.org/10.1002/imt2.216
●2024年6月23日,康乃尔大学医学院郭春君团队在iMeta在线发表了题为“Genetic manipulations of nonmodel gut microbes”的文章。
● 本研究介绍了一系列详细的基因编辑工具相关的实验步骤,针对非模式肠道革兰氏阴性拟杆菌和革兰氏阳性梭菌建立有效的基因转移方法并开发其基因编辑系统。
● 第一作者:金文兵
● 通讯作者:金文兵(wej4002@med.cornell.edu)、郭春君(cj@guo-group.org)
● 主要单位:康乃尔大学医学院 吉尔·罗伯茨炎症性肠病研究所、康乃尔大学医学院 弗里德曼营养和炎症中心
亮 点
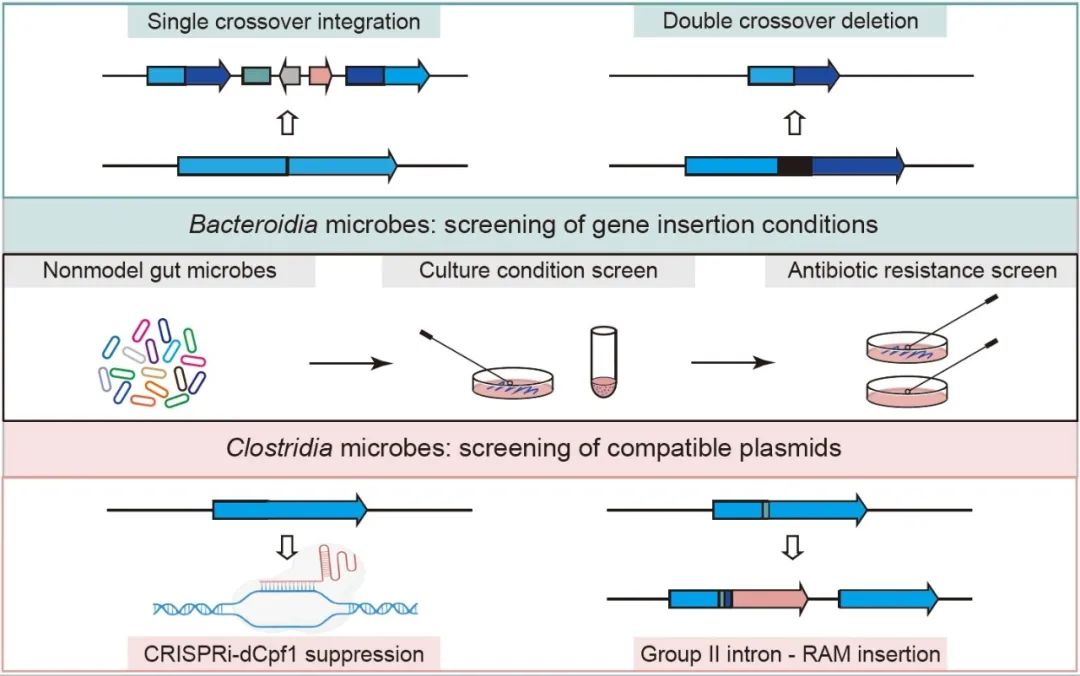
● 此实验方案介绍了一系列详细的基因编辑工具相关的实验步骤,助力研究非模式肠道共生菌所编码基因的功能;
● 此方案可在基因组信息未知的情况下,在非模式肠道拟杆菌和梭菌属中建立基因编辑工具;
● 此方法为人体肠道菌的基因编辑工具提供高通量的筛选和建立方案。
摘 要
健康人与患病人类之间有诸多微生物基因的表达存在显著差异。许多基因由非模式肠道共生菌编码,且这些基因难以进行基因操作,这是阻碍我们研究这些基因如何影响宿主生物学/疾病的分子机制的瓶颈所在。建立有效的基因转移方法并开发其基因编辑系统,将助力我们研究非模式肠道共生菌影响宿主生理的分子机制。本文介绍了一系列详细的基因编辑工具相关的实验步骤,针对非模式肠道革兰氏阴性拟杆菌和革兰氏阳性梭菌建立有效的基因转移方法并开发其基因编辑系统。通过靶向细菌16S rRNA或扩增梭菌可兼容的复制起点,结合CRISPR基因编辑系统,此方案可在基因组信息未知的情况下,在非模式肠道拟杆菌和梭菌属中建立基因编辑工具。此高效且通用的策略将助力肠道共生菌影响宿主生理功能的机制研究。
视频解读
Bilibili:https://www.bilibili.com/video/BV1eE421A7FQ/
Youtube:https://youtu.be/1Ez1EgIhGGQ
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
肠道微生物群通过多种方式影响人体生理功能。多组学研究揭示了许多微生物基因在健康人与患病人类之间的表达显著不同。然而,揭示微生物基因与宿主生物学相互作用的分子机制仍然十分具有挑战性,主要是因为缺乏针对这些与疾病相关的微生物及其代谢基因的基因编辑工具。
开发非模式肠道微生物的基因编辑工具很有必要,原因有二:1)先前的研究表明宿主疾病与微生物基因显著相关。这些基因主要在无法进行基因操作的非模式肠道微生物中表达。建立基因编辑工具将是调控这些基因在宿主体内表达的第一步,并进一步帮助研究这些基因对人类疾病的影响。2)人体生理功能受到肠道微生物群调控,但关于哪些肠道微生物和基因发挥重要作用的知识仍然大多未被研究。基因编辑工具将促进肠道微生物群与宿主之间生理相互作用的功能研究。
在此,我们报道了一系列针对非模式肠道拟杆菌和梭菌的详细的基因编辑工具相关的实验步骤。通过靶向细菌16S rRNA或扩增梭菌可兼容的复制起点,结合CRISPR基因编辑系统,此方案可在基因组信息未知的情况下,建立有效的基因转移方法并开发基因编辑系统。
基因编辑工具
针对拟杆菌建立基因转移方法并开发基因编辑系统
对于拟杆菌,我们利用大肠杆菌(S17)接合转移的方法将外源DNA转移到受体菌中,该方法已被证明在某些拟杆菌和梭菌中可有效使用。我们首先在琼脂平板和液体培养基中筛选菌株的培养条件然后对这些微生物进行一系列抗生素测试,以便:1)找到它们易感的抗生素,从而可以将其抗性基因用作通用选择标记;2)确定受体微生物对其具有抗性而供体大肠杆菌没有抗性的抗生素,从而在接合转移后使用该抗生素抑制大肠杆菌的生长。
拟杆菌是革兰氏阴性专性厌氧菌,能够摄取外源DNA并具有高效的同源重组能力。为了高效建立一种具有普适性的用于拟杆菌的基因编辑工具,我们选择了被广泛用于评估微生物群多样性和构建细菌系统发育树的16S rRNA基因作为通用目标基因。为此,我们将与拟杆菌的16S rRNA基因具有高度同源性的序列合成作为通用16s序列(chi-16s),并将该序列组装到一个自杀型质粒上,通过接合转移的方式转移到受体拟杆菌中。经过后续的验证PCR结合PCR产物测序,那些16S rRNA位点被自杀型质粒插入的接合子被确认为可以进行基因编辑(图1A)。
利用此策略,在使用靶向16S rRNA的方法确定非模式肠道微生物Prevotella bivia DSM 20514的基因编辑工具后,我们通过单交换插入的方式敲除了天冬酰胺酶编码基因(ansB,该基因催化天冬酰胺转化为天冬氨酸),并通过验证PCR和质谱检测确认了冬酰胺酶突变株缺失了将天冬酰胺转化为天冬氨酸的能力(图1B,C)。
在使用靶向16S rRNA的方法利用单交换插入确定拟杆菌基因编辑工具的基础上,我们可以开发更高级的基因编辑工具,例如双交换基因敲除系统。例如拟杆菌Bacteroides sp. 1_1_30中建立了靶向16S rRNA的单交换插入系统之后,我们进一步开发了双交基因敲除系统,对其胸苷激酶编码基因(tdk,该基因能磷酸化胸苷和脱氧尿苷)进行敲除,并进行了PCR验证(图1D,E)。

图1. 针对拟杆菌建立基因转移方法并开发基因编辑系统
(A)建立拟杆菌的基因转移方法;(B和C)通过单交换插入的方式对Prevotella bivia DSM 20514的天冬酰胺酶编码基因(ansB)进行基因敲除,(B)PCR验证基因敲除,(C)质谱检测表明天冬酰胺酶突变株缺失将天冬酰胺转化为天冬氨酸的能力;(D和E)通过双交换敲除的方式对Bacteroides sp. 1_1_30的胸苷激酶编码基因(tdk)进行基因敲除,(D)示意图展示通过双交换敲除胸苷激酶,(E)PCR验证基因敲除。
针对梭菌建立基因转移方法
对于非模式肠道梭菌,我们首先在琼脂平板和液体培养基中筛选菌株的培养条件,然后对这些微生物进行一系列抗生素测试,并且也利用大肠杆菌(CA434或HB101/pRK24)接合转移的方法将外源DNA转移到受体菌中。
相比于拟杆菌,革兰氏阳性梭菌更难进行基因编辑,原因有二:1)难以将外源DNA转移进入梭菌中。常用的大肠杆菌接合转移的方法会将含有受体菌可兼容的复制起点的质粒转移到受体菌,该复制起点使得外源质粒可在受体菌中自我复制。2)梭菌的同源重组效率非常低。
此前的研究中,人们针对梭菌采取过二型内含子插入系统或者CRISPR-Cas9介导基因组双链断裂以促进同源重组的筛选的基因编辑系统。这两种基因编辑系统都需要与受体菌可兼容的复制起点组装,以便在受体菌株中稳定自我复制。因此,寻找梭菌可兼容的复制起点、建立稳定的外源基因转移方法是开发梭菌基因编辑工具关键的第一步。为此,我们将复制起点从4种扩增到9种,以助于筛选梭菌可兼容的复制起点,并且发展了一种混合接合转移的方法,以实现梭菌的外源基因转移方法大规模筛选。
在混合接合转移中,我们将9个含有不同复制起点的质粒分成三组与受体菌进行接合转移操作,随后对长出来的结合子进行PCR验证和基因测序,以确认每组中具体含有哪种复制起点的质粒可被转移进入受体菌。在此之后,该质粒将与受体菌进行第二轮的一对一接合转移,再次确认该质粒可成功被转移至受体菌中。

图2. 针对梭菌建立基因转移方法
针对梭菌开发CRISPRi-dCpf1基因编辑系统
在建立了针对梭菌的外源基因转移方法之后,下一步就是要开发可用的基因编辑系统。此前,由CRISPR-Cas9介导基因组切割和CRISPR-dCas9介导基因组干扰的基因组编辑系统已经被用于针对产孢梭菌和艰难梭菌的基因编辑。但总体而言,由于梭菌的同源重组效率非常低,CRISPR-Cas9介导的基因组切割对受体菌而言通常是致命的。花费大量精力优化各种接合转移参数得到的产孢梭菌中CRISPR-Cas9系统的最佳适用条件,通常也不适用于其他梭菌。
对此,我们针对梭菌开发了CRISPRi-dCpf1基因编辑系统,CRISPRi-dCpf1系统干扰目标基因的转录而非介导的基因组切割,因此相比于CRISPR-Cas9和CRISPR-Cpf1系统具有更小的毒性(基因组断裂引起的细菌死亡),我们在实验过程中发现也的确发现了这一现象。结合CRISPRi-dCpf1系统,我们选取lacZα作为报告基因,通过在受体菌中利用CRISPRi-dCpf1系统对lacZα基因转录抑制的表征来在受体菌基因组信息未知的情况下建立基因编辑系统(图3)。
此CRISPRi-dCpf1基因编辑系统可直应接用于梭菌基因组上的其他目标基因。将靶向目标基因启动子序列和编码序列的向导 RNA基因位点引入含有CRISPRi-dCpf1系统和相应梭菌可兼容复制起点的质粒中。该质粒被转化到供体大肠杆菌CA434(或HB101/pRK24)中,并通过接合转移引入到受体菌中,通过抗生素筛选确定含有CRISPRi-dCpf1质粒的接合子,并通过目标基因的qPCR或质谱检测代谢物等手段验证基因调控。

图3. 针对梭菌开发CRISPRi-dCpf1基因编辑系统
(A)含有CRISPRi-dCpf1基因编辑系统以及报告基因lacZα的pGM-ABCL - pGM-IBCL质粒示意图;(B)由向导 RNA启动子(蓝色背景)、报告基因lacZα启动子(红色背景)、报告基因lacZα编码序列(绿色背景)以及报告基因lacZα终止子(棕色背景)所组成的lacZα基因位点,灰色背景分别是限制性内切酶SbfI和NotI的酶切位点;(C)含有CRISPRi-dCpf1基因编辑系统、报告基因lacZα以及向导 RNA的pGM-ABCF - pGM-IBCF质粒示意图;(D)由三个直接重复序列(绿色背景)、两个向导 RNA序列(蓝色背景)以及终止子序列(红色背景)所组成的向导 RNA基因位点,灰色背景是同源臂序列;(E和F)含有可兼容复制起点的pGM-ABCL - pGM-IBCL质粒(pGM-xBCL,对照组)和pGM-ABCF - pGM-IBCF质粒(pGM-xBCF,实验组)被转移到受体梭菌中,由定量聚合酶链式反应(qPCR)测定报告基因lacZα的表达水平。
针对梭菌开发二型内含子基因编辑系统
除了CRISPRi-dCpf1基因编辑系统,我们还开发了针对梭菌的二型内含子基因编辑系统。二型内含子系统使逆转录转座激活标签(RAM)得以插入目标基因,避免引起类似于CRISPR-Cas9系统所介导的基因组断裂,因此也具有较低的细菌毒性。
与拟杆菌的目标基因类似,我们选择梭菌中具有高度同源性16S rRNA基因作为通用目标基因,用以在受体菌基因组信息未知的情况下开发其二型内含子基因编辑系统。为此,我们将受体梭菌的16S rRNA基因进行比对,找出其中的保守序列,并利用开源ClosTron设计网站(http://www.clostron. com/clostron2.php)选择二型内含子系统可插入的保守位点,进而设计通用的靶向该保守位点的二型内含子插入序列(16s-tron)。随后将该靶向16S rRNA基因的二型内含子插入序列(16s-tron)与受体菌可兼容的复制起点以及适用的逆转录转座激活标签组装成一系列质粒(pGM-ACAQ - pGM-ICAQ,pGM-ACBQ - pGM-ICBQ,图4B,C)。这些质粒被转化到供体大肠杆菌CA434(或HB101/pRK24)中,并通过接合转移引入到受体菌中,通过第一轮质粒自身抗生素筛选确定含有二型内含子系统质粒的接合子,以及第二轮逆转录转座激活标签的抗生素筛选来确定逆转录转座激活标签插入到基因组目标基因位点的突变体,并通过后续目标基因的PCR验证或质谱检测代谢物等手段验证基因插入。
此二型内含子基因编辑系统也可直接应用于梭菌基因组上的其他目标基因。针对目标基因,利用ClosTron设计网站(https://clostron.com/intron-design-tool)设计二型内含子插入序列,将该序列与受体菌可兼容的复制起点以及适用的逆转录转座激活标签组装成可用的二型内含子质粒,即可按上述靶向16S rRNA的方法进行目标基因的插入失活。
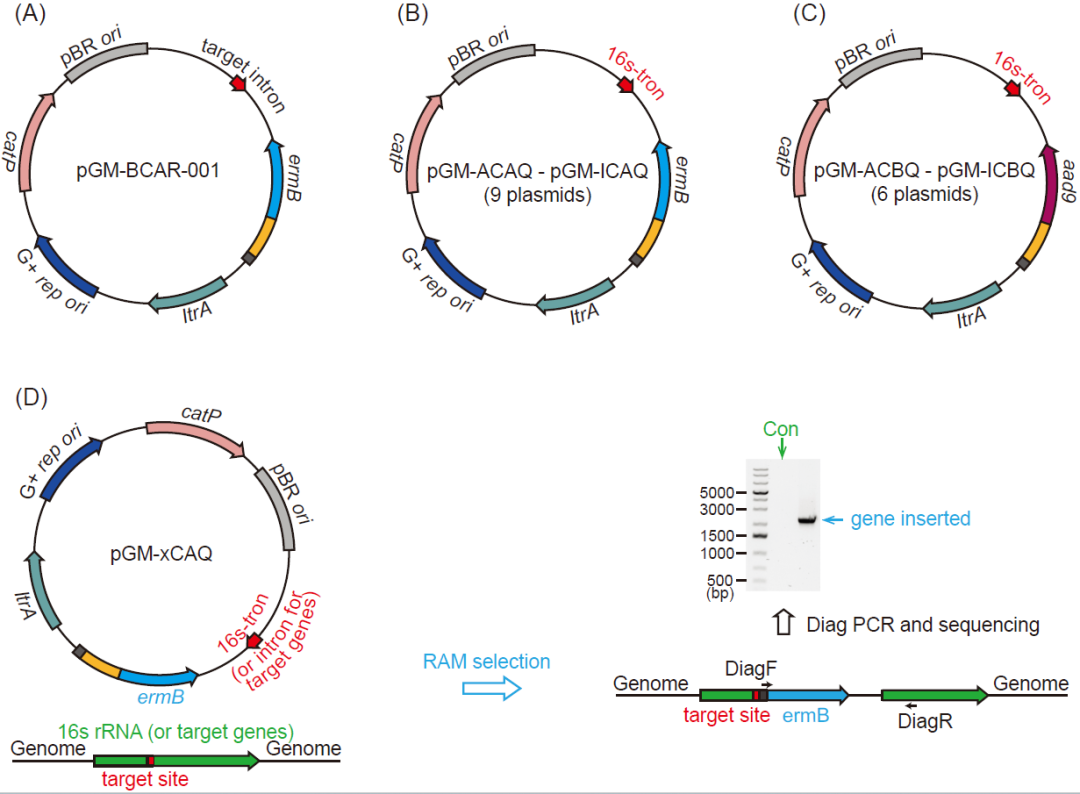
图4. 针对梭菌开发二型内含子基因编辑系统
(A)二型内含子起始质粒pGM-BCAR-001的质粒示意图;(B)含有靶向16S rRNA基因的二型内含子和以catP为逆转录转座激活标签的pGM-ACAQ - pGM-ICAQ质粒示意图;(C)含有靶向16S rRNA基因的二型内含子和以aad9为逆转录转座激活标签的pGM-ACBQ - pGM-ICBQ质粒示意图;(D)确认二型内含子系统逆转录转座激活标签插入目标基因(16S rRNA)的PCR验证策略。
讨 论
许多肠道微生物的基因与人体疾病密切相关,如炎症性肠病和结肠癌,但在分子水平去解析其作用机制目前而言仍具有很大挑战性,因为大多数肠道共生菌是非模式的且难以进行基因编辑。我们在此介绍的方法提供了一种高效且普适的肠道微生物基因编辑工具筛选流程,旨在助力非模式肠道共生菌基因编辑工具的开发及其对宿主生理功能影响的研究。
肠道菌的16S rRNA基因被广泛用于评估微生物群多样性和构建细菌系统发育树,由于拟杆菌具有较高的同源重组效率,其高度保守的16S rRNA基因可以作为拟杆菌的通用目标基因,通过引入自杀型质粒对其进行插入。同样,16S rRNA基因也可以被选作梭菌的通用目标基因,通过将靶向16S rRNA基因的二型内含子插入序列(16s-tron)与受体菌可兼容的复制起点以及适用的逆转录转座激活标签组装整合成可用的二型内含子质粒,对通用目标基因进行插入。此外, CRISPRi-dCpf1系统结合报告基因lacZα,也帮助在梭菌中建立基因编辑系统。这三种策略都可以在受体菌基因组信息未知的情况下,帮助我们时间目标非模式肠道菌基因编辑工具的筛选和建立。
我们在此提供的实验方案有以下三个显著特点。其一,通过以16S rRNA为通用的目标基因以及结合CRISPRi-dCpf1系统和报告基因lacZα,我们可以在未获取菌株全基因组信息的情况下构建其的基因编辑工具。其二,无需调整和尝试,此方案可以在几周内为多种非模式拟杆菌和梭菌建立基因编辑工具。其三,此方案所构建的pGM质粒是模块化的,可以根据具体需求切换、组装不同的遗传操作元件。这些特点赋予了此策略可作为人体肠道微生物基因编辑系统高通量筛选平台的潜力。
尽管具有这些特点,我们的策略仍有局限性。首先,通过单交换插入的方式对拟杆菌进行基因失活的方式有时候只会导致其蛋白质的部分功能丧失,而且当目标基因的大小太小时,单交换整合策略效果不佳。在这些情况下,双交换插入进行基因敲除的方式可以作为备用方案,利用目标基因上下游的同源臂进行一轮单交换插入,随后进行双交换丢失质粒骨架以获得预期的敲除效果。换言之,我们针对拟杆菌的单交换插入筛选可以为开发更高级的基因编辑工具(例如双交换插入基因敲除系统)铺平道路。其次,CRISPRi-dCpf1基因编辑系统有脱靶效应,可能需要测试多个向导RNA以达到理想的抑制效果。多向导RNA是我们当前双向导RNA的扩展版本,可以帮助改善这一不足。在一个质粒中同时引入多个向导RNA,如4个向导RNA,可以帮助减少脱靶效应并增强CRISPRi-dCpf1系统的抑制效率。
尽管上述策略都旨在大规模筛选和建立拟杆菌和梭菌菌株的基因编辑系统,我们的策略也适用于其他门类的微生物,其可行性已被多项研究验证。例如,使用自杀型质粒的单交换插入策略在其他革兰氏阴性肠道微生物中效果良好。我们已经证明,靶向菌株特异性16S rRNA基因的自杀质粒可以通过单交换插入的方式整合到其他革兰氏阴性肠道微生物(如Fusobacterium gastrosuis DSM 101753, Fusobacterium nucleatum ATCC 25751, ATCC 10953, ATCC 23726, Klebsiella oxytoca DSM 29614, DSM 5175, DSM 7342, Proteus mirabilis ATCC 35659 以及 Proteus vulgaris DSM 3265)的基因组16s rRNA中。此外,在筛选可兼容复制起点的质粒转移方法之后,通过结合CRISPRi-dCpf1系统和lacZα报告基因,我们在其他革兰氏阳性肠道微生物(如Bifidobacterium catenulatum DSM 16992和多株粪肠球菌)中也建立起了有效的基因编辑工具。此高效且普适的策略将从以下几个侧面促进我们对肠道微生物的研究:建立具有重要生理意义的非模式肠道微生物的基因编辑工具;精确调控肠道微生物的功能基因,以评估其对宿主生理功能的影响;揭示肠道微生物所产生代谢产物的生物合成过程及其对宿主的影响;激发更多有效的基因编辑工具以求在基因层面研究肠道菌-宿主相互作用的分子机制。
数据可用性:
此方法未产生新的数据和脚本。补充材料(图表、表格、图形摘要、幻灯片、视频及中文翻译版)可在在线DOI或iMeta Science网站http://www.imeta.science/中找到。
引文格式:
Wen-Bing Jin and Chun-Jun Guo. 2024. Genetic manipulations of nonmodel gut microbes. iMeta e216. https://doi.org/10.1002/imt2.216
作者简介

金文兵(第一/通讯作者)
● 美国康奈尔大学威尔康奈尔医学院博士后。
● 主要研究方向为利用基因编辑技术改造肠道菌群,对肠道微生物-代谢产物/药物-宿主之间的相互作用进行分子机制层面的深入探索。已以一作或通讯在Cell、Nat. Commun、Nat. Prod. Rep.和iMeta等期刊发表多篇学术论文。拟于2024年8月入职厦门大学生科院,初创课题组处于起步发展阶段,诚邀博士后共谋发展,有意者请联系 kimwenbing@outlook.com,或访问https://life.xmu.edu.cn/info/1070/14211.htm。招聘长期有效,欢迎联系。

郭春君(通讯作者)
● 美国康奈尔大学威尔康奈尔医学院助理教授。
● 主要研究方向为利用基因编辑技术改造肠道菌群,研究其释放小分子的生物活性及对宿主生理功能的影响,已在Cell、Nature、Science、Cell Host & Microbe、Nat. Commun、JACS、PNAS和iMeta等期刊发表多篇学术论文。课题组长期招聘博士后和访问学者,有意者请将个人简历、推荐人信息和研究兴趣简介发送至 cj@guo-group.org或者chg4001@med.cornell.edu,或访问http://www.guo-group.org/。
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
3卷3期
3卷3期封底
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF 23.7)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、50万用户的社交媒体宣传等。2022年2月正式创刊发行!目前期刊已经被ESCI、PubMed、Scopus等数据库收录。
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science








































 2493
2493

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








