先到PrognoScan: A new database for meta-analysis of the prognostic value of genes.找到想做的基因和对应的疾病。比如我要找stat3和breast cancer

详细页面已经有分析好的结果和图片。但是图片不能修改也非高清,最好自己画。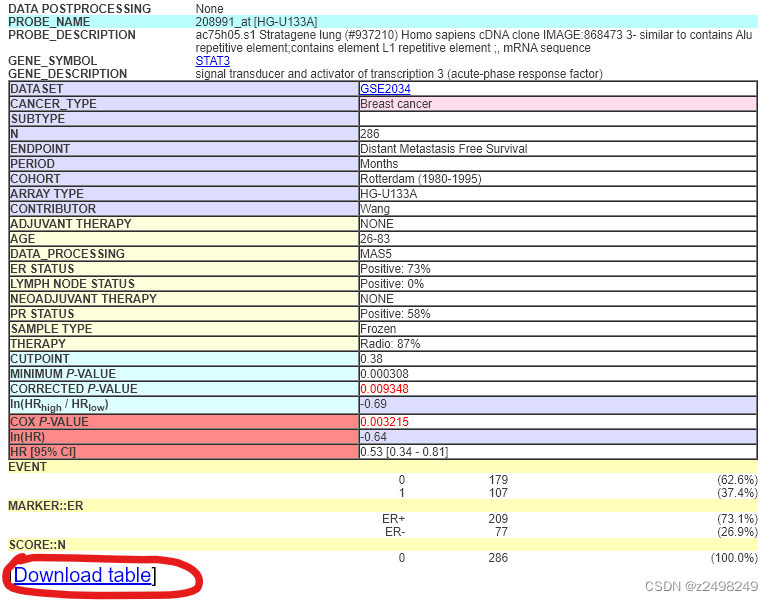
点击红圈进入详细页面,下载数据。
之后用R语言加载下好的数据进行分析
library("survival")
BiocManager::install("DO.db")
library("survminer")
library("clusterProfiler")
library("DO.db")
rm(list=ls())
data <- read.csv("stat3.tsv",header = T, row.names = 1, ,sep = "\t")
cut_off_info <- surv_cutpoint(data, time = "TIME", event = "EVENT", variables = c("EXPRESSION"), minprop = 0.1, progressbar = TRUE)
cut_off <- cut_off_info$cutpoint$cutpoint
data$exp <- ifelse(data$EXPRESSION>cut_off,"High expression","Low expression")
ggsurvplot(
survfit(Surv(TIME, EVENT)~exp, data=data),
conf.int=F,
pval=TRUE,
data=data,
xlab = "Time (years)"
)
需要注意的是cutoff的选择,一般情况下回选择mean或者median作为cutoff。但是有时mean或者median的cutoff分析出是没有意义的,所以我用surv_cutpoint函数选取了cutoff。上面提到的PrognoScan里的结果,高低表达的数量相差很多,应该也不是用mean或者median做cutoff。
另外,我们用这种分析结果作为一个引子,而非直接证据或结果。如果是要作为主要论证依据,建议自己整理数据分析。
参考:
https://github.com/zechuanchen/NGS-/blob/master/GEO%E7%94%9F%E5%AD%98%E6%9B%B2%E7%BA%BF%E6%95%B0%E6%8D%AE%E5%88%86%E6%9E%90.mdhttps://www.jingege.wang/2022/09/14/prognoscan-geo%E6%95%B0%E6%8D%AE%E9%9B%86%E7%94%9F%E5%AD%98%E5%88%86%E6%9E%90%E5%B7%A5%E5%85%B7/






















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








