CHARMM-GUI 构建蛋白质的脂质双分子层
进入 CHARMM-GUI 在线网站
登陆网站后选择左侧边栏的 Input Generator

进入 Input Generator 后选择左侧边栏 Membrane Builder 中的 Bilayer Builder

在 Bilayer Builder 中上传需要进行加膜的蛋白质 pdb 文件,然后点击页面右下角 Next Step: Select Model/Chain

PDB Info: Model/Chain Selection Options
在当前页面选择需要的氨基酸链和起始氨基酸序号,然后点击页面右下角 Next Step: Manipulate PDB

PDB Info: PDB Manipulation Options
在 System pH 选项中设置整个体系的 pH ;在 Protonation state 选项中设置不同质子化状态的氨基酸残基。(有些体系还需要在 Disulfide bonds 选项中设置二硫键状态)
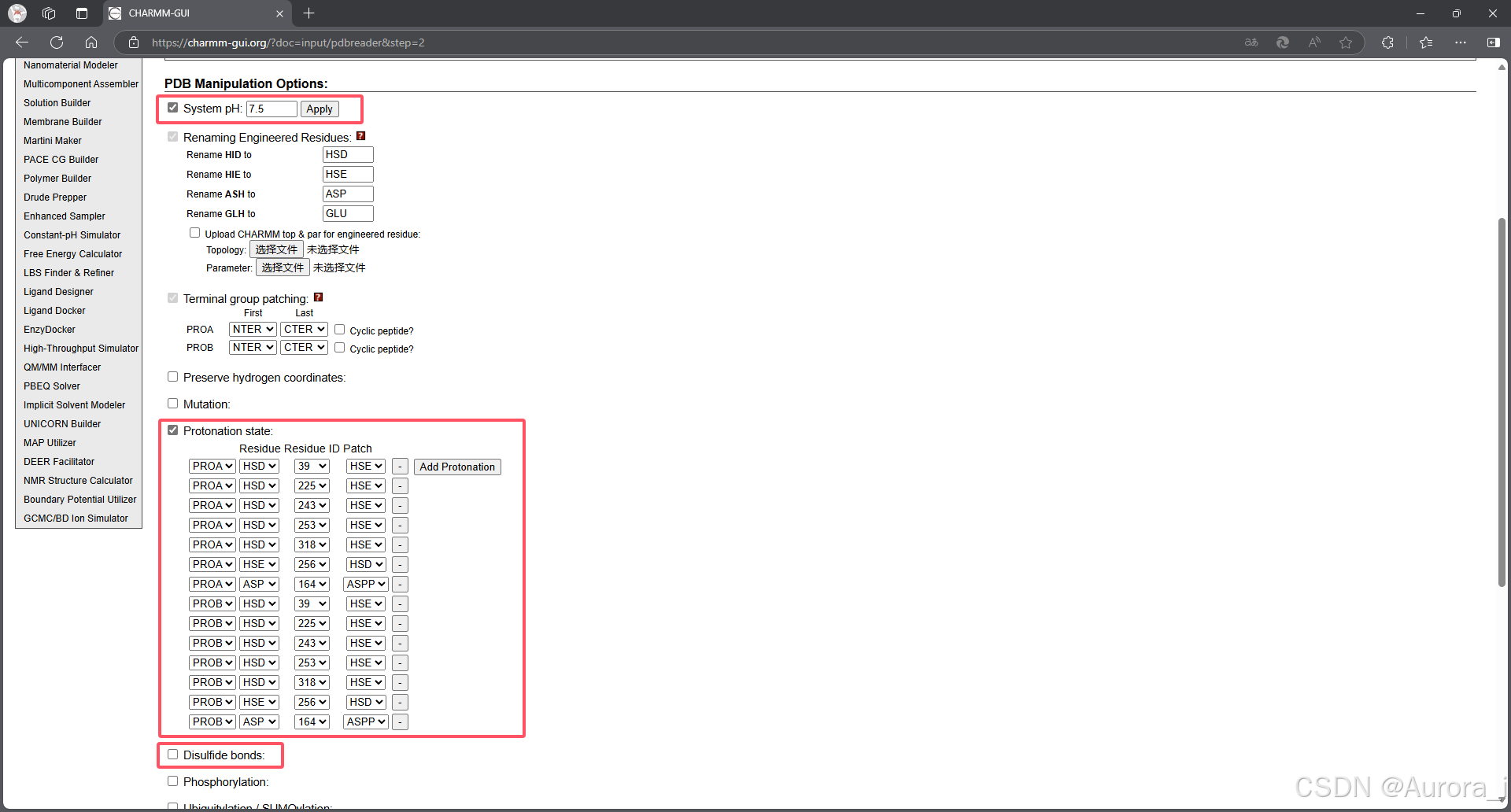
然后点击页面右下角 Next Step: Generate PDB and Orient Molecule

STEP 1: Generate PDB and Orient Molecule
选择 Align a Vector (Two Atoms) Along Z 选项,然后在下方选择两个氨基酸残基作为基准来定位后续膜生成的位置。(此方法适用于运行 Run PPM 2.0 之后膜的预生成位置不准的情况)

然后点击页面右下角 Next Step: Calculate Cross-Sectional Area

STEP 2: Calculate Cross-Sectional Area
首先点击下图红框中的 view structure 查看膜的预生成位置


预生成位置正确后,在 Length of X and Y 中设置大小,然后选择 POPC 膜,最后点击 Show the system info

然后点击页面右下角 Next Step: Determine the System Size

STEP 3: Determine the System Size
首先点击下图红框中的 view structure 查看膜的生成位置


在 Basic Ion Types 中选择 NaCl 然后点击 Add Simple Ion Type
下一步点击 Calculate Solvent Composition
最后点击页面右下角 Next Step: Build Components

STEP 4: Check lipid pentration
点击下图红框中的 view structure 查看膜的生成情况


无问题后点击页面右下角 Next Step: Assemble Components

接着点击页面右下角 Next Step: Assemble Components

STEP 5: Determined System Size
点击下图红框中的 view structure 查看膜的组装情况


在 Force Field Options 中选择 AMBER 力场,对蛋白质设置 FF14SB 力场其他保持默认参数,然后勾选 Hydrogen mass repartitioning
在 Input Generation Options 中选择 AMBER
在 Equilibration Options 中设置 Temperature 为 300 K
接着点击页面右下角 Next Step: Generate Equilibration and Dynamics Inputs

STEP 6: Download Result
已经处理完成,点击 download.tgz 下载压缩包即可

下载好的压缩包进行解压后得到文件如下:

至此,利用 CHARMM-GUI 对蛋白质进行加膜的操作完成!

























 959
959

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








