用pymol打开PI3K分子:点击File—open打开PI3K PDB格式文件

删除水分子:在pymol>框中输入:remove solvent,除蛋白结构中的小分子配体:remove organic,Select选择PI3K,点击save, 储存为PI3K_REV.pdb.


用AutoDockTools打开“PI3K_REV.pdb”

加氢:Edit→Hydrogens→Add,点击OK,后覆盖保存为“PI3K_REV.pdb”。



设置PI3K为大分子蛋白,点击Grid→Macromolecule→Choose,保存成“PI3K_REV.pdbqt”。


处理小分子
删掉PI3K蛋白质,edit->delete,导入小分子,与蛋白质导入相同的步骤,同样的进行加氢处理,ligand->input->choose,选择小分子作为ligand
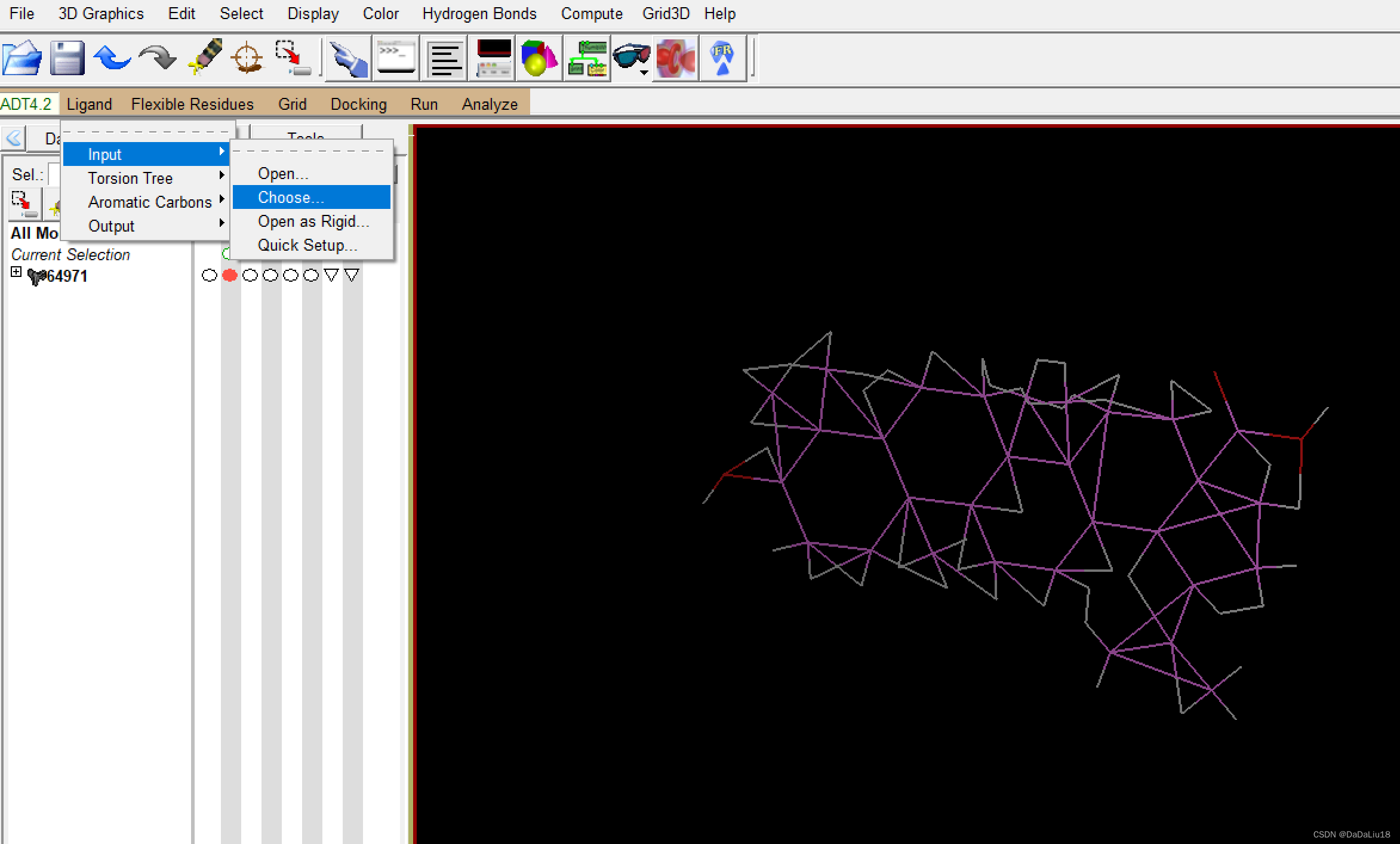

同时对扭转键进行检测。ligand->torsion tree->detect root,得到如下结果

ligand->torsion tree->choose torsions>Done

红色的是不可以被扭转的,绿色的是可以扭转的
输出小分子,ligand->output->save as 64971.pdbqt,得到小分子的pdbqt格式的文件。























 7938
7938

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








