介绍
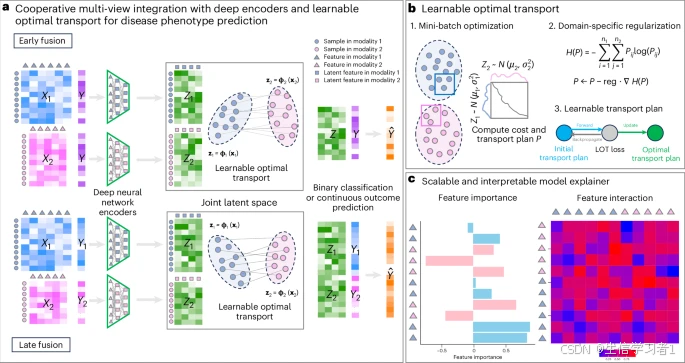
单组学方法往往只能对复杂的生物系统提供有限的视角,而多组学整合则能够通过整合各种数据视角来实现更全面的理解。然而,整合不同类型的异质数据以及解析生物特征(包括同一视角和不同视角之间)之间的复杂关系仍然是一个重大挑战。在此,为应对这些挑战,我们引入了 COSIME(具有可扩展性和可解释模型解释器的协同多视角整合)。COSIME 将可学习的最优传输算法的反向传播应用于深度神经网络,从而能够从多个视角学习潜在特征以预测疾病表型。它还结合了蒙特卡罗抽样,以便能够对同一视角和不同视角内的特征重要性和两两特征相互作用进行可解释的评估。我们将 COSIME 应用于模拟和真实世界的数据集——包括单细胞转录组学、空间转录组学、表观基因组学和代谢组学——以预测与阿尔茨海默病相关的表型。对现有方法的基准测试表明,COSIME 提高了预测的准确性,并具有可解释性。例如,它表明与阿尔茨海默病相关的星形胶质细胞和小胶质细胞基因之间的协同作用更有可能集中在中颞回的边缘区域。最后,COSIME 还作为一个开源工具公开可用。
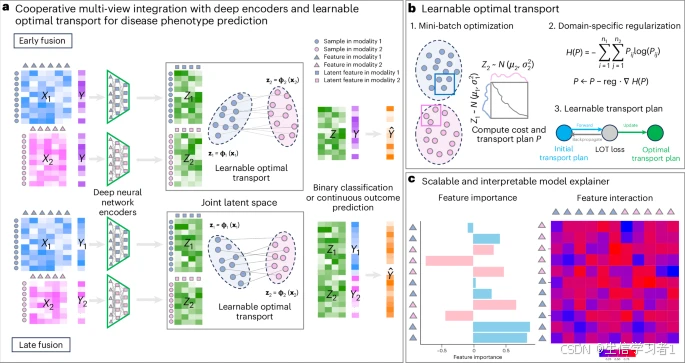
单组学方法对于复杂生物系统的研究作用有限,因为每一个分子层面(例如基因组学、转录组学或代谢组学)都是与其他层面相互协作发挥作用的。多视角数据整合通过整合多个层面来识别新的生物标志物、理解疾病机制并改进表型预测1,2。机器学习能够高效地分析大规模多视角数据,并模拟影响表型的跨视角相互作用。
针对多视角数据整合已开发出多种方法。协同学习(CL)3 是一种有监督学习方法,它将平方误差损失与一种鼓励不同视角下特征相似性的一致性惩罚相结合。该惩罚强度是固定的且需手动调整,这限制了动态适应和跨视角交互的端到端学习。利用潜在成分进行生物标志物发现的数据整合分析(DIABLO)4 通过有监督的特征选择来识别不同组学层面上共有的结构,但假定它们与结果的关联具有同质性,而在异质数据集中这可能并不成立。多组学因子分析 v.2(MOFA+)5 是一种无监督因子分析方法,它对不同视角下的潜在线性结构进行建模,但缺乏交互分析或解释预测特征的机制。此外,这些模型通常假定线性关系,并且不利用深度学习或最优传输(OT)。
可解释性是另一个主要的挑战。SHAP(沙普利可加性解释)6 可以量化特征的解释度,但对于复杂的模型计算成本较高,并且只能为基于树的模型分配交互贡献。LIME7 为单个预测提供了局部可解释性,但缺乏全局解释,并且也不考虑特征之间的交互作用。
为解决这些局限性,我们引入了具有可扩展性和可解释性模型解释器的协同多视图整合方法(COSIME)。COSIME 由两个部分组成:(1)一个整合组件,它使用深度神经编码器和可学习的最优传输算法(LOT)在联合潜在空间中对视图进行对齐,并预测疾病表型;(2)一个解释组件,它使用肖普利值和肖普利-泰勒指数来估计特征重要性(FI)和成对相互作用。我们在模拟和真实世界的数据集上对 COSIME 进行了评估,以使用转录组学、代谢组学、表观基因组学和空间转录组学来预测阿尔茨海默病(AD)表型,并展示了其强大的预测性能、灵活性和可解释性。
代码
https://github.com/daifengwanglab/COSIME
参考
- Cooperative multi-view integration with a scalable and interpretable model explainer
- https://github.com/daifengwanglab/COSIME






















 4万+
4万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










