文章目录
nnUNet自提出以来一直被各种模型作为其baseline,其泛化性很强(目前已被用在多个不同医学分割任务中,均取得较好的结果),究其原因我觉得很大程度在于其数据预处理,以及参数的设置。
目前该框架已经被封装的很完善,只需要将自己的数据按要求整理好在对应的文件夹中,直接通过终端命令便可开始训练模型,整个数据预处理等操作都能够通过命令行实现。下面开始介绍模型的安装和数据前期的整理。
具体可见documentation文件夹,里面详细写了各个阶段的命令以及需要注意的问题,非常全

一、安装nnUNet
方法一:直接安装
pip install nnunet #通过命令行直接安装nnunet包
pip list # 查看安装了哪些包
方法二:下载仓库后安装
nnUNet的官方Github仓库
或在此下载:已下载好的压缩包
解压安装包后,cd到该路径,并运行:
pip install setup.py
二、建立文件夹
首先需要建立三个文件夹用来存放处理后的数据以及训练过程中生成的文件

“nnUNet_preprocessed”文件夹:存放预处理后的数据文件
“nnUNet_raw_data_base"文件夹:按照一定格式分布的原始数据
“nnUNet_trained_models”文件夹:存放训练阶段的参数文件等
三、设置环境变量
设置环境变量对nnUNet来讲很重要,nnU-Net需要知道你打算将原始数据,预处理数据和训练好的模型保存在何处。相当于全局知道数据的路径,这样在终端就可以执行运行命令行而不需要每次训练都指定到数据存放的路径在哪里。
在终端打开环境配准文件/bashrc
cd # 进入根目录
sudo gedit .bashrc # 编辑环境文件
将以下行放入/bashrc文件最后:
export nnUNet_raw_data_base=“/文件路径(需改)/nnUNet_raw_data_base”
export nnUNet_preprocessed=“/文件路径(需改)/nnUNet_preprocessed”
export RESULTS_FOLDER=“/文件路径(需改)/nnUNet_trained_models”
成功设置后,更新文件后可以在终端输入echo $RESULTS_FOLDER 来验证路径RESULTS_FOLDER设置正确,如果成功会自动打印处对应的路径打印出正确的文件夹
source .bashrc # 更新环境文件
echo $RESULTS_FOLDER

四、原始数据文件的整理
按照要求,我们需要对原始数据文件进行整理以符合需要
(1)建立子文件夹
首先在nnUNet_raw_data_base文件夹里创建一个文件夹nnUNet_raw_data,该文件夹存放不同任务的数据,如图
Task001_BrainTumor文件夹对应脑部肿瘤任务的数据;
Task002_Heart文件夹对应心脏分割任务的数据;等等

(2)建立Task文件夹
文件命名必须按照 “TaskID_任务名称” 格式, 其中 “ID” 是必须是三位数的整数,001而不是1。此处要按要求命名,因为后面执行命令会涉及到任务的ID号。
如图,我的数据集是鼻咽癌数据,所以我的文件命名为 Task001_npc
(3)训练测试数据的划分
在Task文件夹下,一般包含三个文件夹:
imagesTr: 训练图像文件夹。
labelTr: 训练集标签文件夹。
dataset.json : 包含数据集的元数据的JSON文件, 如任务名字,模态,标签含义,训练集包含的图像地址等
但是如果还有测试数据,则继续加在此目录下,若无则不同添加:
imagesTs(可选): 测试图像文件夹
labelTr(可选): 测试集标签文件夹
imagesTr文件夹下数据的存放格式
如图,训练测试文件夹的数据要按照case_patientID_modeID.nii.gz 格式来存放
其中case 是任务名,只要保持一致就行,命何名无关紧要
patientID是患者的ID号,必须保持相同的位数,如图中保存的位数为3,则1要写成001
modeID是数据的模态ID号,同样要保持相同的位数,如图中保存的位数为4,则假设CT模态为代号0要写成0000代表CT;假设MRI_T1模态为代号1要写成0001代表T1

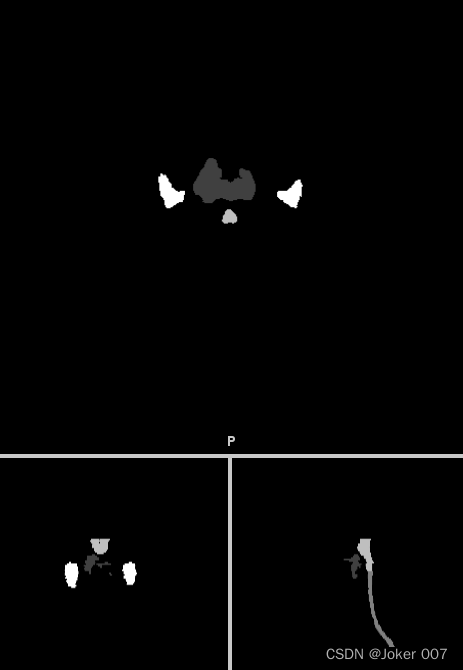
labelTr文件夹下数据的存放格式
标签文件需要将不同的标签整合在一个数组中,如图。不同的标签用不同的one-hot值表示。
标签文件的命名格式为case_patientID.nii.gz
json文件
json文件中是一个字典,也就是字典键值对应的你要修改成自己需要的形式

注意:
① labels内的值:根据分类任务进行修改;
② training内文件名及其数目:训练集及标签的数目、文件名都要对应正确。
(4)文件整理的代码示例
根据上述内容,数据整理需要统一格式。在nnUNet也提供了,许多公开数据集整理成标准格式的代码。
如果训练的是公开数据集,并且nnUNet里面有相关的处理文件可以直接运行。

下面,介绍一下我自己的数据整理成标准格式的代码。
我自己数据文件的原始格式为:

整理后的格式为:

from collections import OrderedDict
from batchgenerators.utilities.file_and_folder_operations import *
import shutil
import os
join = os.path.join
isdir = os.path.isdir
isfile = os.path.isfile
listdir = os.listdir
makedirs = maybe_mkdir_p
os_split_path = os.path.split
if __name__ == "__main__":
"""
REMEMBER TO CONVERT LABELS BACK TO BRATS CONVENTION AFTER PREDICTION!
"""
task_name = "Task001_npc"
data_dir = r"/home/amax/Task/nnUnet_data/ori_data/data" # 原始数据存放路径
label_dir = r"/home/amax/Task/nnUnet_data/ori_data/labels" # 原始标签存路径
nnUNet_raw_data = r'/home/amax/Task/nnUnet_data/nnUNet_raw_data_base/nnUNet_raw_data' #整理后数据存放路径
target_base = join(nnUNet_raw_data, task_name)
target_imagesTr = join(target_base, "imagesTr")
target_labelsTr = join(target_base, "labelsTr")
maybe_mkdir_p(target_imagesTr)
maybe_mkdir_p(target_labelsTr)
patient_names = []
for patient_name in subdirs(data_dir, join=False):
patdir = join(data_dir, patient_name)
patdir_lab = join(label_dir, patient_name)
patient_names.append(patient_name)
# 不同模态数据的原始路径
ct = join(patdir, "ct_Mask.nii.gz")
t1 = join(patdir, "t1.nii.gz")
t1c = join(patdir, "t1c.nii.gz")
t2 = join(patdir, "t2.nii.gz")
t1dix = join(patdir, "T1DIXONC.nii.gz")
seg = join(patdir_lab, "label.nii.gz")
assert all([
isfile(ct),
isfile(t1),
isfile(t1c),
isfile(t2),
isfile(t1dix),
isfile(seg)
]), "%s" % patient_name
# 将数据按照标准格式命名并复制到指定路径中
shutil.copy(ct, join(target_imagesTr, "NPC_" + patient_name + "_0000.nii.gz"))
shutil.copy(t1, join(target_imagesTr, "NPC_" + patient_name + "_0001.nii.gz"))
shutil.copy(t1c, join(target_imagesTr, "NPC_" + patient_name + "_0002.nii.gz"))
shutil.copy(t2, join(target_imagesTr, "NPC_" + patient_name + "_0003.nii.gz"))
shutil.copy(t1dix, join(target_imagesTr, "NPC_" + patient_name + "_0004.nii.gz"))
shutil.copy(seg, join(target_labelsTr, "NPC_" + patient_name + ".nii.gz"))
# 创建json文件内容
json_dict = OrderedDict()
json_dict['name'] = "Npc_Seg"
json_dict['description'] = "nothing"
json_dict['tensorImageSize'] = "4D"
json_dict['reference'] = "nothing"
json_dict['licence'] = "nothing"
json_dict['release'] = "0.0"
# 不同模态对应的ID
json_dict['modality'] = {
"0": "CT",
"1": "T1",
"2": "T1C",
"3": "T2",
"4": "T1DIXONC"
}
# 不同标签对应的one-hot码
json_dict['labels'] = {
"0": "background",
"1": "GTVnx",
"2": "Spinal_cord",
"3": "Brain_Stem",
"4": "Parotid"
}
json_dict['numTraining'] = len(patient_names)
json_dict['numTest'] = 0
json_dict['training'] = [{'image': "./imagesTr/NPC_%s.nii.gz" % i, "label": "./labelsTr/NPC_%s.nii.gz" % i} for i in
patient_names]
json_dict['test'] = []
# 将字典写入json文件中
save_json(json_dict, join(target_base, "dataset.json"))
五、执行终端命令
(1)数据验证以及数据预处理
在终端执行以下代码,其中Task_ID是任务编码,通过文件命令可以看到我的在此处的任务ID是001(要注意ID号的位数,与文件夹命名的ID号要一致)
nnUNet_plan_and_preprocess -t Task_IDxxx --verify_dataset_integrity
执行完该命令后,会自动对数据文件进行验证(是否符合标准格式);完成后会自动对数据进行交叉验证的划分,以及数据crop等预处理的操作。
此时会在nnUNet_raw_data_base目录生成一个nnUNet_cropped_data文件夹,里面有该Task预处理crop后生成每个patient的npz和pkl文件。


同时在文件夹nnUNet_preprocessed中生成该Task的预处理后数据,包括二维的和三维的数据以及交叉验证做的数据split等

(2)训练
以下命令都是在终端执行的
# 训练命令行的格式(--npz不需要softmax输出时不要加,会占用大量空间)
'''
CONFIGURATION:训练的方案,是2d、3d还是级联
TRAINER_CLASS_NAME:训练的模型选择,即model trainer
TASK_NAME_OR_ID:任务的ID,此处位数无要求也就是说如果Task文件夹命名的ID是‘001’,那么此处只写‘1’即可,写‘001’或者‘01’均会报错
FOLD:选交叉验证的某一折,如五折交叉验证那此处可选0,1,2,3,4
'''
nnUNet_train CONFIGURATION TRAINER_CLASS_NAME TASK_NAME_OR_ID FOLD --npz (additional options)
# 举例:训练数据为2维,model trainer是V2,Task的ID是001,选五折交叉验证的第五折
nnUNet_train 2d nnUNetTrainerV2 1 4
# 不同训练方案的命令行格式
# 2D U-Net
nnUNet_train 2d nnUNetTrainerV2 MyTask_ID FOLD --npz
# 3D full resolution U-Net
nnUNet_train 3d_fullres nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz
# 3D U-Net cascade
# 注意:cascade的3D全分辨率UNet需要事先完成低分辨率UNet的五折。经过培训的模型将被写入RESULTS_FOLDER/nnUNet文件夹。
nnUNet_train 3d_lowres nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz # stage1:3D low resolution U-Net
nnUNet_train 3d_cascade_fullres nnUNetTrainerV2CascadeFullRes TaskXXX_MYTASK FOLD --npz # stage2: 3D full resolution U-Net
如果想更改模型训练的参数,比如epoch等,需要根据命令行选择的model trainer,找到对应的.py文件进行更改。
比如,我运行的训练命令是:
# 单卡训练
# 注意:默认在第一块gpu(索引为0)上进行训练,如果想指定某个gpu,请先执行:export CUDA_VISIBLE_DEVICES=X,X为你指定的gpu索引。再执行下面的命令。
nnUNet_train 2d nnUNetTrainerV2 1 4
# 多卡训练:比如我现在要在0和1两张卡上执行训练
export CUDA_VISIBLE_DEVICES=0,1
nnUNet_train_DP 2d nnUNetTrainerV2_DP 1 4 -gpus 2
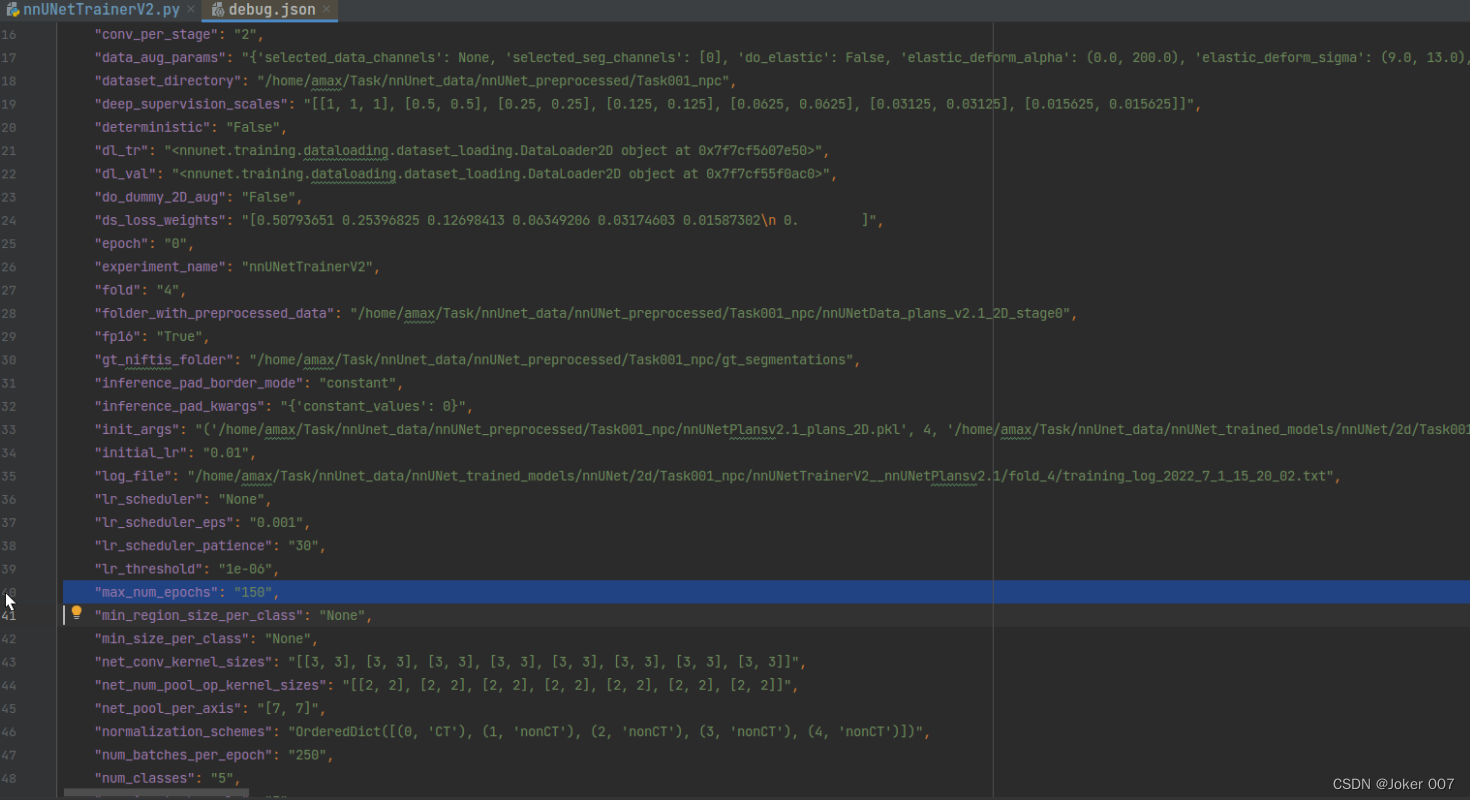
针对单卡训练的情况下,那么我要查看的文件是nnUNetTrainerV2.py, 如图model trainer的py文件都在仓库的nnUNet/nnunet/training/network_training目录中。默认的epoch数是1000,我改成150
 在运行训练命令后,nnUNet_trained_models目录会产生相应的训练文件,并且通过查看训练中产生的debug.json文件便能得到训练阶段各个参数值,可以看到此时训练的epoch数已经变成150了。
在运行训练命令后,nnUNet_trained_models目录会产生相应的训练文件,并且通过查看训练中产生的debug.json文件便能得到训练阶段各个参数值,可以看到此时训练的epoch数已经变成150了。


参数batch_size和patch_size的修改可以参考:
分割模型nnUNet学习日记(三):nnUNet参数batch_size和patch_size的修改方式
在首次执行训练命令时,可能会报错ImportError: /usr/lib/x86_64-linux-gnu/libstdc++.so.6: version `GLIBCXX_3.4.22‘ not found,该错误的解决方案具体可参考:
分割模型nnUNet学习日记(二):常见问题报错解决方案
(3)手动预测
# 预测命令行的格式
'''
INPUT_FOLDER: 测试数据地址
OUTPUT_FOLDER: 分割数据存放地址,即前面的imagesTs文件夹
CONFIGURATION: 训练采用的方案,2d or 3d_fullres or 3d_cascade_fullres等
'''
nnUNet_predict -i INPUT_FOLDER -o OUTPUT_FOLDER -t TASK_NAME_OR_ID -m CONFIGURATION
运行完可能会出现一下警告:
WARNING! Cannot run postprocessing because the postprocessing file is missing. Make sure to run consolidate_folds in the output folder of the model first!
这个问题的解决方案在原github中作者给出了解答 [https://github.com/MIC-DKFZ/nnUNet/issues/410]
即运行(其中,要保证所有的多折交叉验证全部训练完毕)
nnUNet_determine_postprocessing -t TASK_NAME_OR_ID -m CONFIGURATION
参考博客
nnUNet使用指南(一):Ubuntu系统下使用nnUNet对自己的多模态MR数据集训练
nnUNet最舒服的训练教程(让我的奶奶也会用nnUNet(上)








 本文详细介绍nnUNet的安装步骤与数据整理方法,并提供训练与预测的命令行示例,帮助读者快速掌握该医学影像分割框架。
本文详细介绍nnUNet的安装步骤与数据整理方法,并提供训练与预测的命令行示例,帮助读者快速掌握该医学影像分割框架。
















 20万+
20万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








