本研究提供了人类连接组计划基于表面的多模态脑区细分图谱(HCP-MMP v1.0, Glasser et al. 2016)的修改和扩展版本,HCPex。原始图谱包含360个脑皮层区域,本文在HCPex中对其进行了修改,以便更容易地与基于体积(Volume)神经影像软件(例如SPM、FSL和MRIcroGL)一起使用。HCPex也是原始图谱的扩展版本,在此基础上增加了66个皮下区域(每个半球33个),包括杏仁核、丘脑、壳核、尾状核、伏隔核、苍白球、乳头体、隔核和基底核。HCPex使得HCP-MMP v1.0中出色的脑皮层区域细分对基于体积(VOLUME,体素空间)软件的用户(如SPM和FSL)可用,并增加了一些皮层下区域,提供了带标记的人脑冠状视图。本文发表在Brain Structure and Function杂志。(可添加微信号19962074063或18983979082获取原文及补充材料,另思影提供免费文献下载服务,如需要也可添加此微信号入群)
引言
人类连接组计划多模态脑区细分图谱(HCP-MMP)提供了对人类大脑皮层区域的非常有用的细分(Glasser et al. 2016a, b)。图谱是多模态的,因为每个区域都是根据四个标准的组合定义的,包括结构(T1加权/T2加权髓鞘含量和皮层厚度图)、静息态功能连接、基于任务的激活和拓扑地形组织,使用3T MRI扫描仪(Glasser et al. 2016a)。HCP-MMP v1.0图谱包含每个半球180个皮层区域,基于210名参与者的分析。定义的区域包括许多因其功能证据而引起关注的区域,例如顶枕皮层区域LIP、VIP、MT、MST和包括V1、V2、V3、V4等枕叶区域。鉴于HCP-MMP v1.0图谱(Glasser et al. 2016a)提供的细分的实用性,我们在这里提供一个新的扩展版本的图谱,HCPex,目的和理由是通过以体积(Volume)形式提供、并增加66个皮层下区域、提供一个重新排序的版本并提供恢复到原始顺序的选项,使其更加有用,如下所述。我们注意到这个图谱有助于大脑结构和功能的分析,因为在HCP图谱中定义的许多皮层区域在功能上是已知的,而且该图谱帮助新的大脑研究与这些解剖学定义的大脑区域的已知功能相关联。
首先,HCPex以体积(Volume)形式提供,这有助于其与SPM、FSL、FreeSurfer和MRIcroGL等软件的使用,我们为此提供了必要的标签文件。(原始的HCP-MMP v1.0图谱可在fsaverage的表面空间中获得(Glasser et al. 2016a)。之前已经提供了一些转换到体积(Volume)空间的版本(Horn 2016a, b; Coalson et al. 2018)。)我们提供1毫米和2毫米版本的HCPex图谱,并提供用户指南,说明如何使用这些类型的软件安装它。其次,HCPex扩展超出HCP-MMP的360个脑皮层区域,另外包括66个皮层下区域(每个半球33个),目的是使其在许多功能性和结构性连接组学研究中更加实用。第三,HCPex中的脑区域已根据其皮层系统重新排序,因为对一些用户来说,HCP-MMP v1.0图谱的原始标签顺序并不容易遵循。然而,我们确保了两种排序版本都可以使用,通过提供一个转换函数在两个顺序之间转换。第四,为了更好地可视化图谱,我们在本文中为HCPex提供了带有每个HCPex区域标记的大脑冠状视图,以展示这个图谱中定义的426个大脑区域在人脑中的位置。这被证明是对用户非常有帮助的补充。这个HCPex扩展图谱已经在人类海马记忆系统的白质追踪(Huang et al. 2021)、功能性连接性(Ma et al. 2021)和有效连接性(Rolls et al. 2021)分析中证明其用途。
我们指出,像HCP-MMP v1.0提供的基于表面的配准方法比体积(Volume)方法(Glasser et al. 2016b; Coalson et al. 2018)能更好地定位脑皮层区域,如那些常用并在神经影像软件中提供的基于Volume的方法,例如SPM(Friston et al. 2006)(https://www.fil.ion.ucl.ac.uk/spm/)和FSL(Smith et al. 2004; Jenkinson et al. 2012)(https://fsl.fmrib.ox.ac.uk/)。然而,鉴于许多神经影像分析是用像SPM和FSL这样的软件进行的,而且基于体积(Volume)分析使皮下区域可以轻松直接地包含在HCPex的图谱中,这是HCPex的一个目标,因此我们提供了HCPex。同时,当需要比HCPex提供的更好的定位时,我们推荐使用基于表面的版本HCP-MMP v1.0(Glasser et al. 2016a),原因如Coalson et al(2018)所述。我们对HCP-MMP图谱中提供的细分印象深刻(Glasser et al. 2016a),这是我们在包括皮下区域的分析中使用HCPex的原因(Huang et al. 2021; Ma et al. 2021; Rolls et al. 2021),也是我们使HCPex可供他人使用的原因。由于HCPex基于HCP-MMP v1.0,我们建议在发表的研究中使用HCPex时引用Glasser et al(2016a)。我们注意到FreeSurfer(Fischl et al. 1999a, b)(http://surfer.nmr.mgh.harvard.edu/)确实使用基于表面的配准,但只提供有限数量的皮下区域。因此,我们提供HCPex的目的是使HCP-MMP v1.0中提供的卓越的皮层细分以体积(Volume)形式提供,其中还包括一些皮层下区域,并适合与像SPM和FSL这样的软件一起使用。同时,我们推荐使用基于表面的版本HCP-MMP v1.0(Glasser et al. 2016a),当需要更准确的配准时(Coalson et al. 2018)。
方法
概述
在人类连接组计划(HCP)图谱中区域的定义显示在Glasser et al.(2016a, b)的Glasser_2016_Table.xlsx中。这些区域的列表提供在表1中,并且在图1中,我们展示了带有在HCPex图谱中定义的区域标签的冠状切片。图1中的皮层区域是按照人类连接组计划(HCP)图谱(Glasser et al. 2016a)中的定义来的。
表1 HCPex图谱重新排序的皮层区域列表




第1列(重新排序的ID)显示了基于HCP-MMP1_UniqueRegionList.csv的HCPex中的顺序,如方法所述,由Glasser et al.(2016a, b)最初定义的360个皮层区域。第4列显示的皮层分区的名称来自同一个.csv文件。第6列显示了Glasser et al(2016a)使用的原始顺序。
L代表左半球,R代表右半球,MT + _Complex代表MT + _Complex_and_Neighboring_Visual_Areas,SomaSens_Motor代表Somatosensory_and_Motor,ParaCentral_MidCing代表Paracentral_Lobular_and_Mid_Cingulate,Insula_FrontalOperc代表Insular_and_Frontal_Opercular,TPO代表Temporo-Parieto-Occipital_Junction,AntCing_MedPFC代表Anterior_Cingulate_and_Medial_Prefrontal,OrbPolaFrontal代表Orbital_and_Polar_Frontal。
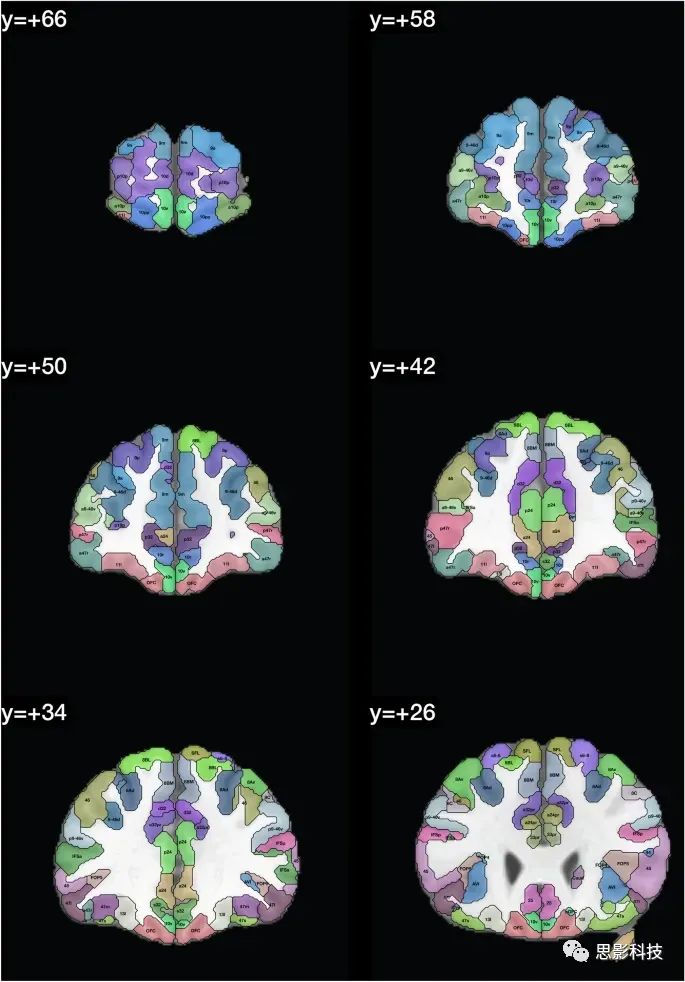
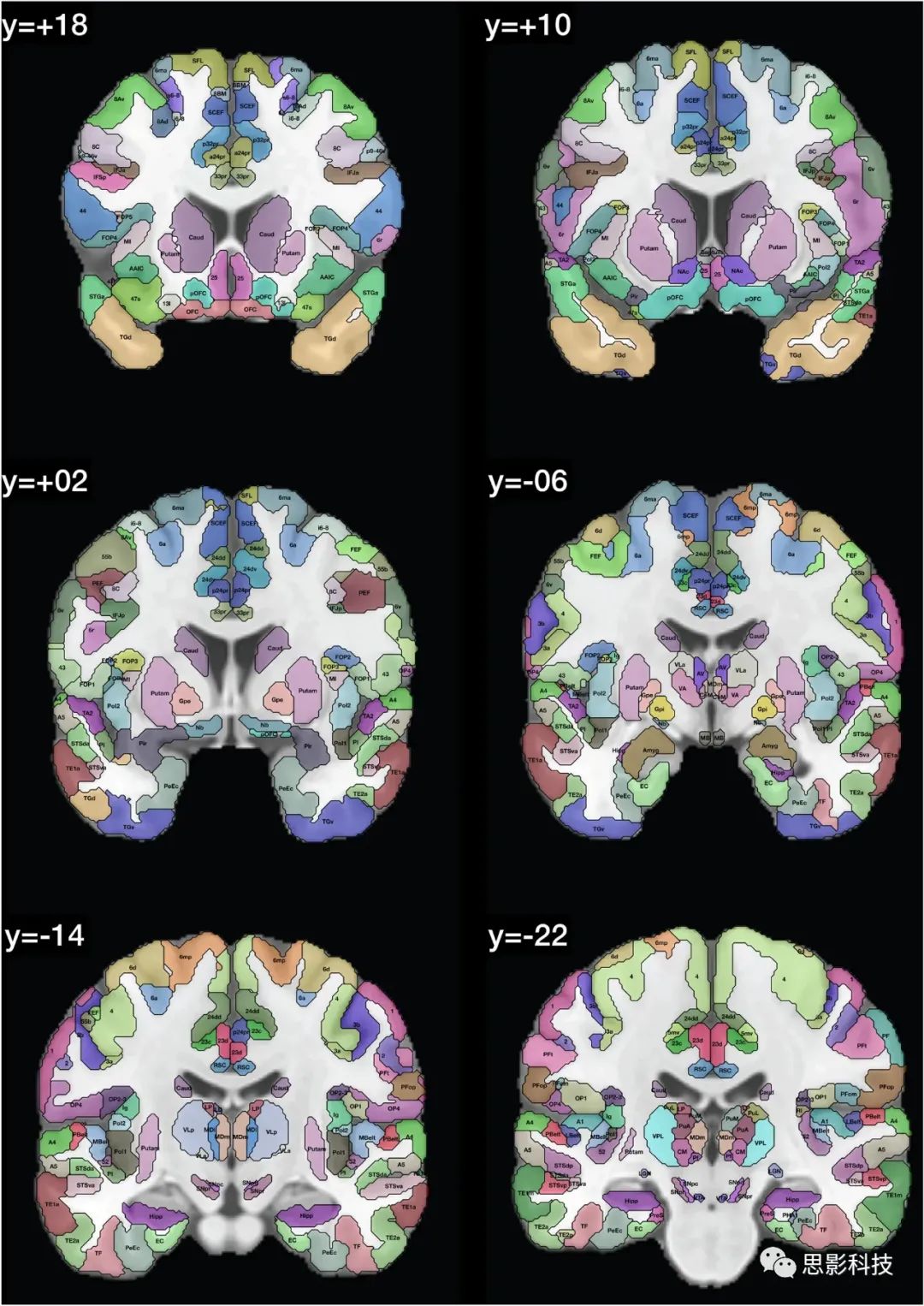
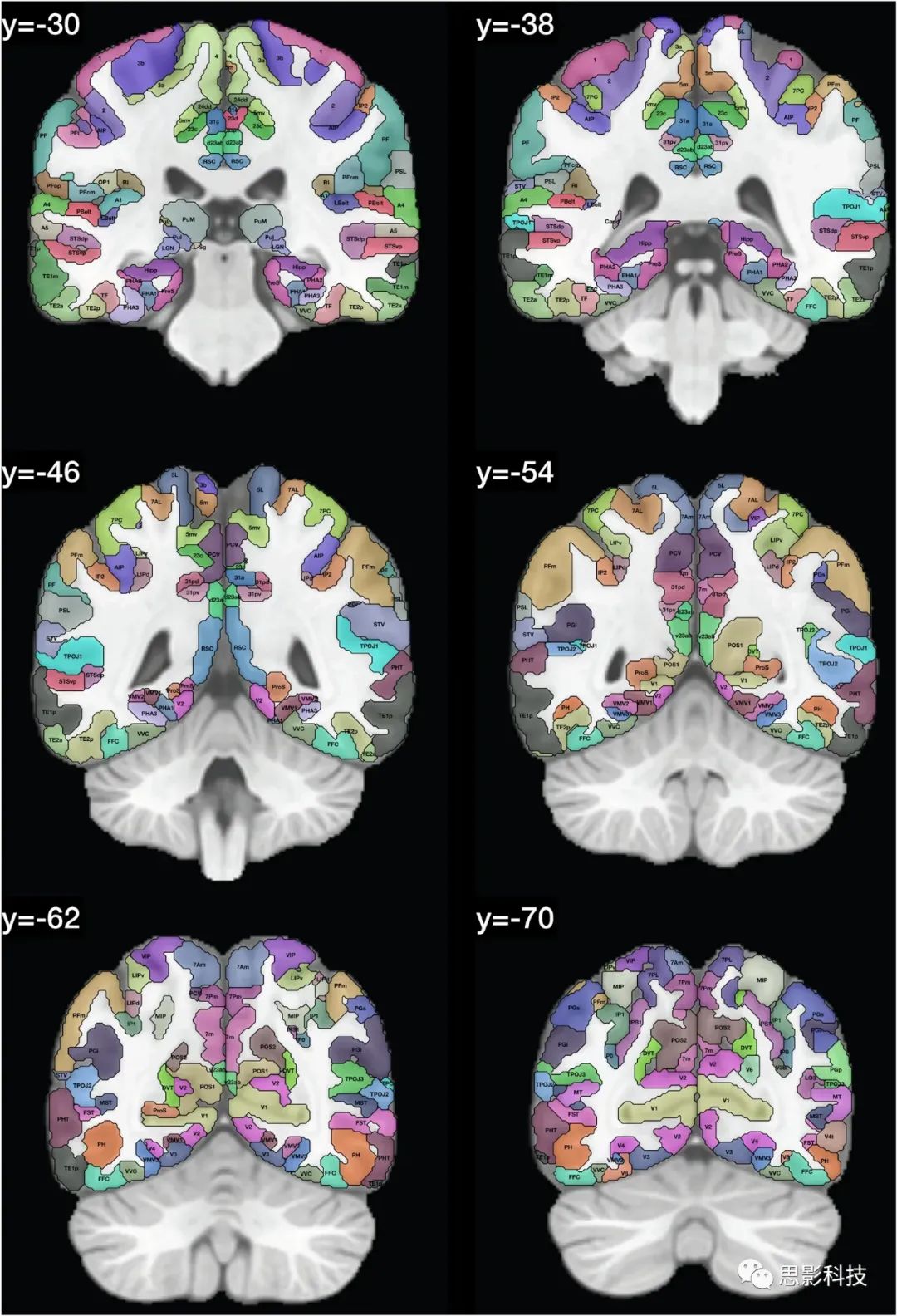
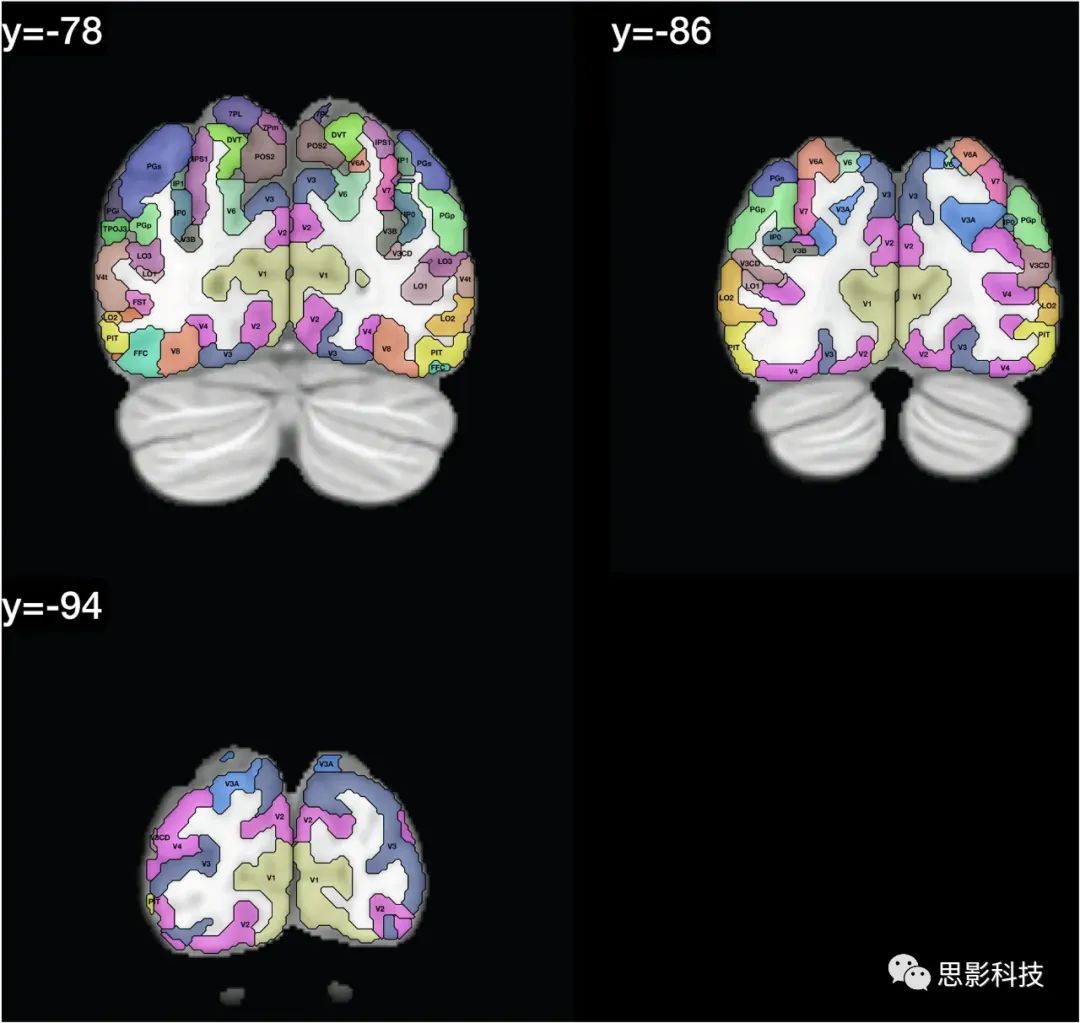
图1:展示了HCPex图谱中定义的区域和新增的皮层下区域的示例冠状切片。这些切片中使用的缩写与表1中的相同。这些冠状切片的y轴坐标是根据MNI(蒙特利尔神经学研究所)坐标系统来确定的。
这里描述的扩展图集结合了以下区域:HCP 的多模态分割 (v1.0),每个半球由 180 个区域组成(Glasser 等人,2016a); 21个丘脑核; 以及其他12个皮质下区域,包括杏仁核、壳核、尾状核、伏隔核、苍白球外部、苍白球内部、黑质致密部、黑质网状部、中脑腹侧被盖区、乳头体、隔核和基底核 。 皮层下区域列于表 2 中,也显示于图 1 中。
表2 图谱中皮下区域的列表

原始的HCP图谱是通过多模态表面匹配(MSM)方法(Glasser et al. 2016a)制作的,该方法将参与者的多模态影像配准到表面空间中,而不是在通常用于神经科学研究的标准体积(Volume)MNI152空间中。Mills(2016)和Beauchamp(2021)将HCP-MMP1图谱映射到体积MNI152模板大脑中,这是通过从多模态表面匹配格式(即原始HCP图谱中使用的MSM算法)转换到基于152名参与者的ICBM 2009c非对称模板完成的(Fonov et al. 2009, 2011)。为了将HCP-MMP1图谱从表面空间转换到MNI152体积空间,他们执行了以下步骤:(1)使用连接组工作程序将图谱从CIFTI格式转换为GIFTI格式。(2)然后将GIFTI格式的图谱重采样到基于标准网格球的FreeSurfer fsaverage空间。(3)使用FreeSurfer命令行工具“mris_convert”将图谱从GIFTI格式转换为注释文件。(4)从ICBM 2009c非对称T1体积图像生成了图谱的基于表面的版本。(5)然后应用基于表面的配准技术在fsaverage与ICBM 2009c的表面空间版本之间进行配准。(6)将HCP-MMP1的注释文件从fsaverage转换到ICBM 2009c空间。(7)再将ICBM 2009c空间中的HCP-MMP1注释转换到ICBM 2009c模板的体积空间。Mills(2016)和Beauchamp(2021)详细描述了这些处理步骤和代码(https://openwetware.org/wiki/Beauchamp:CorticalSurfaceHCP)。在ICBM 2009c的非对称MNI空间中定义的体积HCP-MMP1图谱(Fonov et al. 2009, 2011)可以在AFNI网站找到。
在HCP-MMP1图谱中,每个区域都有其Region ID,我们在表1(第6列)中展示了这些。Glasser et al(2016a)提供的补充材料文件NIHMS68870-supplement-Neuroanatomical_Supplementary_Results.pdf中有关这些区域的详细信息。在该补充材料中,基于地理位置接近性和功能相似性对区域进行了分组,并在标记为Cortical Divisions和Cortex ID的列中展示了这种分组,导致了区域的不同排序。
在这个修改版HCPex的HCP-MMP图谱中,我们根据亚利桑那大学Dianne Patterson在https://neuroimaging-core-docs.readthedocs.io/en/latest/pages/atlases.html的文件HCP-MMP1_UniqueRegionList.csv中描述的Cortex ID对区域进行了重新排序。表1显示了HCPMMP v1.0图谱中原始定义的360个区域(每个半球180个)的重新排序ID,包括缩写名、皮层区域的全名、Cortex Division、Cortex ID、Glasser et al(2016a)论文中的原始ID和体素数。表2展示了扩展的66个皮下区域(每个半球33个)及其定义。
为了进一步方便使用不同软件的HCPex图谱,我们通过在剔除颅骨的MNI152 T1 2毫米等距T1模板与ICBM 2009c非对称T1模板之间执行刚体(具有6自由度)配准,使用高级标准化工具(Ants,Avants et al. 2009)将图谱重新采样到低分辨率的标准MNI152空间中。
修改后的HCPMMP中新区域的描述
在修改后的HCP-MMP1图谱中增加了总共66个新的皮层下区域(每个半球33个)。提供了新添加的皮下细分的列表(表2)。HCPex图谱中的新皮下区域从ID 361到426(左:361–393,右:394–426)。新的皮下区域包括21个丘脑核(361–381,394–414),壳核(382,415),尾状核(383,416),伏隔核(384,417),苍白球外侧部/内侧部(385–386,418–419),杏仁核(387,420),黑质致密/网状部(388–389,421–422),中脑腹侧被盖区(390,423),以及乳头体(391,424),还有胆碱能核:隔核(392,425)和基底核(393,426)。所有的皮层和皮层下区域都配准到了使用ICBM 2009c非对称模板定义的标准MNI空间中。皮层下区域的定义在接下来的部分中描述。
杏仁核
高分辨率的杏仁核分割是基于计算脑解剖学实验室合并图谱(CoBrALab, https://github.com/CoBrALab/atlases)(Entis et al. 2012; Pipitone et al. 2014)调整的。CoBrALab图谱中的皮下区域是基于五个高分辨率的T1和T2模板手动勾画的,并且定义的分割被应用于多重自动生成模板(MAGeT)管道,以提供在MNI标准空间中的公共空间定位(Chakravarty et al. 2013)。杏仁核的分割(Treadway et al. 2015)位于与HCPex相同的MNI ICBM 2009c非线性非对称T1空间(Fonov et al. 2009, 2011)。
丘脑核
丘脑核的分割基于Iglesias et al(2018)描述的定制模块,该模块是基于来自组织学的概率图谱的贝叶斯分割算法。为了在群体水平上获得丘脑核的细分,首先,我们使用FreeSurfer软件(版本7.1.1)中的“recon-all”功能重建了178名受试者的整个大脑T1加权图像(T1w)。重建步骤包括非均匀强度标准化、剔除颅骨、灰/白质组织分割等(Fischl et al. 2002, 2004)。其次,为了获得更可靠的丘脑分割,使用了T2加权扫描作为额外的MRI体积(Volume)与T1w一起,使用“segmentThalamicNuclei.sh”功能和“t2”边界基础配准模式。第三,我们获得了包括前腹核、中央内侧核、中央外侧核、中央正中核、外侧核、外侧/内侧膝状核、外侧后核、上膝状体、内侧小细胞核、内侧大细胞核、连接核、束旁核、前/下/外侧 /内侧枕核、腹侧前核/外侧前核/外侧后核/后外侧核。第四,为了将个体的丘脑核映射到相同的标准立体定位空间,使用了ANTs(Avants et al. 2011)中实现的3阶段(刚性+仿射+非线性)对称标准化(SyN)算法,在每个个体的灰质部分与MNI ICBM 2009c模板的灰质组织部分(mni_icbm152_gm_tal_nlin_asym_09c)(Fonov et al. 2009, 2011)之间进行配准。计算出的线性和非线性变换被应用于每个个体的丘脑核分割,然后变形到MNI ICBM 2009c非线性非对称T1空间。最后,我们采用了赢家通吃策略,使用我们为178名HCP参与者进行的分割,为HCPex图谱中的每个体素标记属于特定丘脑核的最高概率,以获得MNI ICBM 2009c标准空间中丘脑核的最终群体水平图谱。
FreeSurfer模块将丘脑分割为每个半球25个不同的核团。然而,一些丘脑核团非常小,以至于它们在空间上可能不准确或不适用于进一步的基于感兴趣区(ROI)的分析。因此,我们通过根据它们的原始定义或最近邻居合并小核团来减少丘脑核团的数量。在HCPex图谱中定义的结果产生的21个丘脑核团的详细描述显示在表1中。
Iglesias et al.(2018)提供的丘脑细分使得HCPex图谱中每个半球包括21个丘脑核团,并因此被优先使用,而不是Tian et al.(2020)提供的只有8个丘脑核团的图谱。从Iglesias et al.(2018)采用的丘脑细分已通过使用基于人脑组织学的Krauth et al.(2010)图谱进行了验证,如补充材料中所描述的(图S1)。作为进一步的验证,采用白质消除MP-RAGE成像技术,将丘脑分割为12个核团的Thalamus Optimized Multi Atlas Segmentation(THOMAS)图谱中的人类丘脑核团也显示在图S1中(Su et al. 2019)。
壳核、尾状核、伏隔核、苍白球外侧部/内侧部、黑质致密部/网状部、中脑腹侧被盖区、乳头体、隔核和基底核
从强化学习图谱(Pauli et al. 2018)调整了九个区域,包括壳核、尾状核、伏隔核、苍白球外侧部、苍白球内侧部、黑质致密部、黑质网状部、中脑腹侧被盖区和乳头体。强化学习图谱定义了许多有用的皮层下核团,特别是用于奖励学习和决策制定,在168名年龄在22至35岁之间的典型成年人中使用高分辨率T1和T2加权结构图像。在同一个标准MNI ICBM 2009c非线性非对称模板空间(Fonov et al. 2009, 2011)中定义的确定性标签直接应用于当前修改的图谱HCPex。
包含胆碱能神经元的隔核和基底核是由Zaborszky等人(2008)基于组织学连续切片的细胞结构映射定义的。然后将巨细胞群的立体定位概率图分离为Ch1-2、Ch3和Ch4区域。对于HCPex图谱,使用了Zaborszky提供的单一受试者MNI标准空间中Ch区域的最大概率图(MPM),其中HCPex中的Ch1-2结合为隔核,Ch3和Ch4结合为基底核。MPM的生成在之前的研究中有描述(Eickhoff等,2005, 2006)。Zaborszky等(2008)将10个大脑中Ch细胞群的MPM合并成一幅图像(ChAll),通过为每个体素分配一个标签,该体素具有最高概率或超过40%的阈值。Zaborszky等将原始组织学体积标准化到单一受试者T1模板(colin27)(Holmes et al. 1998)。因此,为了将其与人群平均的MNI ICBM空间映射,我们使用了ANTs中的SyN算法(Avants et al. 2011)在提取脑部的单一受试者T1模板和MNI ICBM 2009c非对称T1模板之间进行配准。计算出的转换应用于群体水平的最终ChAll图并标准化到MNI ICBM 2009c非线性非对称T1空间。最后,通过移除位于白质的体素(如前联合)来校正标准化图。
结果
图1提供了扩展HCP图谱的标记版本,以帮助读者识别这些人脑冠状切片中的脑区域。值得注意的是,一些小的脑结构在这些冠状切片中可能显得非常小,因为它们是被8毫米分隔开的(例如隔核就位于MNI坐标y:3–10之间)。表1和表2提供了图谱中脑区域的列表。在这个重新排序的列表中,标签列表提供在HCPex_LabelID.mat中。
用于生成HCPex体积空间中皮层区域的过程,有用地通过与Coalson等人(2018)用来生成HCP-MMP1图谱体积版本的过程进行了验证,如补充材料中所示(图S2)。
扩展的HCP图谱已经在包括SPM、FSL、MRIcroGL、FreeSurfer的查看器(freeview)在内的公开脑成像软件上进行了测试,并且还与AAL3软件兼容。安装的用户指南提供在补充材料中。该图谱位于体积空间中,旨在使其成为研究人脑的有用工具,特别是对结构和功能连接组的重建和分析。为了方便在不同类型的研究中使用,该图谱提供了两种不同分辨率,具有各向同性体素大小1×1×1毫米和2×2×2毫米,以及用于标准化和可视化的剔除颅骨的ICBM 2009c非对称T1模板。
用户应该注意,像丘脑中的连接核和界限核(在1立方毫米版本中<10个体素)这样的小区块,在低分辨率fMRI研究中经过非线性标准化后可能会被遗漏。此外,由于皮层和皮层下区域最初是基于年轻成人的脑影像定义的,在进行与年龄相关的研究时应该小心,并建议进行视觉检查。
讨论
本文描述的HCPex图谱扩展了HCP-MMP1图谱(Glasser et al. 2016a),通过增加66个皮下区域,以体积(volume)形式提供以适用于多种类型的神经影像软件,包括SPM,提供带标签的大脑冠状切片以清晰可视化HCPex图谱中定义的皮层和皮下区域(图1),并提供图谱中皮层区域的可选重排序(表1)。我们已经发现,有了这些扩展,HCPex图谱非常有帮助(Huang et al. 2021; Ma et al. 2021; Rolls et al. 2021)。
HCPex和AAL3(Rolls et al. 2020)提供的丘脑细分相似,但在以下方面有所不同。AAL3使用与HCPex相同的丘脑核团分割方法(Iglesias et al. 2018),但用户应注意应用的T1模板存在差异。AAL3中的丘脑核团是用单一受试者colin27(扫描27次)模板图像分割和定义的,这些图像与MNI305空间线性配准。HCPex中的丘脑核团分割是基于在MNI152数据库中152个无偏差的非线性平均生成的平均模板,因此不会受到任何单一受试者解剖特征的偏见(Fonov et al. 2011)。此外,HCPex中定义了limitans suprageniculate丘脑核团,而在AAL3中未包括。
HCPex图谱在使用弥散追踪分析人类海马记忆系统的连接中已经证明非常有用(Huang et al. 2021)。该图谱使得可以描述在HCP-MMP图谱中如此精美细分的许多视觉和相关皮层区域与人类海马的连接,此外还使得可以将海马记忆系统与其他大脑区域(如海马旁回、扣带皮层和眶额皮层)的连接纳入分析(Huang et al. 2021)。例如,它使得可以揭示海马与HCP图谱区域VMV1-3和PHA1-3(即TH)之间的连接,这些区域包括海马旁回位置区域(Sulpizio et al. 2020)。这是一个重要的发现,因为它有助于阐明海马空间视野细胞如何接收输入,这些细胞对场景中观察到的位置做出反应(Rolls et al. 1997; Georges-François et al. 1999; Rolls and Wirth 2018; Rolls 2021),因此在人类记忆和导航中非常重要(Kesner和Rolls 2015; Rolls 2018, 2021)。如果使用其他图谱,由于它们对这些大脑区域的细分不够详细和多模态,可能无法揭示这一点。
局限性
我们注意到,在HCP-MMP图谱(Glasser et al. 2016a)中定义的区域是基于表面的映射定义的,这对于精确的配准具有优势(Van Essen et al. 2017; Coalson et al. 2018; Dickie et al. 2019)。这里描述的HCPex图谱位于体积空间中,正如Coalson等人(2018)所述,体积(Volume)版本可能不如基于表面的版本准确,因为不同参与者之间皮层折叠的变异性可能会影响体积空间中的配准。HCPex图谱在体积空间中与Coalson等人(2018)提供的体积图谱的验证提供在补充材料中(图S2),其中比较了制作这两个体积图谱的方法。HCPex图谱的优势是它扩展到包括许多皮层下区域,并且以体积(Volume)形式存在,许多神经影像分析程序都可以直接使用,包括但不限于SPM,FSL,AAL-AAL3工具箱,FreeSurfer和MRIcroGL。我们建议,如果需要非常精确地识别皮层区域,则在HCPex分析之后使用基于表面的空间进行分析会是有用的。
总之,这里描述的HCPex图谱通过增加66个皮层下区域、以体积(Volume)形式提供以适用于多种类型的神经影像软件(包括SPM)、提供带标签的大脑冠状切片以清晰可视化HCPex图谱中定义的皮层和皮层下区域(图1)以及提供图谱中皮层区域的可选重排序(表1),从而扩展了HCP-MMP1图谱(Glasser et al. 2016a)。
补充材料:
人类连接组计划扩展图集 (HCPex) 中提供的丘脑分区验证
HCPex图谱中提供的丘脑细分验证 Iglesias等人(2018)提供的丘脑细分使HCPex图谱每个半球包括21个丘脑核团,并因此被优先使用,而不是Tian等人(2020)提供的只有8个丘脑核团的图谱。从Iglesias等人那里采用的丘脑细分已通过使用基于人脑组织学分析的Krauth等人(2010)图谱进行验证,并且如图S1所示,HCPex的分区与之很好对应。为了进一步验证,还展示了图S1中的Thalamus Optimized Multi Atlas Segmentation(THOMAS)图谱中的人类丘脑核团,该图谱使用白质消除MP-RAGE成像技术将丘脑分割成12个核团(Su等人,2019)。

图S1. 使用Krauth等人(2010)的基于组织学的人类丘脑图谱验证HCPex图谱中的丘脑细分。作为比较,还展示了使用白质消除MP-RAGE成像技术将丘脑分割为12个核团的Thalamus Optimized Multi Atlas Segmentation(THOMAS)图谱中的人类丘脑核团(Su等人,2019)。右侧是丘脑的T1图像。
顶部行展示冠状视图;中间行水平视图;底部行展示矢状视图,标有y、z和x的MNI坐标。Krauth等人(2010)的图谱是组织学的,并没有MNI坐标。缩写在表1中展示。
HCPex中体积(Volume)空间皮层区域的验证
用于在HCPex的体积空间内生成HCP-MMP图谱(Glasser et al. 2016)皮层区域的方法(见主论文中的方法),有效地通过了Coalson等人(2018)用于生成HCP-MMP图谱(Glasser et al. 2016)体积版本的方法的验证,如图S2所示。Coalson等人(2018)制作的Glasser等人图谱(2016)的体积版本,已从公开发布的细分版本(https://balsa.wustl.edu/file/show/nvrZ)中下载。HCPex论文中的皮层区域与Coalson等人(2018)采用的方法相当吻合。两种方法的主要区别在于Coalson等人(2018)使用了210名HCP参与者的T1图像,每个都映射到MNI体积空间中,并制作了体积空间中的群体平均;而HCPex则从210名HCP参与者提供的基于表面的空间中的群体平均开始(Glasser et al. 2016),然后如方法所述将其映射到MNI体积空间(Mills 2016; Beauchamp 2021)(另见https://wiki.humanconnectome.org/display/PublicData/HCP+Users+FAQ),并在AFNI网站上提供(https://afni.nimh.nih.gov/pub/dist/atlases/MNI_HCP/)。

图S2. 通过与Coalson等人(2018)制作的相应细分(标记为HCPMMP1)进行比较,验证HCPex中体积空间的皮层区域。在两种情况下,每个半球的180个皮层区域均来源于基于表面的HCP-MMP1图谱(Glasser et al. 2016)。缩写在表1中列出,两个图谱的颜色相同。显示的y值是MNI坐标。两个版本的图谱大体上是一致的,小的差异部分是因为HCPMMP1图谱使用了HCP的T1模板,而HCPex图谱使用了广泛应用的MNI ICBM 2009c模板,这是基于152名参与者的非对称T1模板(Fonov et al. 2009; Fonov et al. 2011)。
如需原文及补充材料请添加思影科技微信:19962074063或18983979082获取,如对思影课程及服务感兴趣也可加此微信号咨询。另思影提供免费文献下载服务,如需要也可添加此微信号入群,原文也会在群里发布,如果我们的解读对您的研究有帮助,请给个转发支持以及右下角点击一下在看,是对思影科技的支持,感谢!























 1054
1054

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








