神经影像可视化是解释和交流科学结果的核心,同时也是数据质量控制的基石。通常,这些图像和图表是通过在图形用户界面(GUI)上手动更改设置来生成的。现在已经存在许多文档齐全的基于代码的脑部可视化工具,允许用户使用代码在诸如 R、Python 和 MATLAB 等编程环境中以编程方式直接生成适合发表的图形。在本文中,我们通过强调与基于 GUI 工具相比,基于代码生成的脑部可视化在可复制性、灵活性和集成性方面的相应优势,为广泛采用代码生成的脑部可视化提供了理由。然后,我们提供了一个实用指南,概述了生成这些基于代码的脑部可视化所需的步骤。我们还展示了一个当前可用于程序化脑部可视化的工具的综合表格,并提供了可视化示例及相关代码作为参考。最后,我们提供了一个网页应用程序,可以生成这些可视化的简单代码模板作为起点。本文发表在Aperture Neuro杂志。
1. 引言
神经影像数据的可视化是评估数据质量、解释结果和传达研究发现的主要方式之一。这些可视化通常使用基于图形用户界面(GUI)的工具生成,用户在每个实例中打开单独的图像,并手动更改显示设置,直到达到所需的输出。在很大程度上,选择使用基于 GUI 的软件是由于其便捷性、灵活性和可访问性的认知。然而,现在已经存在文档齐全的基于代码的软件包,这些软件包不需要高级和全面的编程知识,使其对神经影像社区更加易用。这些工具灵活且能够生成可复现的高质量脑部可视化,且只需几行代码即可生成适合发表的图形,尤其是在 R、Python 和 MATLAB 环境中。在本文中,我们首先通过强调在可复制性、灵活性和集成性方面的主要优势,讨论了广泛采用代码生成可视化的理由。然后,我们提供了一个实用指南,概述了生成基于代码的脑部可视化所需的步骤,并提供了一个网页应用程序(https://sidchop.shinyapps.io/braincode/;见第 4 节),可以生成这些可视化的简单代码模板作为起点。我们还展示了一个当前可用于程序化脑部可视化的工具的综合表格(表 1),并提供了可视化示例及相关代码作为参考(图 2-3)。最后,我们概述了基于代码工具当前功能中的一些局限性和不足。本指南的重点是人脑磁共振成像(MRI)数据,但讨论的许多原则和提供的工具同样适用于可视化来自其他器官和成像模式(如 EEG、MEG、PET 和 CT)的数据。
表 1. 可直接在 R、MATLAB 和 Python 环境中访问的基于代码的神经影像可视化工具示例
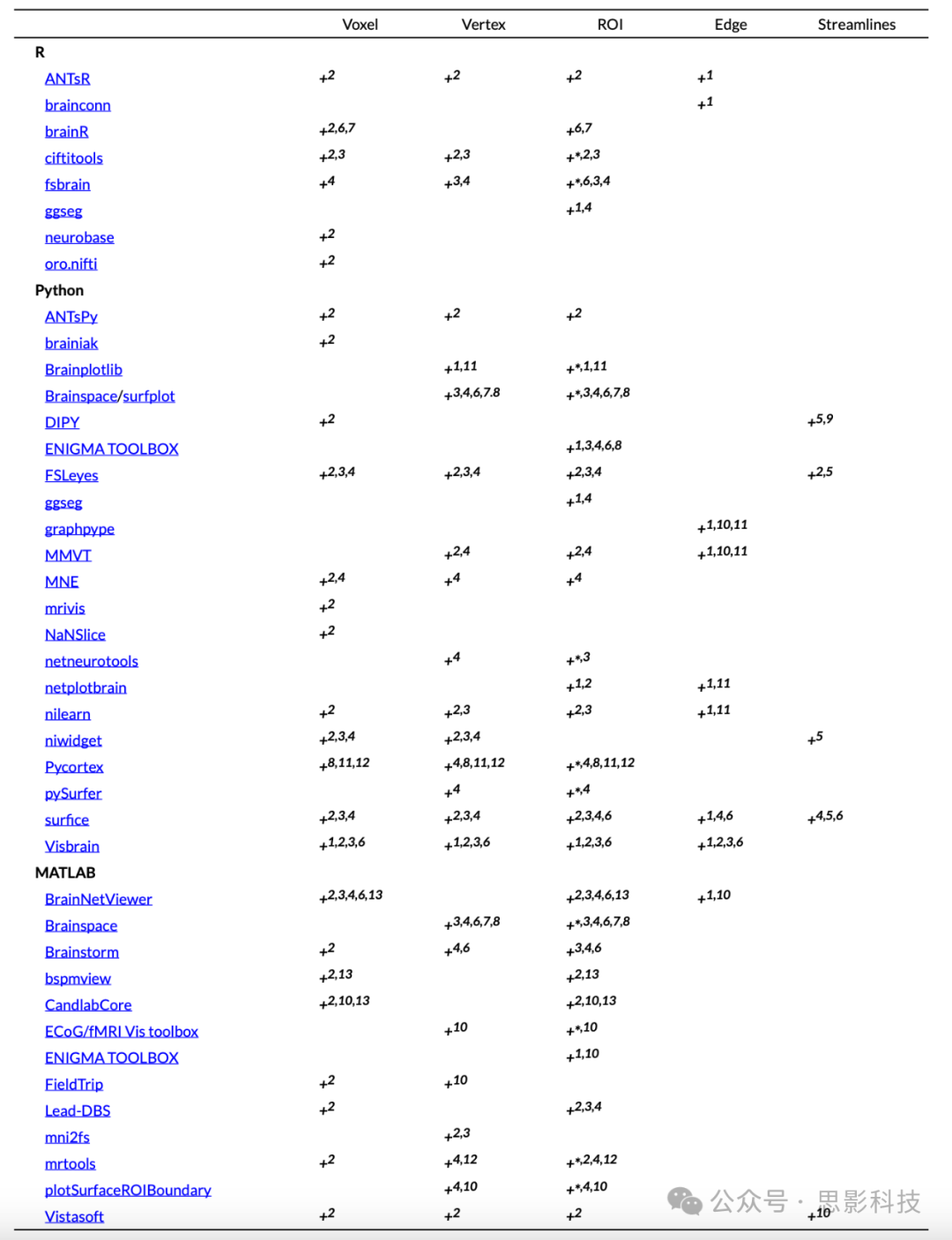
注意: 列出的工具包含通过用户在 R、MATLAB 和 Python 环境中输入代码生成(至少接近)适合发表的神经影像图形所需的功能。该表的互动版本可在此查看:https://sidchop.shinyapps.io/braincode_selector/。此列表不包括跨平台的通用可视化软件。
1 = .txt/.csv(输入为标量、向量、矩阵);
2 = .nii/.nii.gz(输入为 NIfTI 格式);
3 = .cii/.gii(输入为 CIFTI 或 GIFTI 文件,包括任何子类型,例如 dlabel、dtseries、.surf);
4 = FreeSurfer 格式作为输入,包括 .mgz、.annot、.label、.curv、.wm 等;
5 = .trk/.tck(输入为束流图);
6 = .obj(3D 对象格式);
7 = .ply(3D 多边形格式);
8 = .vtk(Visualization Toolkit 格式);
9 = .fib(Legacy vtk 格式);
10 = .mat(MATLAB 格式);
11 = .npy/.npz(Python NumPy 格式);
12 = .off(对象文件格式);
-
= 仅限 Cortex。
2. 学习生成基于代码的脑部可视化的好处
2.1. 可复制性
近年来,多个大规模研究实证证明使用神经影像数据的研究结果缺乏可重复性。实现稳健和可靠发现的一个常见解决方案是鼓励能够透明评估和独立复制的科学成果。实际上,这通常意味着公开分享详细的方法、材料、代码和数据。虽然共享与神经影像分析相关的代码已有上升趋势,但用于生成诸如脑部渲染和空间图的图形代码的共享相对被忽视。这种可复制性差距部分是由于脑部图形通常通过手动过程创建,涉及在 GUI 上调整滑块、按钮和叠加层,最终截取屏幕截图,并有时在图像处理软件(如 Illustrator、Photoshop 或 Inkscape)中进行美化。这样的过程使得神经影像可视化本质上难以复制,甚至连作者自己也难以复制。
用于数据可视化的代码应体现开放科学的核心特征。鉴于脑部图形常常是论文、会议演示或新闻报道中解释的核心部分,确保它们能够被可靠地再生成对于知识的生成和传播至关重要。通过编写和分享用于生成脑部可视化的代码,建立了基础数据与相应科学图形之间的直接且可操作的联系。虽然这些代码不一定反映科学发现的有效性或准确性,但它们允许实现可重复性,增强透明度和稳健性,同时展示了进一步科学知识的意愿。一些人甚至认为,无法复制的图形更接近于广告而非科学。
值得注意的是,一些基于 GUI 的工具历史上提供了命令行访问以生成可复制的可视化(例如 FreeView、FSLeyes、surfice),使其使用潜力上与纯代码工具同样具备可复制性。这些专用命令行接口的使用对于那些缺乏编码环境经验的用户来说,提供了一个有用的中间地带。尽管如此,这些接口通常仍然存在学习曲线,并且可能缺乏编程环境所提供的其他优势,如迭代能力(见第 2.2 和 2.3 节)。同样,其他基于 GUI 的工具通过自动生成批处理脚本(包含可重新执行的专用命令行的文本文件)或内置终端提供可复制性,这些脚本或终端可能具有特殊性,并且可能缺乏文档,使得不熟悉特定软件的用户难以轻松使用或复制。
2.2. 灵活性和可扩展性
通过代码精确复制图形具有开放科学实践之外的显著优势。特别是,重新编程输入(例如统计图)和设置(例如配色方案、阈值和视觉方向)的能力可以简化整个科学工作流程。通过代码更改输入和设置,可以轻松生成多个图形,例如来自需要类似可视化的多重分析的结果。一个简单的 for 循环或带有更改输入和/或感兴趣设置的绘图函数可以成为探索可视化选项或快速创建多面板图形的强大方法。同样,当相关图形可以通过几行代码重新生成时,应审稿人或合作者提出的修改图像处理或分析的艰巨请求将不再是负担,而无需手动重新粘贴和重新绘制图形。拥有一个可修改输入的代码库意味着生成可视化所需的时间、精力比基于 GUI 的特定图像和实例生成更少。这也使得在后续项目中生成一致的图形变得更容易。关键是,为图形编写代码的收益是累积的,除了提高编程技能之外,还可以构建一个用于图形生成的代码库,在整个科学生涯中重复使用和共享。
通过代码对可视化设置(如配色方案、图例位置和摄像机角度)进行精确控制,提供了比可视化更大的灵活性。然而,基于 GUI 的工具的部分吸引力在于,这些设置的预设可以提供一个有用的起点,并减少初学者的决策负担。然而,大多数基于代码的包通常也提供类似的预设,通常以默认设置的形式存在,用户无需手动输入创建图像所需的每一个选择。大多数基于代码的工具还附带文档,CRAN 或 Neuroconductor仓库中的 R 包需要详细的指导。最近的工具已经开始在 GitHub 仓库中包含详细的、适合初学者的文档,甚至包括完整的论文(例如,Pham, Muschelli, & Mejia, 2022; Mowinckel & Vidal-Piñeiro, 2020; Huntenburg et al., 2017; Schäfer & Ecker, 2020),这些论文提供了可供新用户使用的图形示例,作为起点或模板(另见第 4 节)。随着图形代码共享的普及,将会有大量的模板可作为新图形的基础。
虽然脑部可视化通常被认为是分析的最终结果,但它们也是成像数据质量控制的重要组成部分。用于自动检测伪影、去噪数据和生成衍生物的工具正在变得更加稳健,但尚未达到无需可视化数据的阶段。然而,当处理大型数据集(如人类连接组计划或英国生物银行)时,使用传统的基于 GUI 的工具进行视觉检查是不切实际的。打开单个文件并达到所需的可视化设置所需的时间在处理大型数据集时会大幅增加。掌握编程生成脑部可视化的方法可以允许在大型数据集的每个图像上迭代可视化代码,使每个数据处理步骤的质量检查成为可能。每次迭代的视觉输出可以编译成可访问的文档,便于滚动查看,更高级的用法还允许创建交互式 HTML 报告(见第 3.5 节),类似于标准化数据处理工具(如 fmriprep)创建的报告。这种在更大数据集上进行视觉质量控制的能力将改善处理错误的识别,并导致更可靠和有效的研究结果。
2.3. 集成与互动报告
在神经影像学研究中,编程语言如 R、Python 和 MATLAB 通常用于统计分析和生成非脑部图形,但脑部图形则外包给诸如 FSLeyes、Freeview 或 ITK-snap 等独立的基于 GUI 的工具。越来越受欢迎的软件,如R Markdown、Quarto和Jupyter Notebook,允许在单个脚本中混合文本和代码,从而生成完全可重复和可发表的论文。通过使用环境中可用的基于代码的工具,可以将脑可视化直接集成和嵌入到论文或报告中。例如,当前论文的完全可重复版本可以在GitHub上找到。一些发表神经影像学研究的期刊正在推动可重复的稿件,包括可重复的图表(如eLife、Aperture Neuro),其他期刊如F1000Research和GigaScience甚至允许通过云平台如Code Ocean(Code Ocean,2021)重新运行与相关文章关联的代码。
神经影像数据通常在空间上是三维的,并且可能具有多个时间点,增加了第四维(例如,功能性影像数据)。因此,使用静态的二维切片来传达研究发现或评估数据质量具有挑战性,且可能不是数据或相关解释的最佳表示方式。虽然精心策划的三维渲染可以帮助进行空间定位,但最终,静态图像只能提供数据的不完全表示,它们迫使研究人员选择展示“最佳”角度或切片,这通常涉及牺牲一个结果以强调另一个结果。一些基于代码的工具的另一个优势是能够生成“丰富”的媒体,如互动图形或动画,允许用户缩放、旋转和滚动浏览切片。以这种方式与图形互动可以改善研究发现的科学传播。将这些视频或互动图形链接或甚至嵌入到论文中,可以大大增强研究发现的传播效果,并使论文对读者更具吸引力。这种丰富的脑部可视化适合在学术论文之外的科学传播媒介上分享——如演示文稿、网站和社交媒体——所有这些都可以促进与同行的研究交流并接触更广泛的受众。随着社交媒体成为通过科学传播向公众传播发现的核心媒介,这一点变得越来越重要(如向公众、研究社区,甚至成为早期研究人员就业机会的主要途径)。总体而言,公众参与是科学的基石,我们创建的图像处于这一过程的核心。
3. 生成基于代码的可视化
在接下来的部分中,我们将概述生成程序化和可重复的人脑可视化所需的主要步骤(见图 1),并提供工具和启发式方法以指导这一过程。
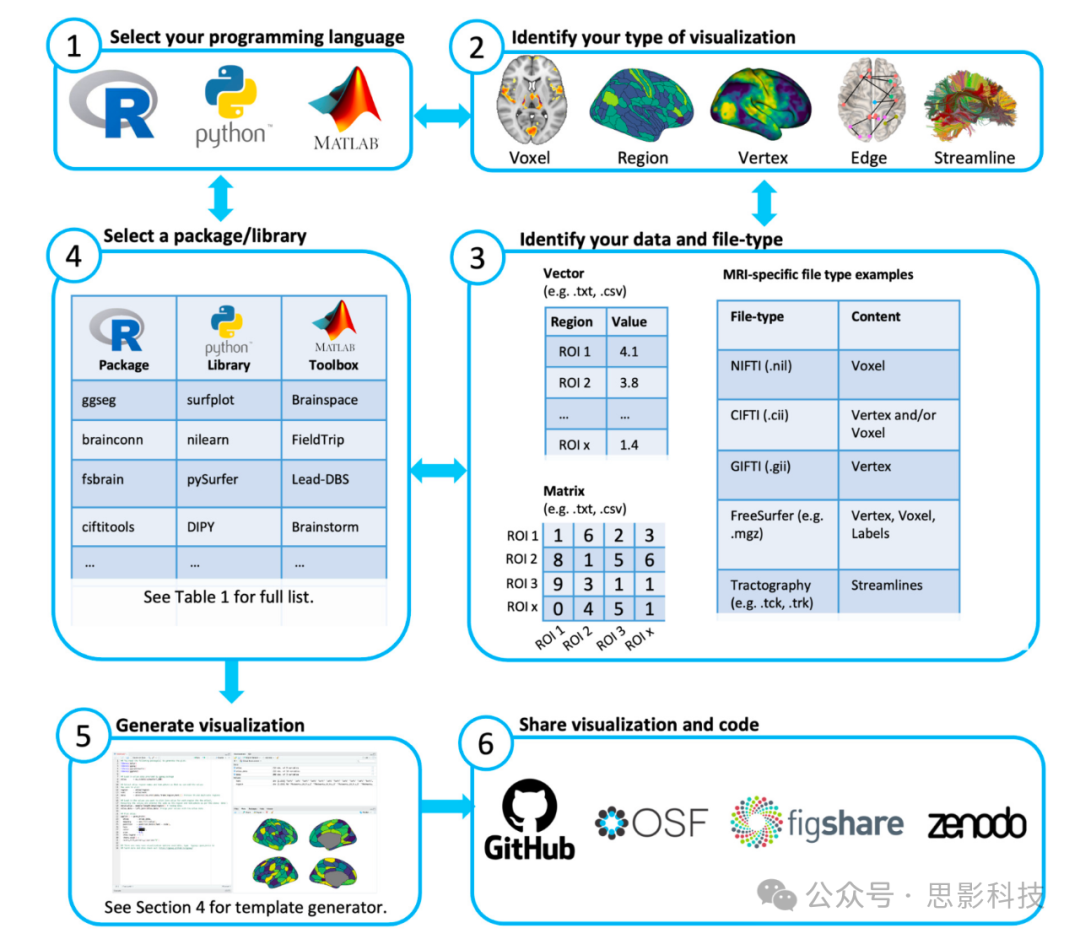
图 1. 生成程序化和可复现的人脑可视化的主要步骤。每个步骤在第 3 节的相应子部分中进行了详细说明。
3.1. 选择编程语言
生成基于代码的可视化的第一步是选择编程语言。三种最流行的语言是 R、Python 和 MATLAB,它们都有许多生成脑部可视化的选项(见表 1)。这一决定可以基于用户之前使用的语言,或者项目分析部分所使用的语言。使用相同的语言进行可视化和分析可能具有优势,因为在不同环境之间切换,或切换到基于 GUI 的可视化软件,可能会对科学工作流程造成繁琐的偏离。这会使调试错误更加困难,因为用户必须定期切换程序以直观地检查对先前分析的任何修改或调整的结果。使用所选编程环境中已存在的脑部可视化工具,可以即时获得对处理或分析修改影响的视觉反馈。
另外,编程语言的选择可能由所需的可视化和数据类型决定。例如,从束流追踪中可视化流线可能在 R 环境中目前不可用(见表 1),因此需要使用 Python 或 MATLAB。其他限制可能包括对 MATLAB 等专有软件的访问受限,这将需要使用开源选项,如 R、Python 或 Octave。
3.2. 确定可视化类型
神经影像数据及其衍生物可以以多种形式进行可视化,这些形式具有不同的关联文件类型和可视化需求(见第 3.3 节)。下面简要描述了一些较为流行的可视化类型:
体素。 在神经影像学中,体素用于表示 3D 扫描(如 MRI 或 CT 扫描)的强度值。体素可以以不同的颜色渲染,以指示组织类型或其他感兴趣的特征。例如,在功能性 MRI 扫描中,体素可以根据其激活水平进行着色,以显示在特定任务期间大脑的哪些区域更为活跃。这些可视化通常以轴向、矢状或冠状平面的切片(图 2A-B;图 3A-B)或整个大脑的 3D 渲染形式显示。统计值以覆盖层的形式显示在遵循常见立体定向坐标系统(例如 MNI152)的模板解剖图像上,或显示在个体特定的解剖图像上。
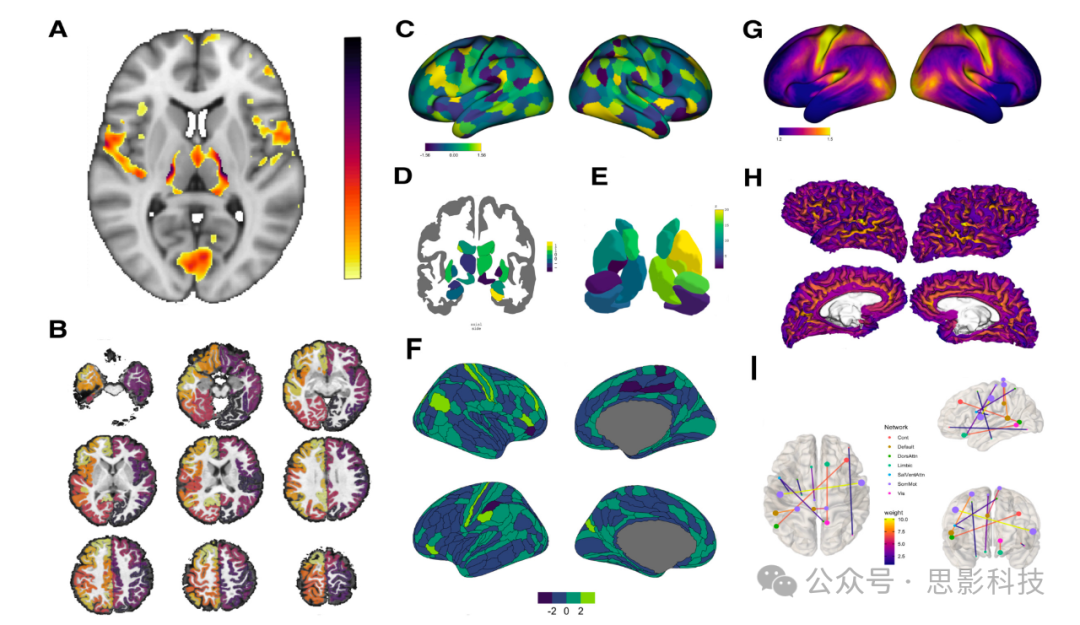
图 2. 使用 R 制作的脑部影像可视化示例
A) 在 T1 加权模板图像上阈值化并叠加的体素级统计图,显示单个轴向切片。使用 neurobase 包中的 ortho2 函数制作。
B) 在个体 T1 加权图像上叠加的体素级皮层分区,以 9 切片轴向方向显示。使用 neurobase 包中的 overlay函数制作。
C) 以 CIFTI 格式的表面 ROI 图集,每个区域分配了相应的统计值,双半球显示在侧视的膨胀模板表面上。使用 ciftiTools 包中的 view_xifti_surface函数制作。
D) 赋值给每个区域的冠状横截面下皮层结构渲染。使用 ggseg 包中的 aseg 图集制作。
E) 赋值给每个区域的 9 个双侧下皮层区域的 3D 渲染。使用 ggseg3d 包中的 aseg 图集制作。
F) 在膨胀的皮层表面上显示 ROI 图集的内侧和外侧视图,每个区域分配了相应的值。使用 ggsegGlasser 包中的 glasser 图集,通过 ggseg 绘制。
G) 在膨胀模板表面上显示的 CIFTI 格式顶点级数据的侧视图。使用 ciftiTools 包中的 view_xifti_surface 函数制作。
H) 在个体的白质表面上显示的顶点级数据的内侧和外侧视图。使用 fsbrain 包中的 vis.subject.morph.standard 函数制作。
I) 在 MNI 坐标空间中的大脑示意图的顶部、左侧和前视图上绘制的加权无向图。使用 brainconn 包中的 brainconn函数制作。所有用于编制此图的代码以及每个面板的内容均提供在附带的在线存储库中。
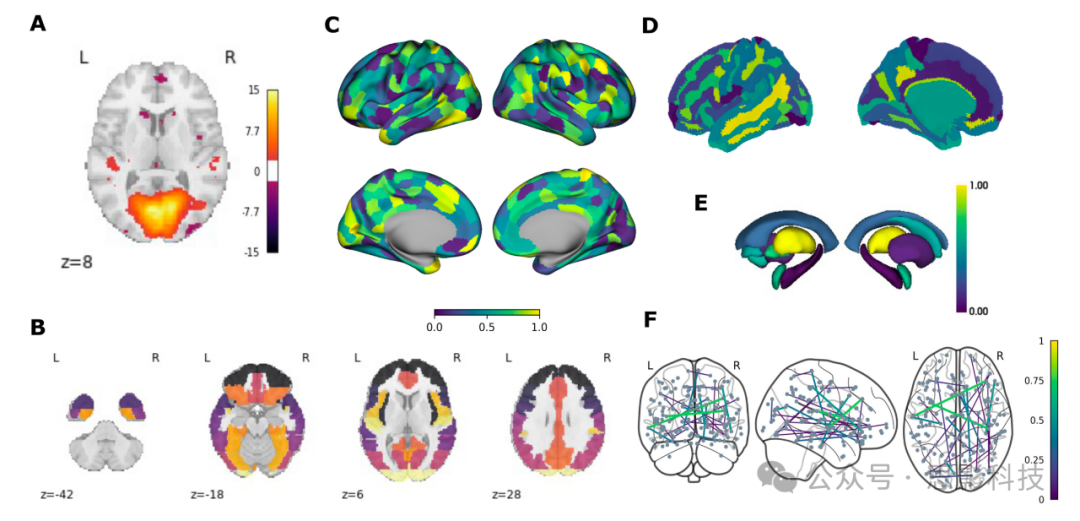
图 3. 使用 Python 制作的脑部影像可视化示例
A) 在 T1 加权模板数据上阈值化并叠加的基于体素的统计图,显示单个轴向切片。使用 nilearn 包中的 plotting.plot_stat_map函数制作。
B) 在 T1 加权 MRI 数据上叠加的体素级皮层分区,显示四个轴向切片。使用 nilearn 包中的 plotting.plot_roi函数制作。
C) 在膨胀的模板表面上显示的全皮层 ROI 图集的内侧和外侧视图,每个 ROI 随机分配了一个统计值。使用 surfplot 包中的 Plot函数制作。
D) 在个体的白质表面上以 3D 渲染方式显示的顶点级数据的外侧和内侧视图。使用 nilearn 包中的 plotting.view_surf函数制作。
E) 来自 Deiskan-Killiany 图集的 16 个下皮层结构的 3D 渲染,每个区域随机分配了一个统计值。使用 ENIGMA TOOLBOX 包中的 plot_subcortical函数制作。
F) 在 MNI 坐标空间中的大脑示意图的前视、右视和顶视图上绘制的加权无向图。使用 nilearn 包中的 plotting.plot_connectome函数制作。所有用于生成每个面板内容及编制此图的代码均提供在附带的在线存储库中。
顶点。 在神经影像学中,顶点用于创建大脑结构(如大脑皮层或下皮层区域)的网格表示。每个顶点都有一组坐标,指定其在三维空间中的位置,并与其他顶点连接形成三角形,构成网格。顶点可以用于创建大脑表面的三维可视化,并根据不同的属性(如脑沟深度、厚度或功能激活)进行颜色编码。这些可视化通常以双半球的内侧和外侧视图的三维渲染形式显示(图2C、G-H;图3C-D)。统计值以覆盖层的形式显示在遵循常见立体定向坐标系统且顶点数量固定的模板表面上(例如 fsaverage;图2C、G;图3C-D),或显示在使用解剖图像重建的个体特定表面上(图2H)。
感兴趣区域(ROI)。 在神经影像学中,ROI用于识别与研究问题相关的特定大脑结构或区域。ROI可以通过各种方法定义,例如手动描绘、基于图集的分区或功能激活模式。可视化ROI可以通过将相同的统计值或颜色分配给一组体素或顶点来实现(图2B-C;图3B-C),也可以通过多边形来实现。多边形大脑可视化是简单的二维或三维形状,图形化地表示大脑或特定结构,但不携带任何关于空间坐标的附加信息,仅大致估计大脑及其结构的形状。每个二维(图2D、F)和三维(图2E;图3E)多边形可视化区域都填充有表示区域标签或统计值的颜色。
边。 在神经影像学的上下文中,边通常表示两个大脑区域之间的物理或统计连接,可以被可视化为连接网络中两个节点(大脑区域)的直线或曲线。一种常见的数据组织方式用于边级可视化的是矩阵,通常称为“连接性”或“邻接”矩阵,该矩阵索引大脑区域对之间连接的存在和强度。可视化工具通常将这些矩阵转换为网络图,其中顶点代表大脑区域,边代表这些区域之间的连接,并可以以各种方式显示,例如覆盖在大脑的二维或三维表示上(图2I;图3F),边以连接顶点的直线形式表示。通常,边和节点的视觉属性也可以调整以传达信息,例如根据连接的强度或数量进行大小或颜色编码。
流线。 虽然与边类似,流线专门用于表示白质纤维,通常是进行束流追踪以扩散加权MRI的输出。流线通常被可视化为连接大脑不同点的弯曲三维线,可以用于创建特定白质束或区域之间所有流线的三维可视化。它们可以根据纤维的方向或束的属性(如髓鞘化)进行颜色编码。这些可视化可以覆盖在体素级或顶点级的解剖大脑表示上,以提供解剖参考点。
以上两个图提供了使用R(图2)和Python(图3)中的开源、文档详尽且对初学者友好的包生成的体素、顶点、ROI和边级可视化示例。这些并不是R和Python中用于可视化大脑数据的包的详尽表示(见表1)。相反,这些图旨在让读者了解众多可用选项,并作为选择所需大脑可视化类型的切入点(另见第4节)。所有用于编制这些图的代码以及每个面板的内容均提供在附带的在线存储库中。
3.3. 确定输入文件格式
神经影像数据及其衍生物有许多不同的文件格式,而基于代码的可视化软件包通常针对特定格式进行设计。表 1 提供了一个关键列表,列出了每个软件包可以使用的输入数据类型。以下简要描述了一些用于可视化的最常见的 MRI 文件格式:
纯文本格式。 最简单的输入格式是标量、向量和矩阵,分别表示单个数据点、一维数据数组(例如单列或单行)和二维数据数组(例如多列或多行)。这些数据通常存储在纯文本格式中,如 .txt、.csv 和 .tsv,并且可以由诸如 SPM、FSL 或 FreeSurfer 等神经影像分析软件生成。这些文件还可以包含区域名称和/或空间坐标的行和列。所有编程语言都有函数可以将这些纯文本格式读入编码环境中。这些格式通常用于区域级可视化,其中一组体素或顶点共享相同的值或颜色(图2B-F;图3B-E),或用于边级别可视化,其中矩阵用于识别由边连接的区域(图2F;图3F)。
NIfTI。 NIfTI(.nii)文件存储三维或四维图像数据,通常是体素强度的矩阵或用于四维数据(如功能性 MRI 和扩散 MRI)的三维矩阵序列。图像数据以体素强度的三维矩阵形式存储,头文件包含附加信息,如图像维度和体素大小。诸如受试者人口统计学信息和扫描仪参数等附加信息也可以以元数据的形式存储在头文件中。
GIfTI。 GIFTI(.gii)是 NIFTI 格式的扩展,以基于表面的格式存储数据,表现为一组定义表面网格的顶点、边和面。该格式还支持在表面网格上存储诸如曲率、厚度和功能活动图等数据。通常,.gii 文件名会有一个前缀指示文件包含的信息类型,例如 .surf.gii,仅包含定义表面网格的顶点、边和面;或 .func.gii 文件,包含每个顶点的数据值,这些数据值本质上是数据数组,其索引对应于一个表面文件,并需要一个相应的表面文件来确定在大脑中的数据值分配位置。GIFTI 文件还可以在单个文件中存储多个表面,并且可以包含有关表面拓扑的信息,如顶点、边和面的数量。附加的元数据也可以存储在头文件中。
CIfTI。 CIFTI(.cii)文件可以存储基于表面和基于体积(volume)的神经影像分析数据,结合了 NIFTI 和 GIFTI 文件的特点。对于基于表面的数据,文件包含顶点坐标以及每个顶点的数据值。对于基于体积的数据,文件包含体素强度的三维矩阵。通常,这种数据类型用于将皮层表示为顶点,而将下皮层、脑干和小脑结构表示为体素。CIFTI 文件可以分为三种主要类型:.dtseries(存储时间序列数据,例如功能性 MRI 数据)、.dtscalar(存储标量数据,例如厚度或曲率图)和 .dtlabel(存储标签数据,例如大脑的分区)。这些文件还可以包含额外的元数据字段,如表面和体积配准信息。
FreeSurfer。 FreeSurfer 是一种常用的图像处理和分析软件,配备了多种专有格式用于存储输出。主要格式是 .mgh 及其压缩版本 .mgz,这些格式与 .nii文件类似,通常用于存储体素级数据。顶点级数据存储在多种格式中,如 .pial、.white 和 .inflated。FreeSurfer 还使用 .label 文件存储每个大脑结构的顶点列表及其相关标签,以及 .annot 文件存储注释信息,如顶点、标签和颜色信息,这些信息可以覆盖在表面重建上。
束流追踪(Tractography)。 常用的束流追踪文件格式包括由 TrackVis 和 MRtrix 软件包分别开发的 .trk 和 .tck。这两种格式都以一系列三维点的形式存储流线的坐标,每个点由其 x、y 和 z 坐标表示,以及包含流线中点数、流线属性(例如平均扩散率、各向异性分数)以及流线起始和结束索引的束头信息。
关键点: 这些数据类型中的许多是可互换的,可以相互转换。转换格式的方法有很多,例如,Connectome Workbench 和相关的 R 包如 ciftitools 允许在 NIFTI、CIFTI 和 GIFTI 格式之间进行转换。FreeSurfer 的函数如 mri_convert、Python 中的 NiBabel 库以及 R 中的 fsbrain 都允许在专有格式与开源的 NIFTI、CIFTI 和 GIFTI 格式之间进行转换。因此,如果某个文件格式与可视化软件包、库或工具箱不兼容,用户可以将数据转换为所需的格式。不过,在转换数据结构后,重要的是要可视化和验证数据,以确保数据未被误解,并且还需注意在二维表面和三维体积格式之间映射可能产生的意外后果。
3.4. 选择软件包、库或工具箱
前面的步骤,包括决定编程环境、可视化类型和输入数据类型,将帮助用户选择合适的软件包、库或工具箱。表 1 提供了 R、Python 和 MATLAB 中工具的列表,这些工具按是否能够生成基于体素、顶点、ROI、边或流线的可视化进行分类。虽然每个工具都可能具备生成基于代码的可视化的能力,但某些工具对初学者更友好且文档更完善。通常,这些工具是专门为大脑数据可视化设计的,而不是那些旨在进行大脑数据分析但仅提供有限可视化功能的工具。一些文档详尽且对初学者友好的工具示例通过代码模板生成器提供(见第 4 节)。
选择工具时一个重要的考虑因素是它是否能够生成适合发表的图表。适合发表的图表具有高分辨率、带有标签、包含所有颜色条和图例,并且不需要额外的手动图像处理。虽然表 1 中列出的所有工具都具备这些特性中的一部分,但某些工具能够更精确地控制适合发表的特性,如图例位置、颜色条位置、注释、标签和多面板图形。这些工具通常依赖于主流的通用绘图引擎,如 R 中的 ggplot 和 Python 中的 matplotlib 来生成可视化。这使得用户能够利用许多额外的功能,使其大脑可视化适合发表。例如,R 中的 ggseg 和 ggsed3d 包分别生成与广泛使用的图形语法(即 ggplot2)和 plotly 引擎兼容的图表。类似地,Python 中的 Nilearn 库允许使用 matplotlib 或 plotly 引擎生成图表。我们注意到,表 1 中列出的许多其他工具也依赖于常见的绘图引擎来生成可视化。
3.5. 生成可视化
流行的集成开发环境(IDE),如 RStudio、Visual Studio 和 Spyder,内置了在代码执行时显示和更新图形的能力。生成的可视化图形可以通过多种不同的方式进行共享或嵌入到论文中,具有不同的可复制性水平(见第 2.3 节)和视觉质量。共享可视化图形的一种常见方式是将其导出为图像栅格格式,如 .png、.tiff 或 .jpeg,其中图像以像素网格的形式出现,网格中的每个像素包含该图像特定点的颜色和强度信息。这些格式依赖于分辨率,放大后会变得像素化并且难以解析。而矢量格式,如 .svg、.eps 和 .pdf,可以在不丢失质量的情况下放大或缩小。虽然所有编码环境都提供将可视化图形导出为栅格格式的方法,但使用矢量格式进行导出虽然在视觉上更优,但取决于具体工具。通常,只有依赖于主流通用绘图引擎(如 R 中的 ggplot 和 Python 中的 matplotlib)的可视化工具才能将图像导出为真正的矢量格式。
越来越流行的软件,如 R Markdown、Quarto、Jupyter Notebook 和 Google Collab,可以创建将代码、文本和可视化图形组合在单个文件中的动态文档。这使得记录工作流程和共享完整分析变得更容易,增强了协作和可复制性。在包含生成图形和其他结果的代码以及格式良好的基于文本的解释(见第 2.3 节)的同时,用户和其他人可以准确地复制工作。这些工具还提供了广泛的文档输出格式,包括 PDF、HTML、Word 和 LaTeX。这种多功能性使得可以从单一源文件生成精炼的报告、演示文稿、手稿,甚至是互动仪表板。目前,虽然 R Markdown 主要与 R 相关联,Google Collab 与 Python 相关联,但 Jupyter Notebook 和 Quarto 支持更广泛的编程语言,包括 Python、Julia、R 等。
3.6. 分享可视化代码
使用代码生成的图像可以插入到手稿中,并且相关代码可以包含在补充材料中。虽然这是共享可视化和代码的最简单方式,但它可能不像将代码上传到专用的代码共享和版本控制平台(如 GitHub 或 GitLab)那样易于访问。这些服务目前被广泛使用,并允许代码保持格式、版本控制和搜索功能。
另一个选择是广泛使用的通用研究数据仓库,如 Open Science Framework、Zenodo 或 FigShare,这些平台使研究人员能够免费发布和共享他们的数据集、软件、代码和其他研究成果。这些平台的一个优势是它们可以为共享材料分配数字对象标识符(DOI),使其能够独立引用并增强可见性,因为 DOI 被各种仓库和搜索引擎索引。一些平台甚至提供使用度指标,使用户能够了解代码和材料被访问、引用或重用的频率,这对于评估工作的影响力和覆盖范围是有价值的信息。
在编写和共享用于图形生成的代码时,一个重要的考虑因素是代码的长期保存和对软件崩溃的抵抗力。虽然关于代码和软件保存的讨论超出了本文的范围,读者可参考本文中的相关引文,以及旨在保存和归档全球所有软件源代码的项目,如 Software Heritage,确保有价值的软件源代码不会随着时间的推移而丢失。除了所使用的平台外,代码对用户和他人的可访问性还取决于代码的编写、格式化和注释的清晰程度。我们建议读者参考其他实用指南 [引文24,25]。
虽然共享代码对于实现可重复性是必要的,但通常并不充分,因为可视化所基于的底层数据源可能是代码正确运行所需的。虽然上述许多共享平台也可以与代码一起托管源数据,但现在已经存在专门的平台,如 OpenNeuro 和 NeuroVault,允许共享神经影像特定的数据集,如包含 NIfTI 和 CIFTI 图像的数据集。如果无法共享可视化的源数据,也可以生成并提供合成数据与代码一起使用。
4. Brain-Code:用于生成脑部可视化代码模板的网页应用
为了帮助研究人员过渡到生成基于代码的脑部可视化,我们开发了一个网页应用(https://sidchop.shinyapps.io/braincode/),该应用可以交互式地为 R 和 Python 中对初学者友好的库/包生成代码模板。在网页应用中,用户可以选择 R 或 Python 作为他们的编程环境,并选择体素级、ROI(感兴趣区域)级、顶点级和边级的可视化类型。然后,他们可以手动调整有限的可视化设置,例如颜色尺度和视图,并获得一个响应式代码模板,该模板可以复制并在各自的编程环境中使用。提供的代码模板需要用户自定义代码,例如更改文件路径。可用的设置已被有意限制,以便用户在自己的编程环境中探索和微调其他可视化选项。代码模板还包含链接和提示,指向更详细的文档、替代的软件包/库和教程,这些都允许创建更复杂且适合发表的脑部可视化。用户可以通过 GitHub 仓库下载打包版本的网页应用。

图4. BrainCode网络应用的界面,用于生成基于代码的简单脑可视化模板。
(1) 选择编程环境(R或Python)。
(2) 选择可视化类型(体素、区域、顶点和边)。还有一个常见问题(FAQ)标签页来帮助用户。
(3) 手动调整有限的可视化设置,并观察它如何实时改变可视化效果(4)和代码模板(5)。
(4) 可视化预览区域。
(5) 代码模板区域。将代码模板复制到所选的编程环境中,更改数据的文件路径,并探索函数提供的其他可视化设置。
5. 局限性与功能缺口
虽然许多基于代码的工具有详细的文档,并且不需要深入的编程知识,但对于新用户来说,相比使用 GUI,仍然存在较陡峭的学习曲线。这在创建适合发表的图形时尤为明显,因为可能需要对视觉特征(如图例位置、字体大小和多面板图形布局)进行细致调整。尽管大多数基于代码的工具在这些细节步骤上提供了一定的控制,但在功能可用性和可用性方面它们之间存在差异,使用如 ggplot2、matplotlib 和 plotly 等成熟图形引擎的工具提供了最灵活且文档最完善的辅助可视化功能。相关地,虽然一些交互式图像查看器可以在集成开发环境(如 R-Studio)中打开(例如,Muschelli, 2016),但对于快速和交互式查看单一 GUI 工具来说,使用这些工具可能更快捷和实用。
大脑小脑和脑干区域在可视化软件中往往表现不佳(例如,图 2-3),这可能反映了在人类神经影像研究中长期存在的以皮层为中心的观点。同样,自定义的非皮层图集,如非标准的下皮层图集方案,尚不直观,通常需要多个函数和包来进行可视化。这些结构的可视化通常需要将多个基于 GUI 的工具串联起来(见 Madan, 2015)。或者,用户可以将神经影像特定的文件格式转换为通用的可视化或多边形格式,如 .vtk、.ply 或 .obj,这些格式可以使用通用的基于代码的工具进行读取、操作和可视化。这类工具的例子包括 Python 中的 PyVista 和 Mayavi,以及 R 中的 rayshader 和 plotly。此外,一些神经影像衍生的数据类型,如基于 DWI 的束流追踪结果的流线,仍未在基于代码的可视化工具中得到很好的支持,未来的发展应着重于增强这些数据类型的可视化能力。
如表 1所示,在每个编程环境中通常有多个包可以可视化每种数据类型。虽然这为高级用户提供了选择,但也可能导致初学者感到困惑,因为他们可能不熟悉工具之间的细微差异。虽然我们概述的流程、以及我们提供的表格和网页应用将帮助用户选择理想的软件包,但未来的工作应继续将脑部可视化方法整合为统一的、对初学者友好的基于代码的工具,这些工具依赖于成熟且文档完善的图形引擎,并且能够绘制多种数据类型。























 3897
3897

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








