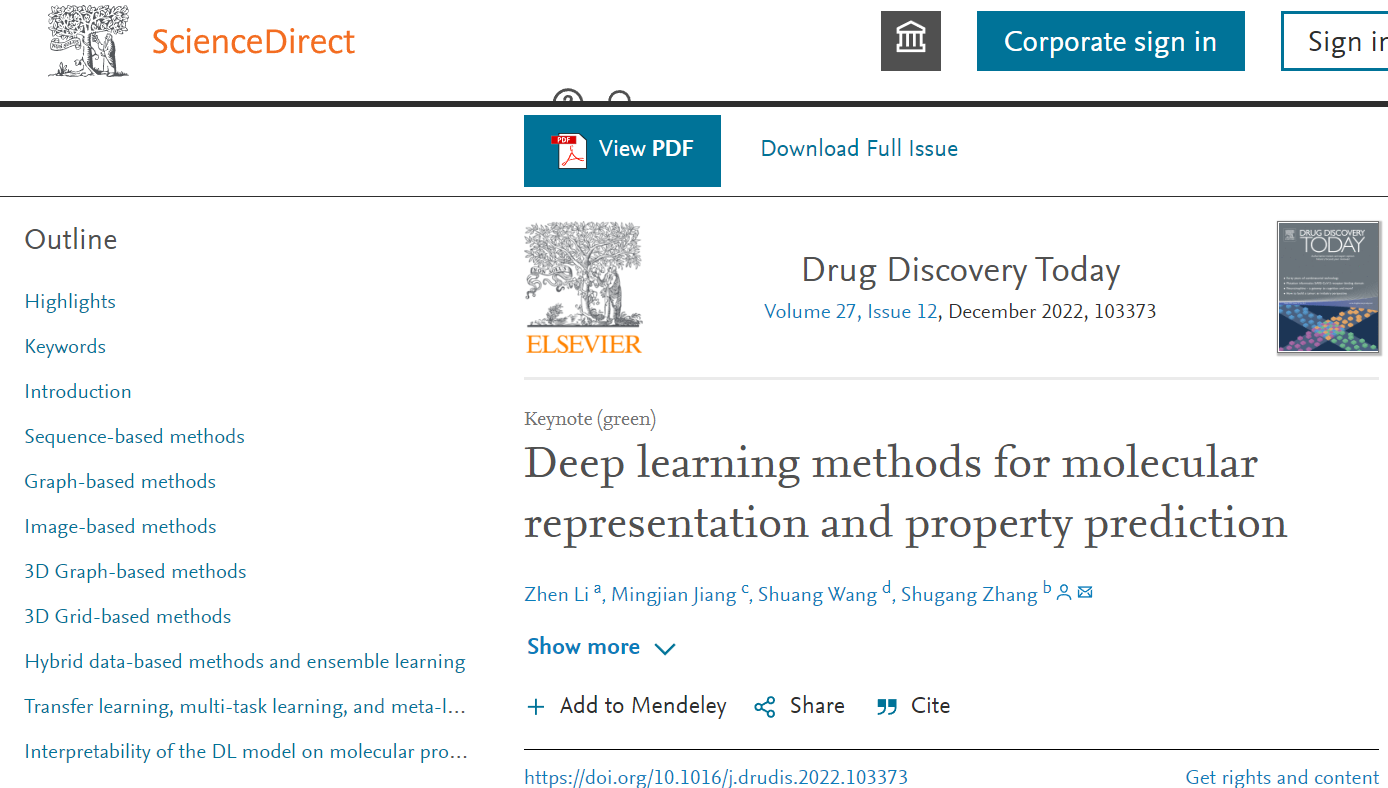
一、分子表征
分子表征形式:
1D:SMILES、Fingerprints (ECFP)、molecular access system (MACCS)、mathematical representation
SMILES:字符串虽然简单、快速,但仍然不能全面捕捉原子之间的空间关系。如图1所示,在C = C5苯环上,5个碳原子被分组为C5;然而,不同的碳原子与其他原子有不同的关系,并且位于分子中不同的位置。此外,它们可能对应不同的属性。因此,仅使用SMILES是不足以预测某些属性的。
2D:graph、image,提供结构信息
graph:有效方法。原子被看作是分子图中的节点,而化学键被看作是边。随着图卷积网络(GCNs)的发展,可以更直接和有效地收集相邻节点的信息,这对于捕获分子内原子之间的空间关系非常有用。
image:基于像素的栅格化图像是分子的另一种二维表示格式,图像中的每个像素代表一个键、原子或空白背景。
3D: 3D graph 、3D grid,提供了关于分子的最详细的信息
3D graphs :记录每个原子的三维位置
3D grid:特殊的三维图像,网格中的体素通过不同的方法表示分子构象的不同元素或属性。
二、模型
1、Sequence-based methods
Data augmentation methods:
根据SMILES语法可以有许多有效的SMILES序列。起始原子和遍历顺序可能对应于一个序列,规范的SMILES保证每个分子根据一定的规则只有一个SMILES串。然而,当非规范smiles作为输入时,同一分子的不同smile格式可以增强DL模型的学习能力。这是因为通过提供与smile语法和化学性质相关的潜在特征,非规范smile也可以使DL模型受益。因此,建议通过数据扩充或枚举来扩大字符串的覆盖范围,从而保证模型能够学习到一个分子的多个字符串。
考虑到每个分子的长度不同,短SMILES可能比长SMILES要少。为了克服数据集不平衡的问题,Smiles字符串较少的分子被重复的SMILES字符串补充,以确保所有分子都有相同数量的SMILES字符串。【Maxsmi: Maximizing molecular property prediction performance with confidence estimation using SMILES augmentation and deep learning】对五种不同的SMILES增强方法进行了综合分析发现,增强方法提高了DL模型的性能,并且使用规范Smiles得到的结果优于使用单一随机Smiles。
Convolutional neural network models:
CNNs可用于序列数据处理。【Different molecular enumeration influences in deep learning: an example using aqueous solubility (2021)】将SMILES转换为一个整数列表,然后添加positional embedding以对应字母的位置。基于cnn的方法需要固定长度的输入样本,SMILES必须被填充或截断。通常,可以选择数据集中SMILES字符串的最大长度或平均长度作为模型输入样本的固定长度。然而,这两种方法都会导致数据丢失和噪声的引入,这是基于cnn的方法的主要问题之一。
Recurrent neural network models:
SMILES序列是提取分子特征的关键。Bi-LSTM具有贝叶斯优化改进的通道和空间注意网络,能够明确识别smile序列中的主要因素。【Dielectric polymer property prediction using recurrent neural networks with optimizations(2021)】提出了RNN的两种反向传播方法,并比较了smile的二进制和十进制表示在聚合物性能预测中的性能。发现二进制表示比十进制表示更准确。CNN和RNN的结合也可以提高表示的性能。【A novel molecular representation learning for molecular property prediction with a multiple SMILES-based augmentation(2022)】使用one-hot编码将SMILES中的每个字符转换为一个向量,并引入一种堆叠CNN和RNN层的混合体系结构进行表示提取。据报道,与普通RNN模型相比,CNN能够提高预测性能。
尽管RNN适合于序列处理,但仅使用序列而忽略其他信息(如化学上下文或分子结构)并不是学习分子表示的全面方法。特别是,分子内的原子关系、原子基团以及键类型也可能与分子性质有关,可以通过某种方式引入分子性质,以提高分子性能。
此外,利用序列的模型的可解释性还存在缺陷。由于分子分支与主序列融合,如果没有其他更具体的设置,RNN模型很难区分基序和分支;因此,来自同一官能团的关键原子可能位于彼此很远的地方。即使引入了注意机制,它也只关注单个字母或相邻字母。
Substructure learning methods:
官能团是分子的关键部分,分子性质和活性与官能团和亚结构高度相关。然而,SMILES序列并不直接包含这类信息;因此,已经开发出着重于隐藏在SMILES中的官能团的方法。【SMILES pair encoding: a data-driven substructure tokenization algorithm for deep learning(2021) 】学习一个由高频SMILES子串组成的词汇表,并根据学习到的词汇表对SMILES进行转换,将其输入DL模型。【Mol2Context-vec: learning molecular representation from context awareness for drug discovery (2021)】在ECFP的帮助下提取子结构。该子结构包括多个原子,包括一个中心原子和围绕中心原子的给定半径内的所有原子。每个子结构都有自己的标识符。子结构序列是BiLSTM的输入,它捕获原子组之间的相互作用。
【Mol-BERT: an effective molecular representation with BERT for molecular property prediction (2021)】将每个子结构的向量总结为无监督学习和下游任务的分子表示。【S2DV: converting SMILES to a drug vector for predicting the activity of anti-HBV small molecules(2022)】定义了一个分割字符来保留子结构信息,并定义了一个具有预定义大小的滑动窗口来处理序列。分支链和指示单个原子的双字符替换为单个字符,并且以相同的方式展开和处理每个分支。
Sequence-based self-supervised learning methods:
基于序列的SSL模型可以分为对比学习方法和生成学习方法,对比学习是通过构造伪标签数据来学习正样本和负样本之间的差异,生成是将输入编码为潜在特征并解码重建输入,从而将潜在特征作为输入的表示。
MOL-BERT结合了三个任务来生成分子表示(BERT中MLM;SMILES等价法,将来自相同分子的两个SMILES序列作为一类,将来自不同分子的两个SMILES序列作为另一类进行训练 ;利用分子化学特性进行预测)。
SMILES-BERT也是基于BERT的,但是只用MLM,并且引入了一个自注意层来使用顺序信息。
【A deep learning-based chemical system for QSAR prediction(2020)】:使用GRU-base的encoder-decoder生成模型生成固定大小的潜在特征来表示SMILES中的分子,并引入CNN模型用于下游预测任务。
【Inductive transfer learning for molecular activity prediction:next-gen QSAR models with MolPMoFiT, 2020 J Cheminform】:利用语言模型根据单词序列预测下一个单词,还可为下游任务提取特征。
【Learning continuous and data-driven molecular descriptors by translating equivalent chemical representations】:提出了一种翻译分子结构的两种语义等价表示形式(即SMILES和IUPAC名称)的方法。SMILES使用seq2seq方法时,主要目标是找到两个相应的序列进行训练。
【Transformer-CNN: Swiss knife for QSAR modeling and interpretation, 2020 J Cheminform】:训练一个Transformer模型执行SMILES规范化任务,其中输入为非正则SMILES,输出为相应的规范化SMILES。

2、Graph-based methods
图是一种更直接的结构,可以存储和表示大多数结构信息。GCNs:能够捕获有关连接节点之间关系的信息,利用分子内部的结构信息。一般有两种类型的GCNs:空间卷积(spatial convolution)和光谱卷积(spectral convolution)。空间卷积通过一定的空间域消息传递规则来收集相邻节点的信息,从而更新每个节点的特征。光谱卷积通过对拉普拉斯矩阵进行特征值分解,将图数据转化为谱域。
Spectral GCN models:
【Lanczosnet: multi-scale deep graph convolutional networks(2019)】:使用Lanczos算法构建了图光谱卷积的拉普拉斯低秩近似,可用于挖掘多尺度信息和设计可学习的光谱滤波器。
【Multi-view spectral graph convolution with consistent edge attention for molecular modeling,
Neurocomputing(2021)】:从Chebyshev近似出发,用一种新的柔性光谱滤波器提出了一种一致的边缘感知多视角光谱GCN模型;根据边缘的类型将分子图分解为图的多个视图,并采用一种学习边缘注意权重的一致边缘映射方法来保证边缘一致性。
在分子表征和性质预测方面,光谱方法比空间方法少。这是因为具有不同原子的分子会产生不同大小的图,而光谱GCN模型只能处理固定大小的图。因此,在处理输入图数据样本时,仍然需要进行数据对齐操作,如填充或截断,这将损害数据的完整性,影响模型的最终性能。
Spatial GCN models:
Spatial GCN模型在药物发现和MPP中得到了广泛的应用。通常,Spatial GCN需要两个矩阵作为输入:一个相邻矩阵(表示分子内原子在空间上的相互联系)和一个特征矩阵(后者通常有不同的定义方法)。【DeepAtomicCharge: a new graph convolutional network–based architecture for accurate prediction of atomic charges(2021)】:使用带有跳跃式连接的消息传递神经网络(MPNN)来预测原子电荷。
【Pushing the boundaries of molecular representation for drug discovery with the graph attention mechanism(2019)】:是由GCN衍生而来的另一种分子表示方法,它通过注意机制自动学习非局部的分子内相互作用,并捕获指定任务的隐藏边缘。
【Multiphysical graph neural network (MP-GNN) for COVID-19 drug design(2022)】:结合了尺度特定的图神经网络和元素特定的图神经网络,以捕获来自不同尺度的各种原子相互作用,以实现多物理表示。
【Cross-dependent graph neural networks for molecular property prediction(2022)】:认为原子和化学键同样重要。构造了原子中心视图和键中心视图,并在这两个视图之间提出了一种交叉依赖的消息传递方案。
【TrimNet: learning molecular representation from triplet messages for biomedicine(2021)】:提出了一个三重注意边缘网络,通过原子-键-原子排列来收集信息,以提高边缘信息的提取。一对原子和它们之间的键被连接成一个三元组,多头注意被用来从相邻的节点和边缘收集节点的信息。
有向图是一种特殊类型的图,其中包含指示消息传递方向的有向边。这种性质常用于处理图学习模型中经常出现的超平滑问题。
【Building attention and edge message passing neural networks for bioactivity and physical–chemical property prediction(2020)】:专注于传递边的消息,而不是节点。每条边有两个状态,对应两个相反的方向;因此,基于其上游节点每个状态都会被更新,从而避免了超平滑问题带来的有用信息的丢失。
Tree-based methods:
通过选择一个起始原子,可以将图转换为树形结构,这将打破图的圆圈,并提供图的另一个视图。此外,根据广度优先搜索(BFS)或深度优先搜索(DFS)方法,可以使用RNN模型将树转换为原子序列进行表示学习。
【An architecture of deep learning in QSPR modeling for the prediction of critical properties using molecular signatures(2019)】和【Predictive deep learning models for environmental properties: the direct calculation of octanol–water partition coefficients from molecular graphs(2019)】都开发了基于分子树结构的QSAR建模方法。分子被编码为特征描述符,树结构长短期记忆(tree- lstm)用于描述分子树数据结构,并将它们与分子性质关联起来,树结构长短期记忆(tree- lstm)善于捕获长期依赖关系。
【Junction tree variational autoencoder for molecular graph generation(2018)】首先将分子分解成子结构,然后根据这些子结构生成树状图。虽然连接树被提出用于分子生成,但编码器部分可以被隔离用于性质预测。受此启发,【Molecular property prediction based on a multichannel substructure graph(2020)】提出了一种基于多通道树的分子预测方法。将分子转化为基于子结构的图,并应用BFS方法遍历图生成树形结构。然后,利用具有注意机制的基于GRU的神经网络在多个层次上学习分子特征。
然而,由于不同的遍历方法(如BFS或DFS)和不同的根原子选择方法,树结构不是唯一的。生成的分子树的多结构会影响模型的泛化。规范结构的定义不是健壮的,并且不能确保所有与属性相关的信息,特别是连接信息,在结构中得到全面的显示,因为将图传输到树将不得不分解一个或多个连接
Graph-based self-supervised learning methods
与序列数据的性能相似,SSL方法在图数据上也取得了显著的性能。
【Self-supervised learning on graphs: contrastive,generative, or predictive(2021)】在图数据上定义了另一种类型的SSL方法,即预测方法,该方法基于自生成标签设计基于预测的pre-task。
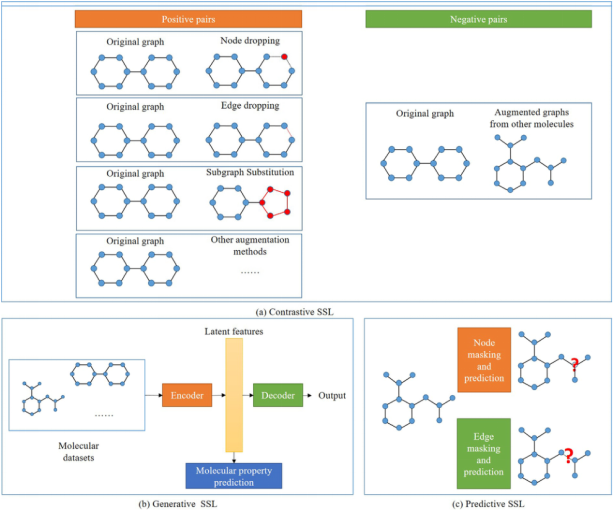
在对比学习方法中,如何构建正样本和负样本至关重要。在这方面,【MolCLR: molecular contrastive learning of representations via graph neural networks(2021)】通过三种方式增强分子:原子掩蔽、键删除和子图删除。同一分子的样本记为正对,其他的记为负对,进行对比损失计算。但【MoCL: data-driven molecular fingerprint via knowledge-aware contrastive learning from molecular graph】认为,节点下降、边缘扰动、子图提取等增强方法会影响分子的性质,不适合进行对比学习。MOCL采用了子结构替代策略,即一个子结构被具有相似属性的生物等体所取代。在自然语言处理领域,许多特征提取在单词和句子级别。NLP中的这两个层次类似于图中的节点层和图层,分别代表局部特征和全局特征。【An effective self-supervised framework for learning expressive molecular global representations to drug discovery(2021)】:在节点和图级别对模型进行了预训练。在图的层面上,每个分子被分解成两个部分,模型需要预测这两个部分是否来自同一个分子。分子图对比学习方法的过程如图a所示。
生成方法通过编码器-解码器模型重构输入。【MG-BERT: leveraging unsupervised atomic representation learning for molecular property prediction(2021)】将局部消息传递和GNN结合到BERT模型中进行预训练。 【Embedding of molecular structure using molecular hypergraph variational autoencoder with metric learning(2021)】:使用分子超图语法变分自编码器(VAE)提取分子嵌入,将分子结构和物理性质嵌入到VAE的潜在特征中。基于分子图的生成性学习方法的过程如图b所示。
MPP中通常使用的预测模型用于预测节点或边的类型或属性。【Self-supervised graph transformer on large-scale molecular data(2020)】:结合了两阶段ssl。第一个SSL任务在节点/边缘级别定义,是预测子图的属性。第二个SSL任务,定义在图级别,是预测不同主题的出现;结合这两个层次可以提供分子的结构和语义信息。此外,SSL和监督学习的连接为理解分子提供了新的途径。例如【SUGAR: Subgraph neural network with reinforcement pooling and self-supervised mutual information mechanism(2021)】结合了基于子图的监督和SSL方法,来自分类和互信息最大化的两个损失被分组到最终的损失函数中。评价结果表明,引入SSL后,模型性能得到了改善。基于分子图的预测学习方法过程如图c所示。
3、Image-based methods
基于图像生成的方法,最简单的方法是直接使用分子图像,这些图像可以通过软件进行转换,如RDKit和Open Babel,然而,这种类型的图像引入了大量空白区域,没有有效信息
缩放敏感度也是一个问题,因为所有的分子都被转换成相同大小的图像。换句话说,同一原子/结构的大小在不同分子中是振动的,因为整个分子图像的大小是固定的。
【Prediction of molecular properties using molecular topographic map(2021)】为每个原子生成一张图,并将所有原子图相加形成分子topographic图,即28*28 heat map,
【MolMap:Out-of-the-box deep learning prediction of pharmaceutical properties by broadly learned knowledge-based molecular representations, Nat Mach Intell 3 (2021)】 将分子描述符和指纹特征映射到2D特征映射中,从而形成了一种将手工制作的特征组合到2D空间中的方法,以捕获分子特征的内在相关性。
基于图像的方法不是MPP的主流方法,因为它们需要将数据样本转换到欧氏空间,由于缺乏原子和键属性,不适合分子性质的预测(molImage是基于图像的分子性质预测模型,nature machine intelligence 2022文章 ,效果是很显著的)。然而,近年来,基于图像的DL模型已经被提出和发展起来。为了利用基于图像的DL模型,能够从某些角度发现原子间关系的图像生成方法值得进一步研究,这将有助于生成新的有效分子表示。
4、3D Graph-based methods
分子的构象通常包含分子的原子三维坐标,可以为MPP提供额外的空间信息。首先要解决的问题是三维分子构象数据集有限,与一维和二维的方法相比,专注于基于几何的数据的方法仍然相对较少。与二维数据类似,基于几何的方法也分为两类:基于3D-graph和基于三维网格的方法。
基于3D Graph-based methods的MPP方法和基于3D Grid-based methods的MPP方法仍然缺乏,需要较长的分析时间是一个严重的问题(计算量太大)。
1)3D Graph-based methods:
通过引入几何信息来调整或改进GCN。除了相邻矩阵和特征矩阵外,3D-GCN引入了由原子间三维位置组成的相对位置矩阵,以保证卷积过程中的平移不变性。结果表明,训练后的模型具有旋转随机性的内在特征,无需额外的增强方法。
【Molecular property prediction: a multilevel quantum interactions modeling perspective(2019)】:设计了一种从分子的构象和空间信息捕获多级量子相互作用的GCN。模型中依次涉及相邻节点的不同阶数,以确保节点表示能够涵盖高阶交互。然而,与社交媒体和引文网络图等其他大型图数据不同,分子图中的节点数量是有限的。在某些情况下,高阶近邻可能会覆盖分子中的大量原子,从而导致超平滑
【Spherical message passing for 3D graph networks, arXiv 2021】:为了解决三维图形中消息传递的问题,每个原子的3D坐标被转换为球坐标系,原点节点的邻近节点由一个由边长、边间角和扭转角组成的3元组指定。与普通的三维坐标相比,三元组更加灵活,能够更准确地描述分子的结构。球形消息传递对输入分子的平移和旋转是不变的。
【GemNet: Universal directional graph neural networks for molecules(2021)】:使用了分子的球形表示,以确保模型对平移不变性,对排列和旋转等变。
【Geometry-enhanced molecular representation learning for property prediction(2022)】:提出了一种充分利用分子几何信息的自监督框架。构建了一个新的键角图,在这个图中,分子中的化学键被视为节点而不是边,而两个键之间形成的角度被视为它们之间的边。对某些原子的一跳邻域项进行屏蔽,预测键长和键角,提取局部表示。
【Pre-training molecular graph representation with 3D geometry(2021)】:SSL方法,其中包含分子的3D和2D视图之间的对比学习和生成学习方法。在对比学习中,将同一分子的三维图和二维图都作为正对来训练模型。在生成学习中,训练模型从二维拓扑生成三维构象。
2)3D Grid-based methods:
将每个原子放在网格的一个或多个体素中,像蛋白质这样的大分子可以用3D网格更好地表示,但这样的数据在MPP中仍然表现良好,特别是在一些量子力学性质方面。3D CNN是3D网格数据的最佳选择;因此,一个更强大、信息量更大的三维CNN网格可以提高MPP的性能。
【Libmolgrid: graphics processing unit accelerated molecular gridding for deep learning applications(2020)】:提供了一个生成三维分子数据体素网格的库来表示分子。网格分辨率是影响计算结果的一个重要因素。
【Multiresolution 3D-DenseNet for chemical shift prediction in NMR crystallography(2019)】:采用原子中心高斯密度模型创建三维网格,生成不同尺度的多通道网格(4-14 Å)作为3D- densenet处理和预测的输入数据。
【Prediction of energetic material properties from electronic structure using 3D convolutional neural networks,(2020)】:使用高斯模型生成了两个三维空间点网格(电子电荷密度和静电势)。采用了旋转增强的方法来放大训练数据集,而不是改变数据集的大小
【DeepNose: using artificial neural networks to represent the space of odorants, Proc Machine Learn Res 97 (2019)】:选择C, H, O, N, S, Cl作为3D网格的独立通道,并使用基于cnn的自编码器进行分子表示。
【3D molecular representations based on the wave transform for convolutional neural networks (2018)】:发现分子的常规三维网格过于稀疏,影响了CNN的性能,提出了波变换平滑方法来填充每个原子的相邻体素。
5、Hybrid data-based methods and ensemble learning
所有1D、2D和3D表示方法都以不同的方式呈现分子,结合它们可以提供一个分子的多视图。
1)Hybrid data-based methods
【GraSeq: graph and sequence fusion learning for molecular property prediction2020】:结合分子图和SMILES序列,使用GCN和BiLSTM进行编码。
【Quantitative toxicity prediction via meta ensembling of multitask deep learning models(2021) 】:将smile、指纹、分子图、2D和3D描述符与多个DL模型相结合,用于定量毒性预测。
通常,指纹被认为是一种流行的辅助因子,基于image的【Learning drug functions from chemical structures with convolutional neural networks and random forests(2019)】和基于graph的【DeepHIT: a deep learning framework for prediction of hERG-induced cardiotoxicity(2020) 】都将指纹结合起来提高了预测性能。
2)ensemble learning
将多个分类器连接起来,以提高联合模型相对于单个分类器的性能。
【On graph neural network ensembles for large-scale molecular property(2021)】:建立了三个gin的集合。
【Calibrated uncertainty for molecular property prediction using ensembles of message passing neural networks(2021)】:构建了多个mpnn的集合,这些mpnn使用随机参数初始化,并在同一数据集上单独训练。此外,使用所有分类器的方差来表示预测的不确定性,校正结果提高了模型的性能。
【Toxicity prediction by multimodal deep learning(2019)】:将SMILES字符串转换为一个one-hot vector,表示每个字符的缺失。以分子图像和二维数字特征为输入,分别对这三类数据进行RNN、CNN和MLP的训练。这些网络的输出通过集合平均法或元神经网络进行组合。
6、Transfer learning, multi-task learning, and meta-learning
实验数据集的不足是MPP的另一个问题。使用来自其他领域的大数据集来帮助寻找数据较少的目标领域的模式,将有效地克服这个问题。
1)迁移学习
【MRlogP: transfer learning enables accurate logP prediction using small experimental training datasets(2021)】:在大数据集(50万个分子)上对低精度的logP预测值使用迁移学习,并在244个类药物化合物的小数据集上对参数进行微调。
2)多任务学习
同时训练所有任务并共享表示形式,以提高预测的泛化能力。
【Transferable multilevel attention neural network for accurate prediction of quantum chemistry properties via multitask learning(2021)】:使用多任务学习从QM9和Alchemy数据集中预测了12个量子化学性质。此外,在该框架中选择以原子为中心的对称函数作为辅助预测目标,提高了模型的泛化性和可移植性。
3)元学习
为了解决few-shot的问题,也被称为“学习学习”。在训练过程中,元学习将训练数据集划分为不同的元任务,学习初始化良好、泛化能力强的模型参数。通过对新任务进行少量的梯度下降来更新模型,以增强模型的性能。
【Few-shot graph learning for molecular property prediction(2021)】:结合图神经网络、SSL和Tox21中的任务权重感知元学习以及SIDER中的六个任务用于MPP。
【Property-aware relation networks for few-shot molecular property prediction(2021)】:提出了一种考虑不同分子性质和不同分子子结构之间关系的属性感知嵌入方法,以及一种选择性更新任务中的参数的元学习方法,以分别对泛型和属性感知知识建模。
因此,这三种学习方法都能找到不同任务之间的关系,解决数据有限的问题。通常,迁移学习方法可以使用大规模的数据集,如ChEMBL和ZINC来学习分子的通用表示,并对其进行微调以适应特定的数据集。多任务学习通过同时学习几个相关任务来提高模型的泛化能力,而元学习则预测数据有限的看不见的任务,这在MPP领域对于 few-shot问题很有前途,并且可以避免昂贵、耗时和费力的实验数据收集。
7、Interpretability of the DL model on molecular property prediction
可解释的DL方法分为两类:被动的和主动的。被动方法利用DL模型中的参数进行解释,而主动方法通过改变训练过程来提高模型的可解释性。
【Explainability methods for graph convolutional neural networks(2019)】:评价了GCN上三种显著的可解释性方法,并报道显著子图可以解释为官能团。酰胺、三氯甲基、磺胺和芳香族结构都是通过可解释性方法强调的。
【Coloring molecules with explainable artificial intelligence for preclinical relevance assessment(2021)】:提出了一种使用训练过的MPNN模型为每个节点生成重要性评分的特征归因方法,并根据其重要性评分为原子着色。该方法可以识别药效团基序,并识别属性。
在分子性质预测方面,被动方法仍然是用来了解分子精确子结构与其性质之间关系的主要方法。注意机制可以学习输入的不同部分的权重,以确保DL模型能够关注到重要的部分。注意机制的概念也可以用于DL模型的可解释性,它可以找到重要的原子或基团及其对分子性质的相应贡献。
【A self-attention based message passing neural network for predicting molecular lipophilicity and
aqueous solubility(2020)】:通过可视化的自注意值来检测分子的哪些部分有助于其亲油性或水溶性。【Mining toxicity information from large amounts of toxicity data(2021)】:开发了毒性预测的多任务图注意框架。该框架提取了分子子结构的特征,并为每个子结构分配了一个注意权重。检测到最高权重的亚结构,如酰氯、缩氨基脲、亚硝酸盐和亚硝胺(称为结构警报)。
另外,不确定度估计是评价模型可靠性的一种重要方法。
【A Bayesian graph convolutional network for reliable prediction of molecular properties with uncertainty quantification(2019)】:使用贝叶斯GCN预测分子性质。它用“具体的”退出替换了标准退出,并通过贝叶斯方法估计了模型的不确定性,可以作为预测的置信度指标。
【Uncertainty quantification using neural networks for molecular property prediction(2020)】:评估了分子性质预测的许多不确定性估计器,并提出加入多个弱不确定性估计器可以导致更一致的性能。
三、结论
一般来说,高维数据比低维数据包含更多的信息。如果是这样的话,那么基于二维数据的模型的性能应该比基于一维数据的模型更好。然而,混合方法使用了1D、2D和3D数据,消融实验已经证明了它们在MPP中的各自作用,表明低维数据贡献的信息并没有被高维数据完全覆盖。
当高维数据(2D或3D数据)已经被使用时,为什么1D数据仍然有助于模型的性能?
这个问题可以从以下两个方面来回答。首先,在将一维序列转换为二维图形时,可能会丢失一些信息。其次,DL模型不能充分挖掘高维数据中的隐藏信息;因此,一维数据仍然是辅助信息源。
无论原因是什么,如何确定数据的适当类型(或多种类型的最佳组合)仍然是一个悬而未决的问题。
迁移学习、多任务学习和元学习都是用来解决某些特性实验数据不足的问题。认为元学习方法是目前最有前途的研究方向之一。更具体地说,对于实际应用来说,元学习是一种理想的方法,因为对于某些任务(例如,预测一些罕见的分子性质)可能只有少数实例,而传统的ML或DL模型由于数据样本数量有限而无法使用。因此,MPP的元学习方法值得进一步研究。
妥妥的论文天堂(地狱)啊,太强了(好多好多都看不懂啊,慢慢...),论文100+......100+,哭了

























 3107
3107











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








