前几天一个小伙伴问:怎么查看一个基因和某一个通路之间的相关性。这里就简单的提供一个可能的解决办法,供有相同需求的小伙伴参考。
这里我们就假设我们想要查看ACE2和Cell Cycle信号通路之间有没有关系。对于这样的目的,我们第一步肯定是要查询两者在之前的研究当中是否有关系。
1. 确定之前的研究结果
对于之前研究结果的确定的话,我们可以通过genecards来查询。genecards当中总结了这个基因应该参与哪些经典的通路。在genecards里面汇总了KEGG等多个通路数据库当中的信息。所以在这里,我们基本上能清楚这个基因主要是参与哪些基因。
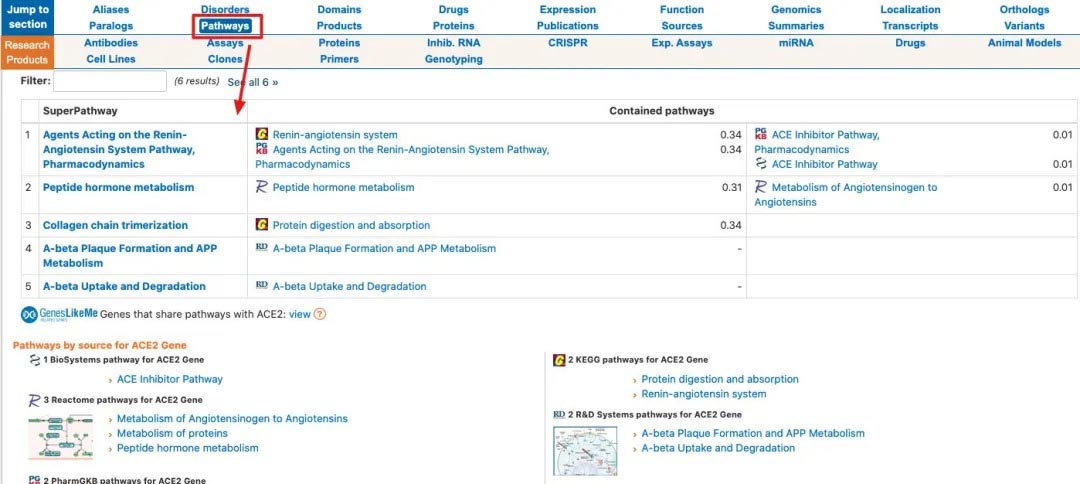
经过查询,我们发现基于这个基因本身的功能,它和Cell Cycle信号通路是没有关系的。
2. 确定基因和信号通路内的基因的关系
经过上面的查询,我们发现这个基因并不参与细胞周期信号通路的调控。那这个基因不参与这个通路并不能代表这个基因和通路内的基因没有关系。因此,我们查看查看这个基因和通路内的基因是否存在关系。那么首先第一步,就是需要知道通路内都有哪些基因。





















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








