如何获取kegg通路的基因列表?相信很多人都有这个疑惑。
哪些时候需要知道通路的基因列表?举两个例子:
- 在找差异基因这一步,只想针对特定通路的基因去找。可以在找完差异基因后,和通路的基因列表取个交集。
- 单细胞分析中,经常会用到基因集打分,除了看文献收集,基因集还能怎么找?可以根据kegg等数据库去找。
今天介绍的这种方法,非常简单,就几行代码:
# BiocManager::install("KEGGREST")
# BiocManager::install("EnrichmentBrowser")
library("KEGGREST")
library("EnrichmentBrowser") #这个包里面的一些函数会调用KEGGREST里面的函数
### download the pathways
hsapathway <- downloadPathways("hsa") #只有在第一次运行这句代码时,耗时较长
### retrieve gene sets
hsa <- getGenesets(org = "hsa", db = "kegg", gene.id.type = "SYMBOL",cache = TRUE, return.type="list") ##只有在第一次运行这句代码时,耗时较长
writeGMT(hsa, gmt.file = "20230205_kegg_hsa.gmt")
之后就能得到gmt格式的基因列表了。
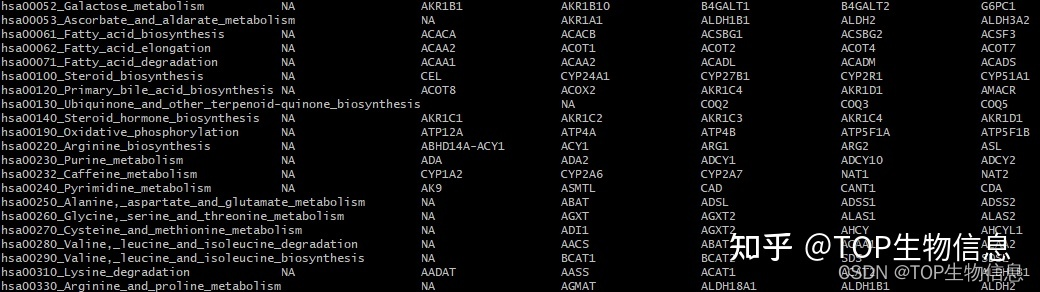
gmt格式也可以很容易地转换为我们熟悉的数据框格式,方便我们做别的分析。
keggdf=clusterProfiler::read.gmt("20230205_kegg_hsa.gmt")
head(keggdf)
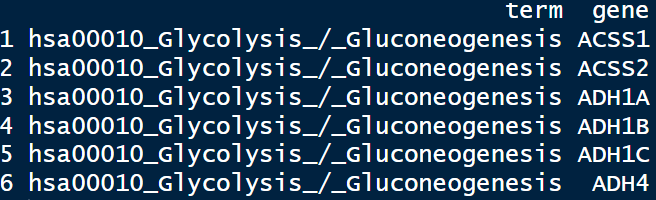
代码很简单,应该很容易实现。此外,20230205_kegg_hsa.gmt这个文件可以通过以下链接获取,链接永久有效:
链接:https://pan.baidu.com/s/1pV7Qjzj-fmSW1umqIOWEfA
提取码:17yl
往期相关教程

























 2772
2772











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








