#使用aspera能够帮助我们批量高速下载转录组等数据,现在使用conda进行安装
conda install -c hcc aspera-cli -y#检查是否安装成功,有东西出来就行
ascp -h#另外看看asperaweb_id_dsa.openssh文件是不是在miniconda3/etc文件夹中,务必确认该文件的位置。
#我们看看NCBI上面有什么样的转录组,在官网上打好毛果杨拉丁名,选择SRA搜索,我选了这个,点进去看看

#看到工程号,复制这个号,顺便看看别的一些信息,了解该转录组,看到这个是过表达了个PtrVCS2,在PUBMED搜下,文章发表在IJMS,DOI: 10.3390/ijms24054458。
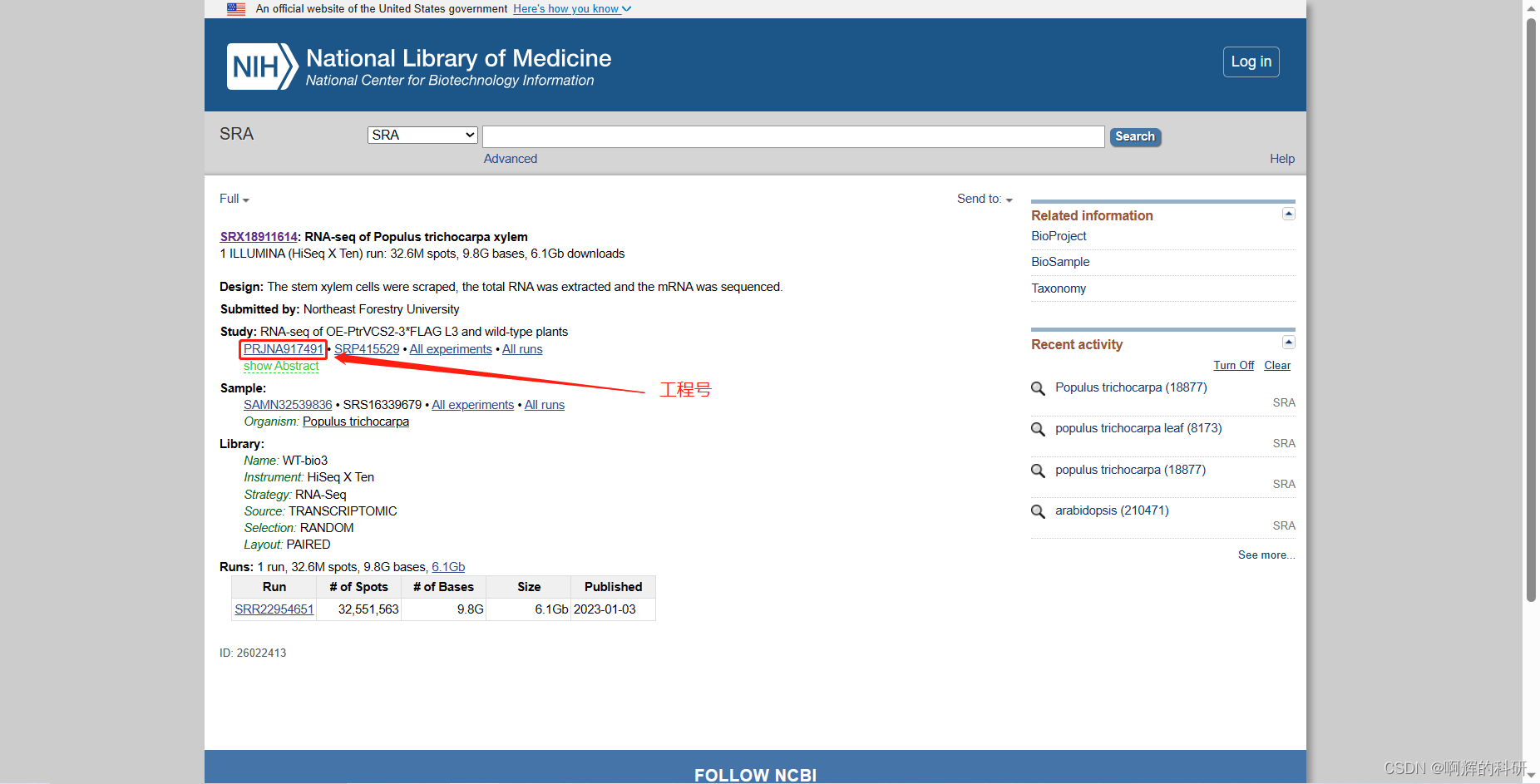
#复制工程号后,在ENA网站(https://www.ebi.ac.uk/ena/browser/home),搜索
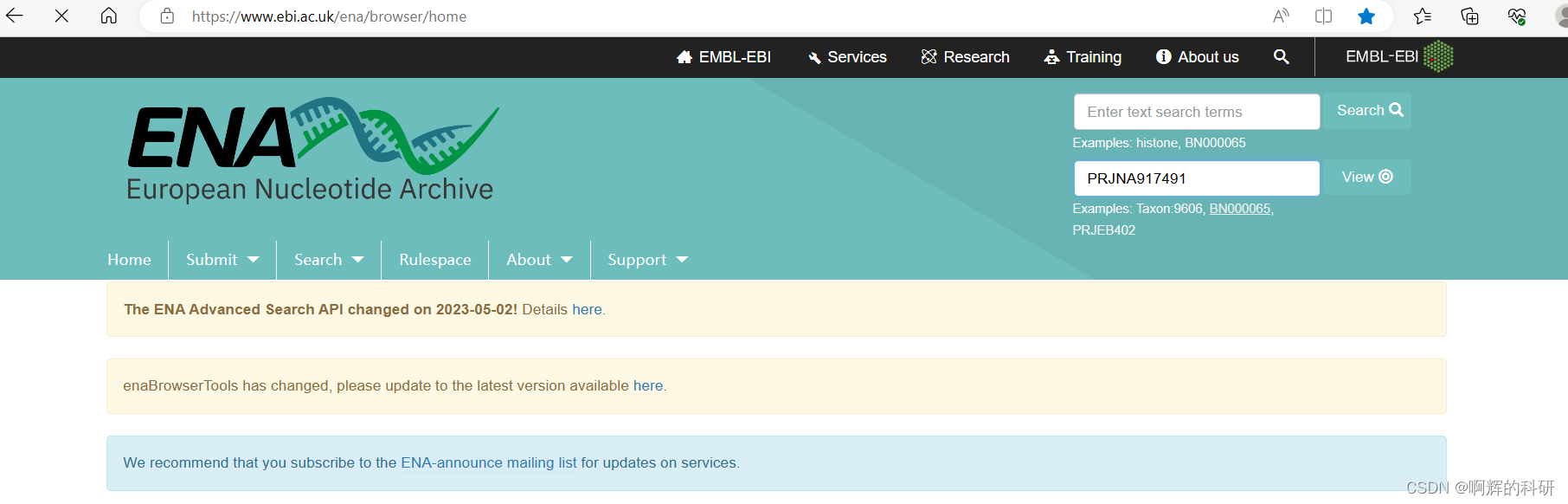
#往下拉,找到TSV,下载文件
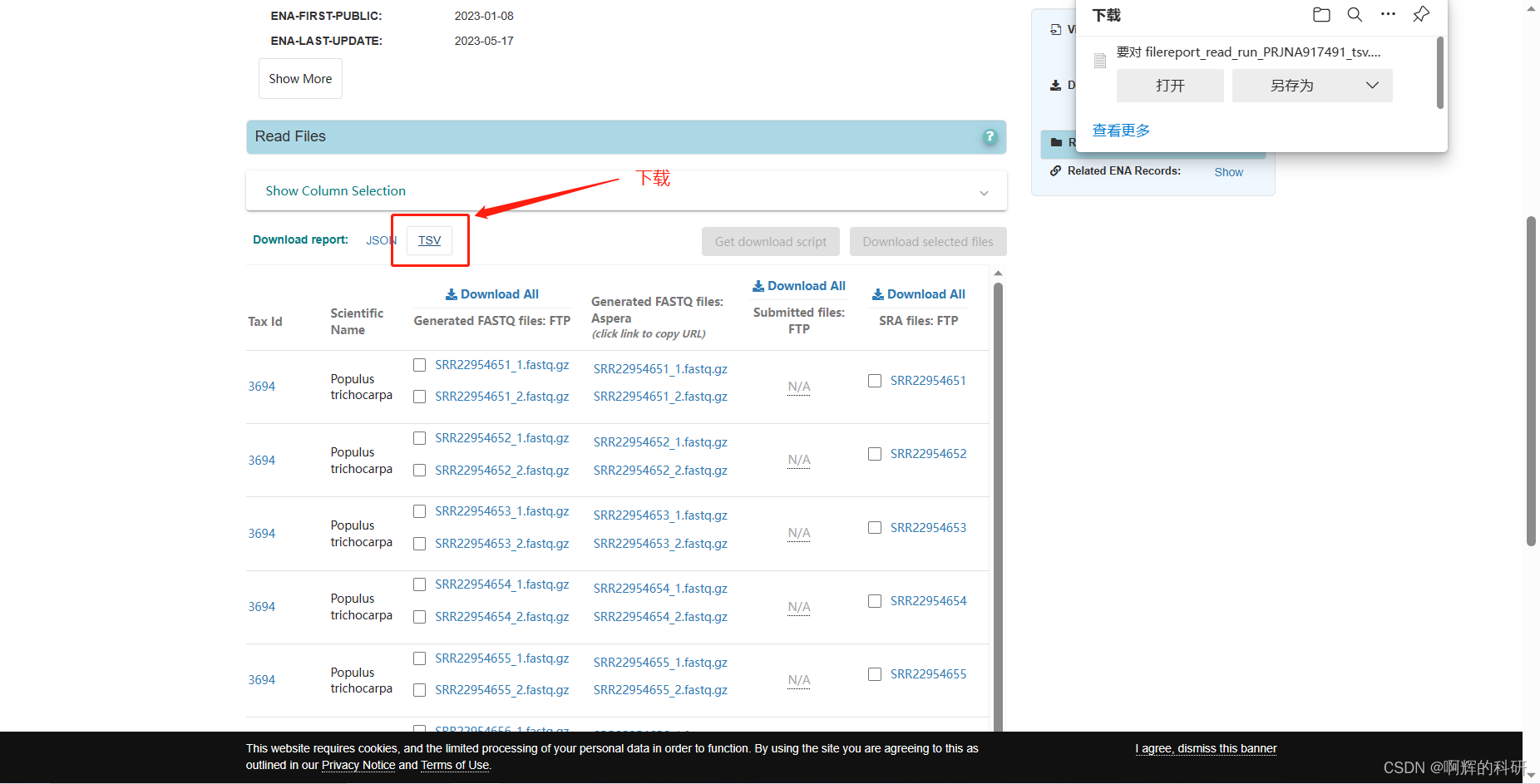
#看见文件里面有每个SRR的下载地址,如果是单个文件下载的话,命令如下,下载到当前文件夹,速度居然有280Mb/s,学校服务器真强:
ascp -vQT -l 500m -P33001 -k 1 -i \
~/miniconda3/etc/asperaweb_id_dsa.openssh \
era-fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR229/051/SRR22954651/SRR22954651_1.fastq.gz ./
#但是不可以这么笨笨哦,要学会批量下载。将刚刚下载的TSV文件整理成以下的样子,每行后面留一个空格,并且最后留一行空行出来,命名为id.txt:
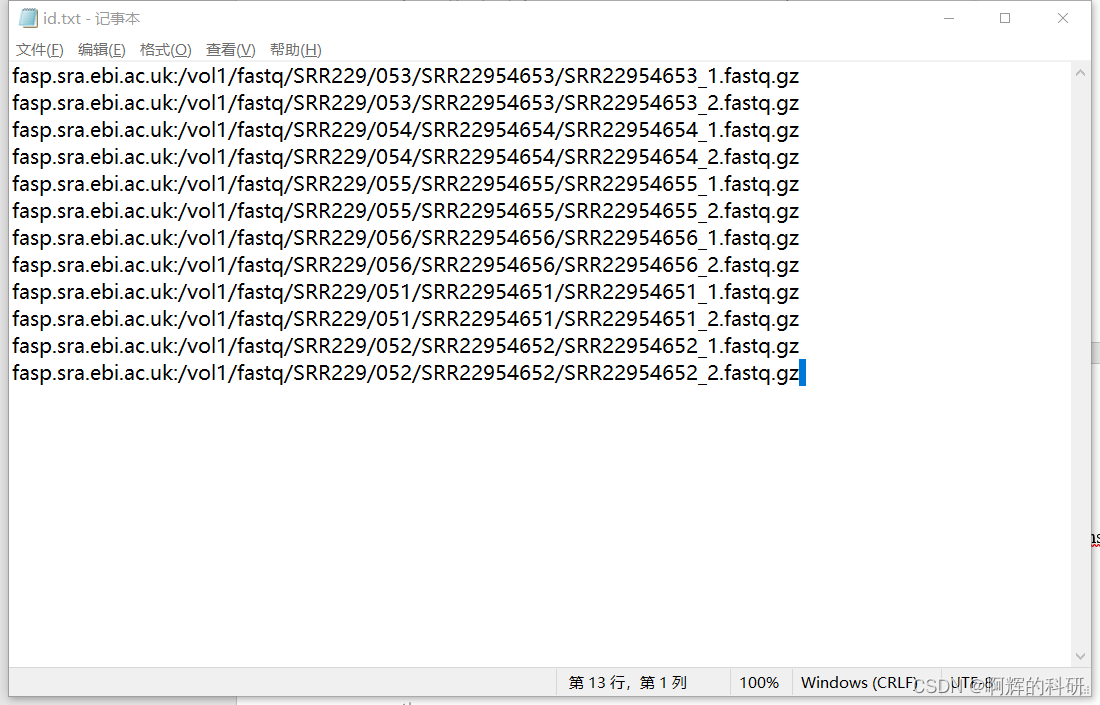
#将id.txt传到linux系统中,批量生成命令行,输出文件command_ascp.sh:
awk '{print "ascp -vQT -l 500m -P33001 -k 1 -i ~/miniconda3/etc/asperaweb_id_dsa.openssh era-fasp@"$1" ./ &"}' id.txt > command_ascp.sh#查看一下这个command_ascp.sh文件:
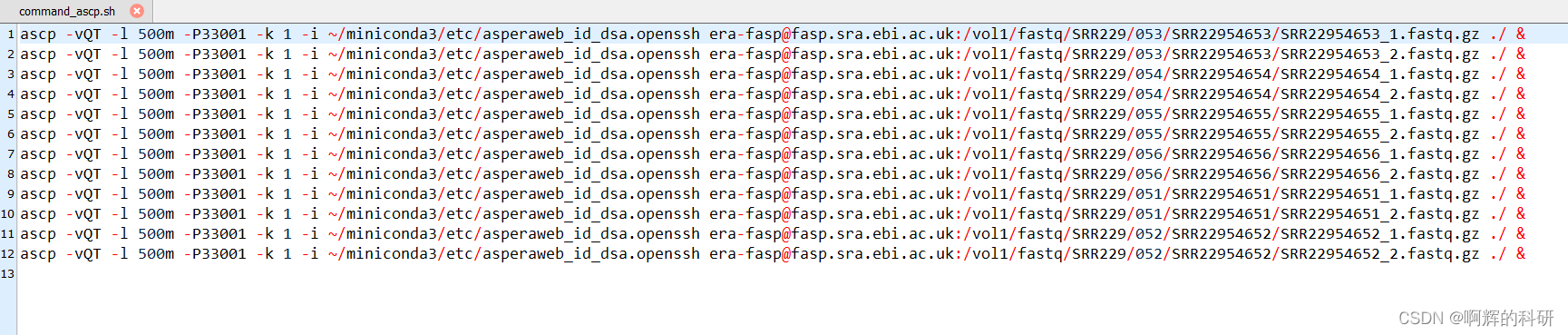
#在工作文件夹中,运行这个脚本,并挂载后台,记得两次enter:
nohup sh command_ascp.sh & 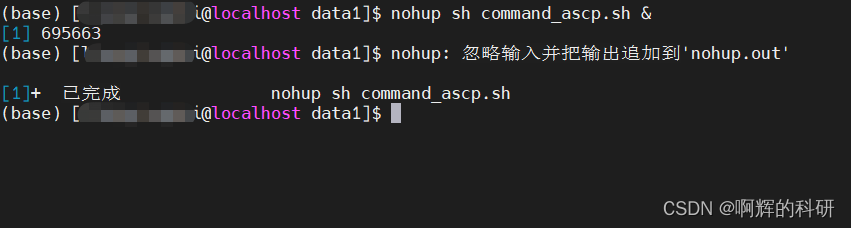
#最后看看结果,3个实验组,3个对照组,双端测序,共12个文件
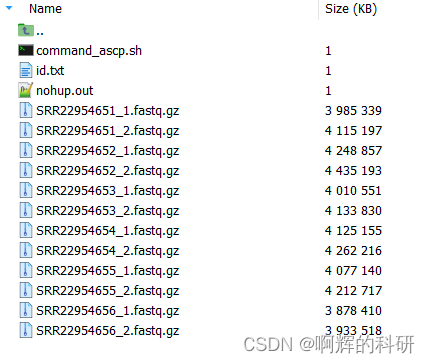
#一定要自己看懂理解代码,不可以照抄,aspera的教程有很多,有问题就百度吧。
#这两天突然高产






















 1538
1538











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










