最近在windows系统里下载了虚拟机VirtualBox(6.1.34),并在虚拟机上用Qiime2(2022.2)处理16sRNA数据,需要从NCBI数据库上下载数据。本文提供用虚拟机ubuntu或者linux系统下载Aspera的方法和问题解决,以及从NCBI上批量下载数据库、最后得到一个项目里的所有fastq文件。
【前提】本人是win10系统的电脑,下载了虚拟机VirtualBox,并用Qiime2处理数据
Aspera下载数据巨快,100多条fastq文件几分钟就下载好了,比SRA Toolkit快很多
一、安装Aspera
1. 在虚拟机终端里,先新建一个文件夹:
mkdir ~/Aspera #先建立一个放工具的文件夹
cd ~/Aspera #进入该文件夹
wget https://d3gcli72yxqn2z.cloudfront.net/downloads/connect/latest/bin/ibm-aspera-connect_4.2.8.540_linux_x86_64.tar.gz #从官网地址下载
tar xvf ibm-aspera-connect_4.2.8.540_linux_x86_64.tar.gz #解压,注意版本号要对应
bash ibm-aspera-connect_4.2.8.540_linux_x86_64.sh # 解压后得到一个脚本文件,运行该脚本,即可完成自动安装
注意上面的链接是截止2024.5.9的最新下载Aspera的链接,可以去https://www.ibm.com/aspera/connect/查看最新版本(选择IBM Aspera Connect下面的Linux系统,复制链接,见下图),同时更改上面代码里的版本号即可。

安装完毕:

2. 添加环境变量:
echo 'export PATH=~/.aspera/connect/bin/:$PATH' >> ~/.bashrc # 所有安装文件都在~/.aspera/connect目录下,在 ~/.bashrc添加环境变量 (cd /home/qiime2/.aspera/connect)
source ~/.bashrc # 使环境变量生效
which ascp # 查看ascp可执行文件所在的路径,应该是:~/.aspera/connect/bin/ascp

用以下指令查看程序能否正常运行
ascp -h
说明能正常运行:
如果出现’GLIBC_2.28’ not found的错误,参考本号内的上一篇文章《冒险升级ubuntu系统解决sk-learn和glibc版本低的问题》
二、导入密钥文件
安装完Aspera如果直接爬取数据,会出现找不到密钥文件的错误:Private key file not found at path /home/qiime2/.aspera/connect/etc/asperaweb_id_dsa.openssh
IBM官网写的,Aspera 4.2版本之后需要用微软加的OpenSSH。所以我们要先下载OpenSSH再导入密钥文件。
我参考了很多文章,整合了很完整的导入密钥文件的做法,如下:
1. 下载OpenSSH:
打开电脑设置,搜索“可选功能”-管理可选功能
 点击上方添加功能,输入OpenSSH,添加OpenSSH服务器(一般电脑自带OpenSSH客户端)
点击上方添加功能,输入OpenSSH,添加OpenSSH服务器(一般电脑自带OpenSSH客户端)
 待安装好后,按win+R输入命令services.msc,打开服务窗口双击这条,设置启动状态为为自动
待安装好后,按win+R输入命令services.msc,打开服务窗口双击这条,设置启动状态为为自动

2. 制作密钥文件
在windows系统里新建一个txt文档,输入以下key,再重命名为asperaweb_id_dsa.openssh,把该文件放到共享文件夹里:
-----BEGIN DSA PRIVATE KEY-----
MIIBuwIBAAKBgQDkKQHD6m4yIxgjsey6Pny46acZXERsJHy54p/BqXIyYkVOAkEp
KgvT3qTTNmykWWw4ovOP1+Di1c/2FpYcllcTphkWcS8lA7j012mUEecXavXjPPG0
i3t5vtB8xLy33kQ3e9v9/Lwh0xcRfua0d5UfFwopBIAXvJAr3B6raps8+QIVALws
yeqsx3EolCaCVXJf+61ceJppAoGAPoPtEP4yzHG2XtcxCfXab4u9zE6wPz4ePJt0
UTn3fUvnQmJT7i0KVCRr3g2H2OZMWF12y0jUq8QBuZ2so3CHee7W1VmAdbN7Fxc+
cyV9nE6zURqAaPyt2bE+rgM1pP6LQUYxgD3xKdv1ZG+kDIDEf6U3onjcKbmA6ckx
T6GavoACgYEAobapDv5p2foH+cG5K07sIFD9r0RD7uKJnlqjYAXzFc8U76wXKgu6
WXup2ac0Co+RnZp7Hsa9G+E+iJ6poI9pOR08XTdPly4yDULNST4PwlfrbSFT9FVh
zkWfpOvAUc8fkQAhZqv/PE6VhFQ8w03Z8GpqXx7b3NvBR+EfIx368KoCFEyfl0vH
Ta7g6mGwIMXrdTQQ8fZs
-----END DSA PRIVATE KEY-----
把文件从共享文件夹移动到官方指定文件夹(套用代码注意更改共享文件夹名字sf_qiimeshare和用户名文件夹qiime2,其他部分都一样):
mv /media/sf_qiimeshare/asperaweb_id_dsa.openssh /home/qiime2/.aspera/connect/etc/asperaweb_id_dsa.openssh
查找一下密钥文件的位置(这个很重要,后面代码都会用到):
find ~ -name asperaweb_id_dsa.openssh
可以的,很正确的:
三、利用Aspera下载SRA数据(批量下载直接跳转至下一节)
参考了好几篇攻略,可用以下代码(以提取SRR1342456号码下的数据为例):
对于双端序列,用下指令,注意修改_1和_2,代表两个序列(如果是单端序列则去掉_1):
ascp -v -Q -T -l 200m -P 33001 -k 1-i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/SRR134/006/SRR1342456/SRR1342456_1.fastq.gz ./
还可以用下面这个代码,双端序列会生成一个文件夹里两个.fastq.gz如下图(单端序列则生成一个)
ascp -v -Q -T -l 200m -P 33001 -k 1 -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/SRR134/006/SRR1342456/ ./

上述代码需要修改的只有末尾数字部分(前面的200m最大可填500m,代表下载速度)
难点是006这个三位数,对于示例样本编号为SRR+7位数,三位数取00和样本号最后一位;对于样本编号为SRR+8位数,取0和样本编号最后两位;对于样本编号为SRR+6位数,则去掉这个三位数(SRR134/006/SRR134246 改为 SRR134/SRR134246)
对于.fastq.gz文件,使用以下代码解压缩,生成.fastq文件,即可进行后续分析
gzip -d SRR1342456_2.fastq.gz
以上方法适合数据少,单次下载。
四、Aspera批量下载SRA数据
Aspera下载fastq文件真的巨巨巨巨巨快,下载一百多条估计也只要几分钟,但中间会有数据下载失败的情况,解决方法都放在了本小节后面。
1. 批量导出Accession list
在网址NCBI网址上搜索项目号:
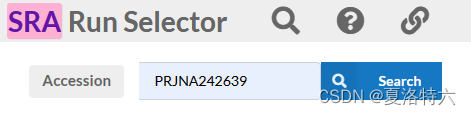
点击Accession list即可下载序列号:

2. 利用cat指令生成idname文件
在虚拟机建一个文件夹放数据,我这里数据文件夹叫Aspera
打开虚拟机终端,进入Aspera文件夹,并建立存放SRR序列号的idname文件夹
cd ~/Aspera
cat idname #点击回车后,把Accession list数据复制下来,再按ctrl+C保存文件
注意每行不能有空格,否则后续运行脚本可能会出错。
我选的这个项目里一共有16条序列号:
3. 制作批量运行的脚本
输入以下代码,进行脚本编辑
vim Download.sh
在弹出来的框框里,复制粘贴以下内容,以下脚本能自动根据SRR号的位数下载对应数据。运行前注意修改密匙存放路径和自己的项目路径:
date
## 目录设置
mkdir -p ~/Aspera #这个文件路径可以自行修改,是存下载数据的地方
cd ~/Aspera
pwd
## 密匙路径
openssh=/home/qiime2/.aspera/connect/etc/asperaweb_id_dsa.openssh
cat ~/Aspera/idname | while read id
do
num=`echo $id | wc -m ` #这里注意,wc -m 会把每行结尾$也算为一个字符
echo "SRRnum+1 is $num" #因此SRRnum + 1才等于$num值
#如果样本编号为SRR+8位数 #
if [ $num -eq 12 ]
then
echo "SRR + 8"
x=$(echo $id | cut -b 1-6)
y=$(echo $id | cut -b 10-11)
echo "Downloading $id "
( ascp -QT -l 500m -P33001 -k 1 -i $openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/$x/0$y/$id/ ./ & )
#如果样本编号为SRR+7位数 #
elif [ $num -eq 11 ]
then
echo "SRR + 7"
x=$(echo $id | cut -b 1-6)
y=$(echo $id | cut -b 10-10)
echo "Downloading $id "
( ascp -QT -l 500m -P33001 -k 1 -i $openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/$x/00$y/$id/ ./ & )
#如果样本编号为SRR+6位数 #
elif [ $num -eq 10 ]
then
echo "SRR + 6"
x=$(echo $id |cut -b 1-6)
echo "Downloading $id "
( ascp -QT -l 500m -P33001 -k 1 -i $openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/$x/$id/ ./ & )
fi
done
在末尾输入:w保存文件,再输入:q退出文件。
运行脚本文件:
sh Download.sh
一共16个文件,三四分钟就下载好了,存在了Aspera文件夹里。但是有时候会因为网络不稳定而漏掉几个文件,找出这几个文件的SRR号,运行cat指令重新输入几个文件的SRR号码,注意不要留多余空格:
cat >idname
之后重新运行程序
sh Download.sh

中间出现了一次Error: Client unable to connect to server (check UDP port and firewall),查了发现是本地服务器开启防火墙导致33001端口没有打开。
可以用以下指令打开:
sudo iptables -I INPUT -p udp --dport 33001 -j ACCEPT
sudo iptables -I OUTPUT -p udp --dport 33001 -j ACCEPT

【注意:如果需要下载很多条数据,建议分批次运行,一次运行十条左右】
4. 批量操作文件夹(批量移动、解压缩)
现在得到的是,放数据的文件夹里,每个SRR文件夹里有两个.fastq.gz文件:

将所有.fastq.gz文件都移到/Aspera文件里,并解压缩所有fastq.gz文件:
find /home/qiime2/Aspera/ -name "*.fastq.gz" | xargs -i cp {} /home/qiime2/Aspera
ls *.fastq.gz | xargs -n1 gzip -d #批量解压缩所有文件
最后得到是Aspera文件夹里的32个fastq文件(双端的),再根据自己需求把这些fastq文件移动到所需文件夹,用qiime_share共享文件夹可以把数据移动到win10系统的盘里
linux系统里的共享文件夹:

win10系统里的共享文件夹:

里面的内容是可以同时传递的
注:关于虚拟机的安装、初始设置、共享文件夹的设置等可自行查阅其他资料























 1193
1193

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








