
【Title】ChIP-R: Assembling reproducible sets of ChIP-seq and ATAC-seq peaks from multiple replicates
【Publication Book】Genomics
【Publication Time】2021 April
【Abstract】
The oocyte cytoplasm can reprogram the somatic cell nucleus into a totipotent state, but with low efficiency. The spatiotemporal chromatin organization of somatic cell nuclear transfer (SCNT) embryos remains elusive. Here, we examine higher order chromatin structures of mouse SCNT embryos using a low-input Hi-C method. We find that donor cell chromatin transforms to the metaphase state rapidly after SCNT along with the dissolution of typical 3D chromatin structure. Intriguingly, the genome undergoes a mitotic metaphase-like to meiosis metaphase II-like transition following activation. Subsequently, weak chromatin compartments and topologically associating domains (TADs) emerge following metaphase exit. TADs are further removed until the 2-cell stage before being progressively reestablished. Obvious defects including stronger TAD boundaries, aberrant super-enhancer and promoter interactions are found in SCNT embryos. These defects are partially caused by inherited H3K9me3, and can be rescued by Kdm4d overexpression. These observations provide insight into chromatin architecture reorganization during SCNT embryo development.
【Background】
体细胞核转移(SCNT)是将体细胞重新编程为全能胚胎并产生有活力的动物的唯一途径。
染色质3D结构具有高度动态性,与许多生物过程有关。相间染色质3D结构的分层原则包括染色体区域、染色质隔间(A/B)、TAD和环。A和B室是两种多兆碱基结构域,其特征是活性和非活性染色质的空间分离
我们检查了SCNT胚胎发育连续阶段的三维染色质结构,发现在SCNT胚胎发生过程中,包括隔间和TAD在内的高阶染色质结构以特定阶段和协调的方式溶解和重新建立。H3K9me3修饰可能是一个表观遗传屏障,在SCNT胚胎发育期间损害了染色质结构的重新编程。因此,我们的发现提供了一张高分辨率的地图,说明体细胞的成熟3D染色质结构在移植到无核卵母细胞后如何重新编程到全能状态。
【Result】
1.SCNT胚胎的三维染色质结构
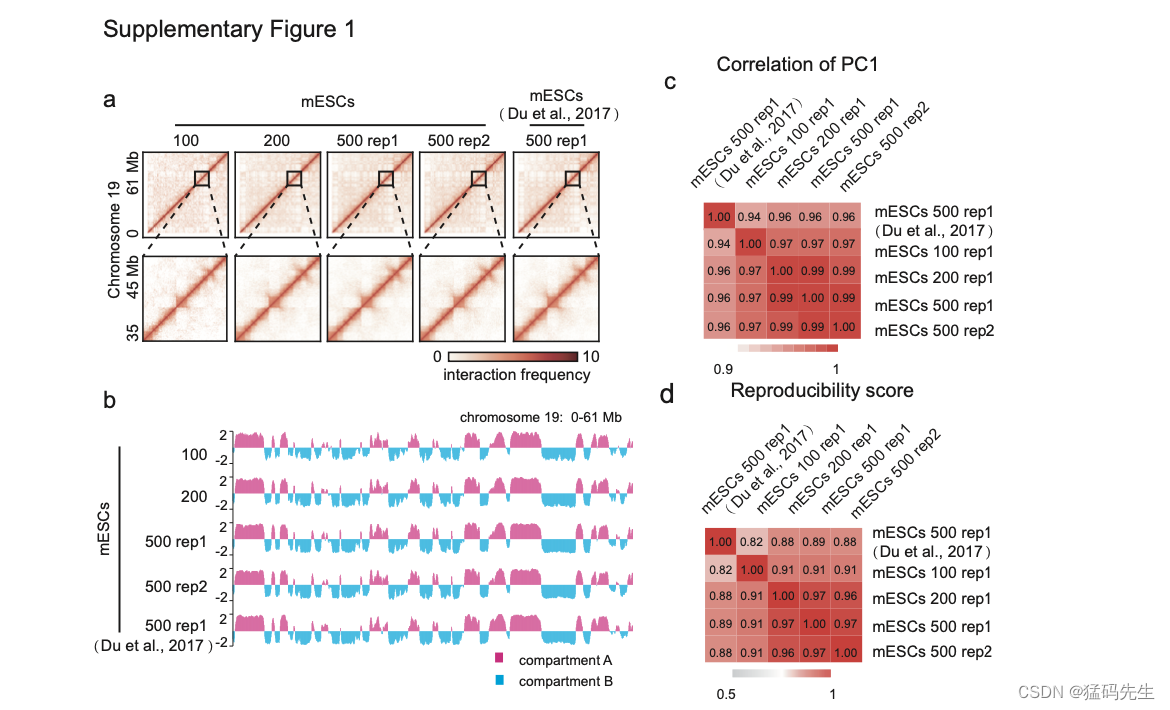 使用100-500个小鼠ES细胞生成了高质量的Hi-C数据,这些数据与之前报告的染色质相互作用模式和架构准确一致(补充图1a-d)。
使用100-500个小鼠ES细胞生成了高质量的Hi-C数据,这些数据与之前报告的染色质相互作用模式和架构准确一致(补充图1a-d)。
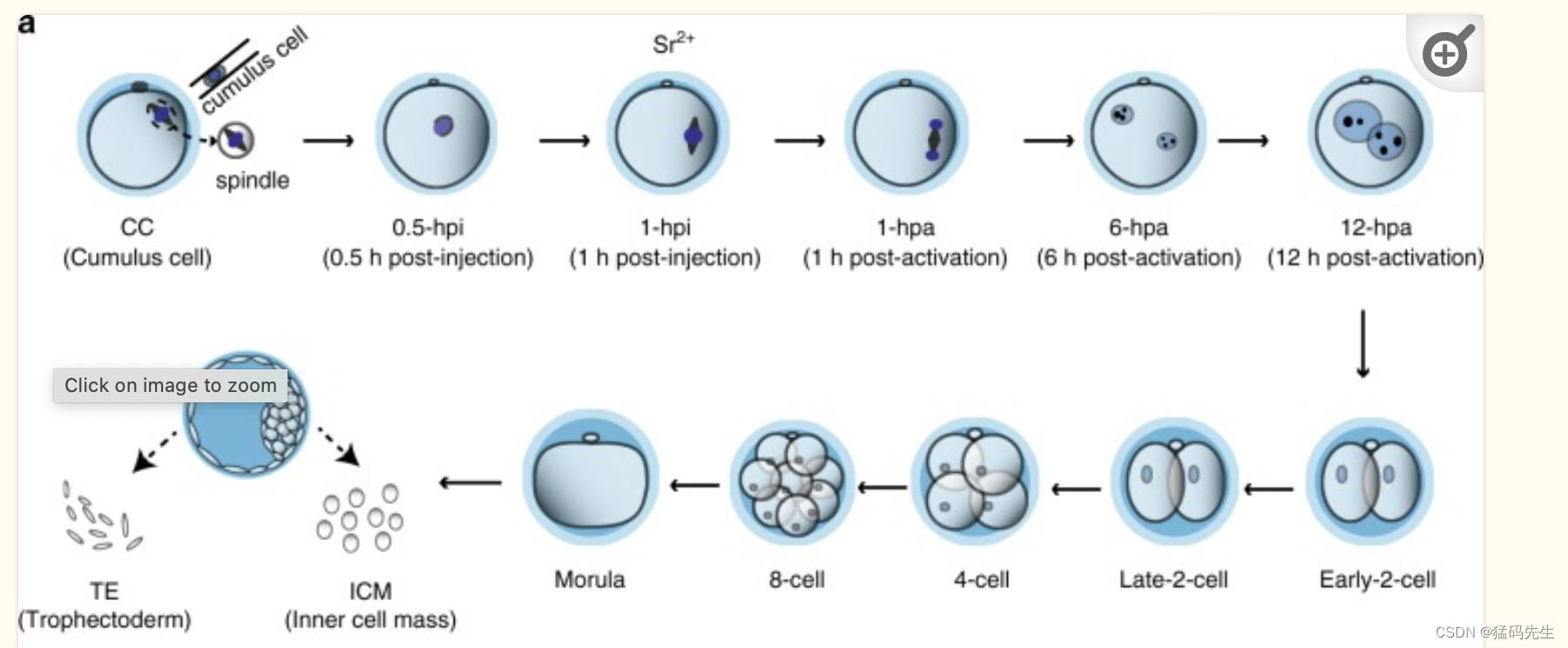
在每个阶段进行了Hi-C实验
0.5 h post-injection (0.5-hpi), 1-hpi, 1 h postactivation (1-hpa), 6-hpa, 12-hpa, early-2-cell embryo, late-2-cell embryo, 4-cell embryo, 8-cell embryo, morula embryo, as well as inner cell mass (ICM) and trophectoderm (TE)
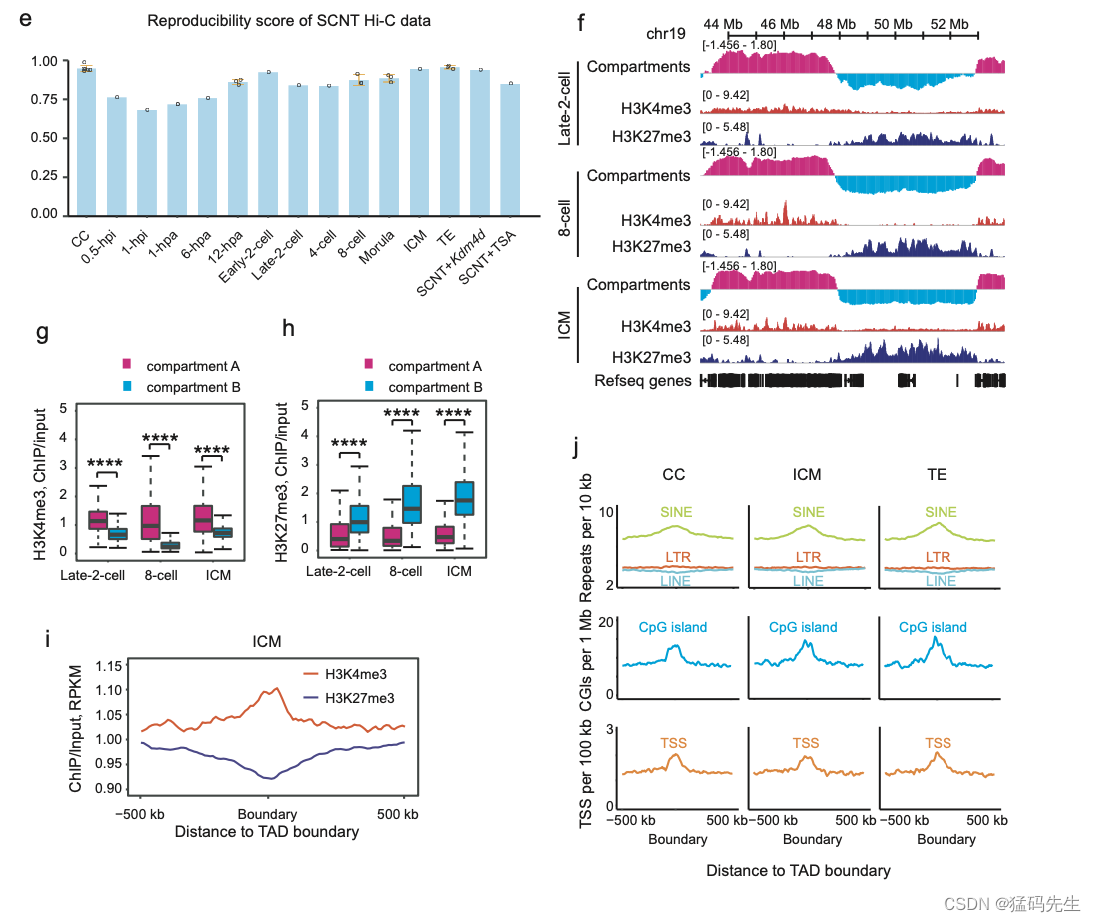 复制的Hi-C数据具有高度可复制性(补充图1e)。与与高阶染色质架构相关的报告特征一致,H3K4me3主要在A室中富集,H3K27me3主要在B室中富集(补充图1f-h)。同样,H3K4me3在TAD的边界上富集,而H3K27me3则耗尽(补充图1i)。此外,SINE逆转录转座子、CpG岛和基因启动子也富含TAD边界(补充图1j)。
复制的Hi-C数据具有高度可复制性(补充图1e)。与与高阶染色质架构相关的报告特征一致,H3K4me3主要在A室中富集,H3K27me3主要在B室中富集(补充图1f-h)。同样,H3K4me3在TAD的边界上富集,而H3K27me3则耗尽(补充图1i)。此外,SINE逆转录转座子、CpG岛和基因启动子也富含TAD边界(补充图1j)。
2.SCNT胚胎激活后的中期过渡
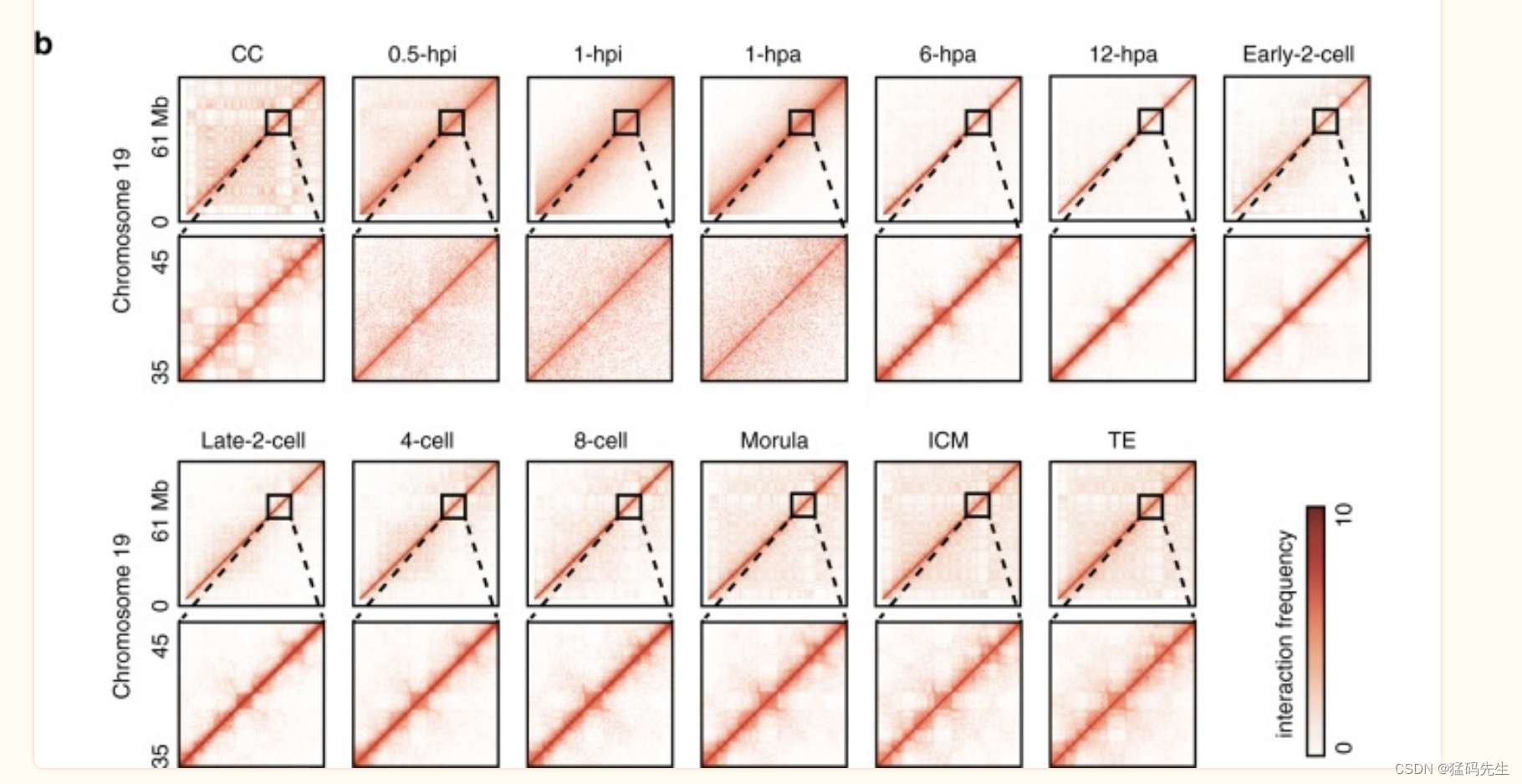
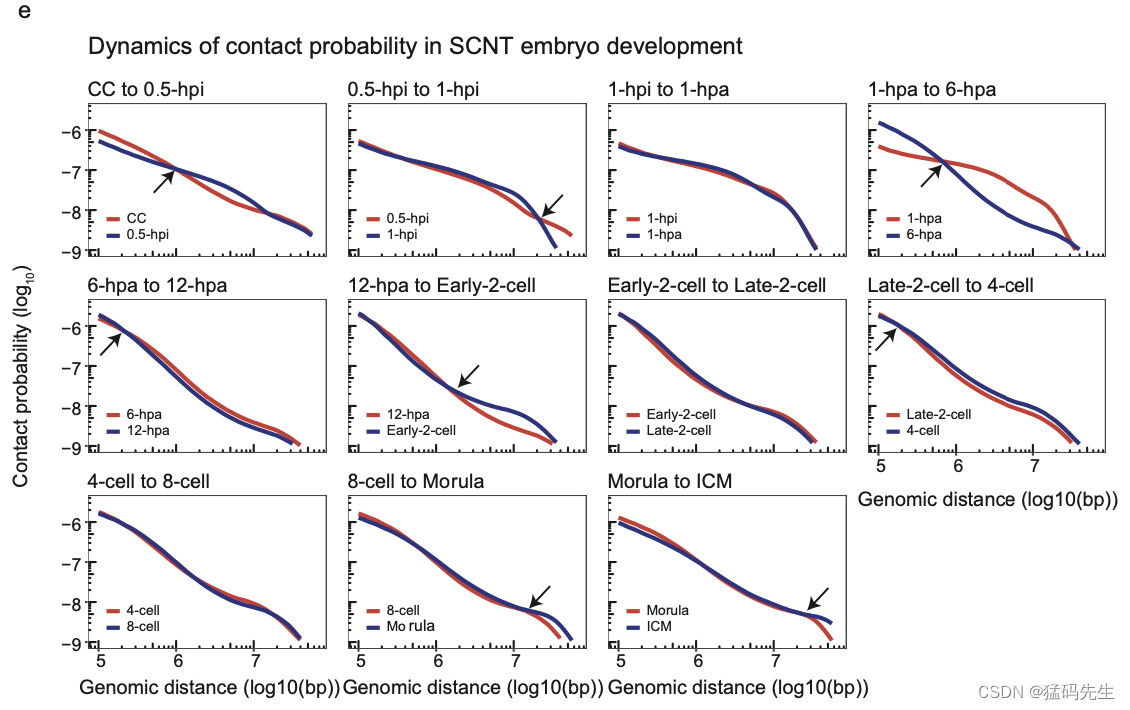
末端分化CC中的TAD和隔间,在将CC核注入无核卵母细胞后迅速溶解,并在6hpa胚胎中部分恢复(图1b),1-hpi和1-hpa单细胞胚胎的远端相互作用明显减少,并表现出均匀的相互作用模式(图1b),这与在中期染色质组织中观察到的非常相似
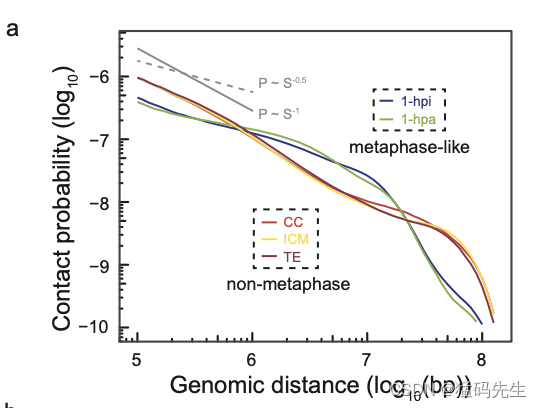
然后,我们分析了1-hpi和1-hpa阶段的染色质构象,
1-hpi和1-hpa胚胎的P(s)曲线(相对于基因组距离的染色质接触概率)更好地匹配P(s)~s-0.5曲线,代表预测的中期染色质状态;CCs、ICM和TE的P(s)曲线更好地匹配P(s)~s-1曲线,代表预测的分形球状态(相间染色质状态)(补充图2a)
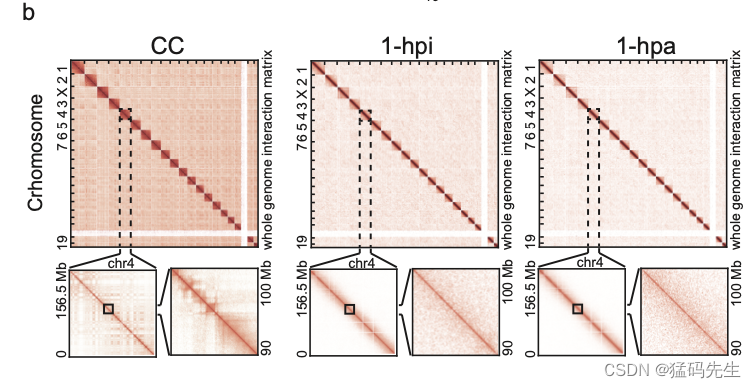
中期样染色质状态的特点是缺乏染色质间相互作用,这与1-hpi和1-hpa阶段胚胎的染色质结构一致(补充图2b)
这一结果表明,体细胞基因组在注射卵母细胞质后不久(在1hpi和1-hpa单细胞阶段)后转换为中期样状态
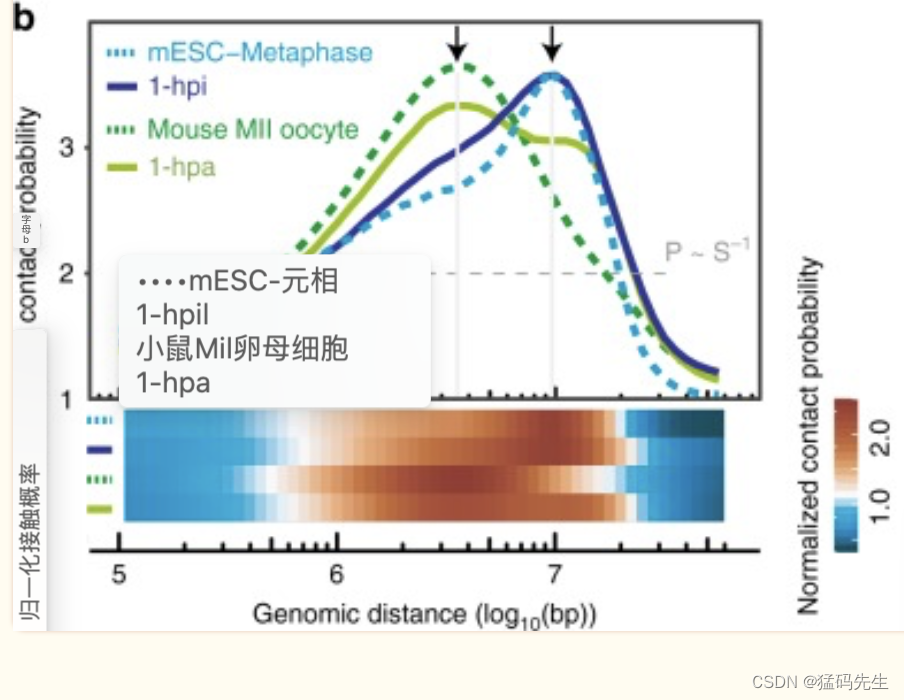
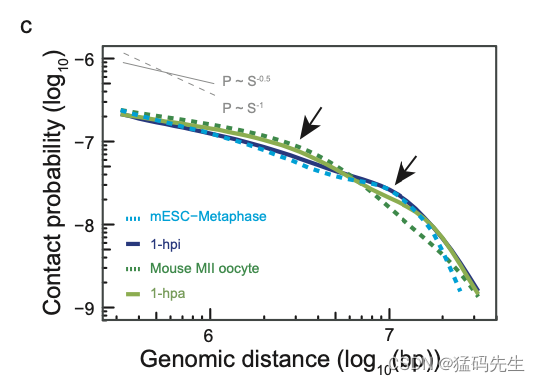
进一步分析表明,1hpi胚胎的P(s)曲线类似于小鼠ESC中期,在10 Mb时快速下降。相比之下,1hpa胚胎的P(s)曲线类似于小鼠MII卵母细胞,突然下降,直到4 Mb(图2b;补充图2c)。这一结果与之前的发现一致,即在MII卵母细胞中,染色质相互作用急剧减少超过4 Mb超过10 Mb的有丝分裂染色质,表明1-hpi胚胎中的有丝分裂中期样染色质状态在激活后转变为减数分裂中期II样染色质状态。
3.短距离交互在SCNT后首先溶解
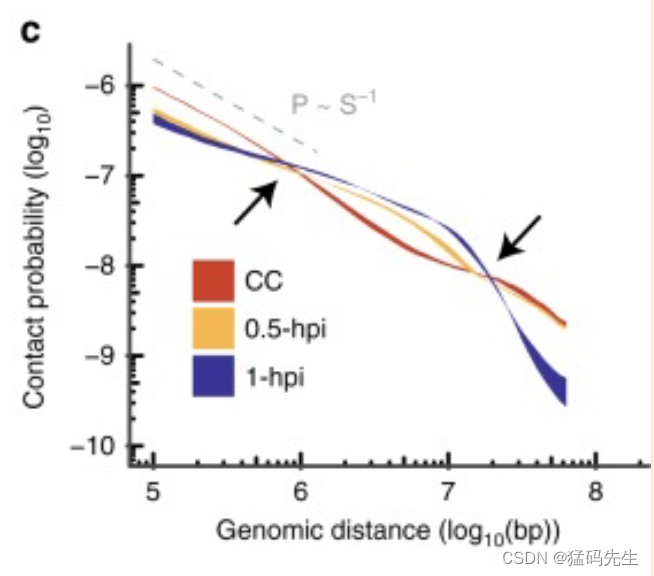
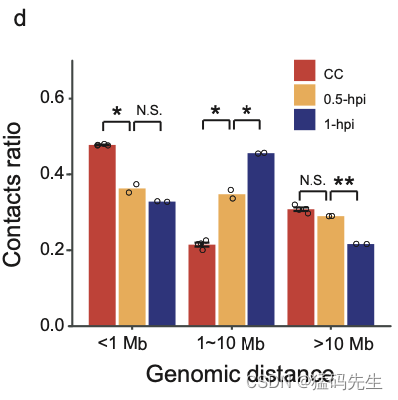
准确的时空染色质包装对胚胎发生至关重要。因此,我们比较了连续阶段之间的P(s)曲线。将CC核注射到去核卵母细胞后,短距离(<1 Mb)相互作用减少,中间距离(1-10 Mb)相互作用在前30分钟内增加。然而,长距离(>10 Mb)相互作用需要更长的时间才能改变,并在1hpi阶段显著减少(图2c,补充图2d)。这一结果表明,从间期到中期样染色质结构的重组从短距离相互作用开始,并发展为长距离相互作用。这些发现与最近的一项研究一致,该研究表明,在有丝分裂出口和G1重新进入期间,远程分隔发生得更慢。有趣的是,时空染色质包装在6hpa阶段逆转,短距离相互作用增加,中间距离相互作用减少。短距离相互作用在6hpa后变化不大,并下降到ICM阶段的水平。相比之下,中距离和长距离相互作用在早期-2细胞阶段增加,超长距离(>20 Mb)相互作用在morula和ICM阶段都增加(补充图2e)。总的来说,这些结果表明,时空染色质包装是一个协调的过程,涉及早期阶段的高阶动力学,最终在ICM阶段涉及染色质压实。
4.SCNT胚胎发育期间的隔间重新编程
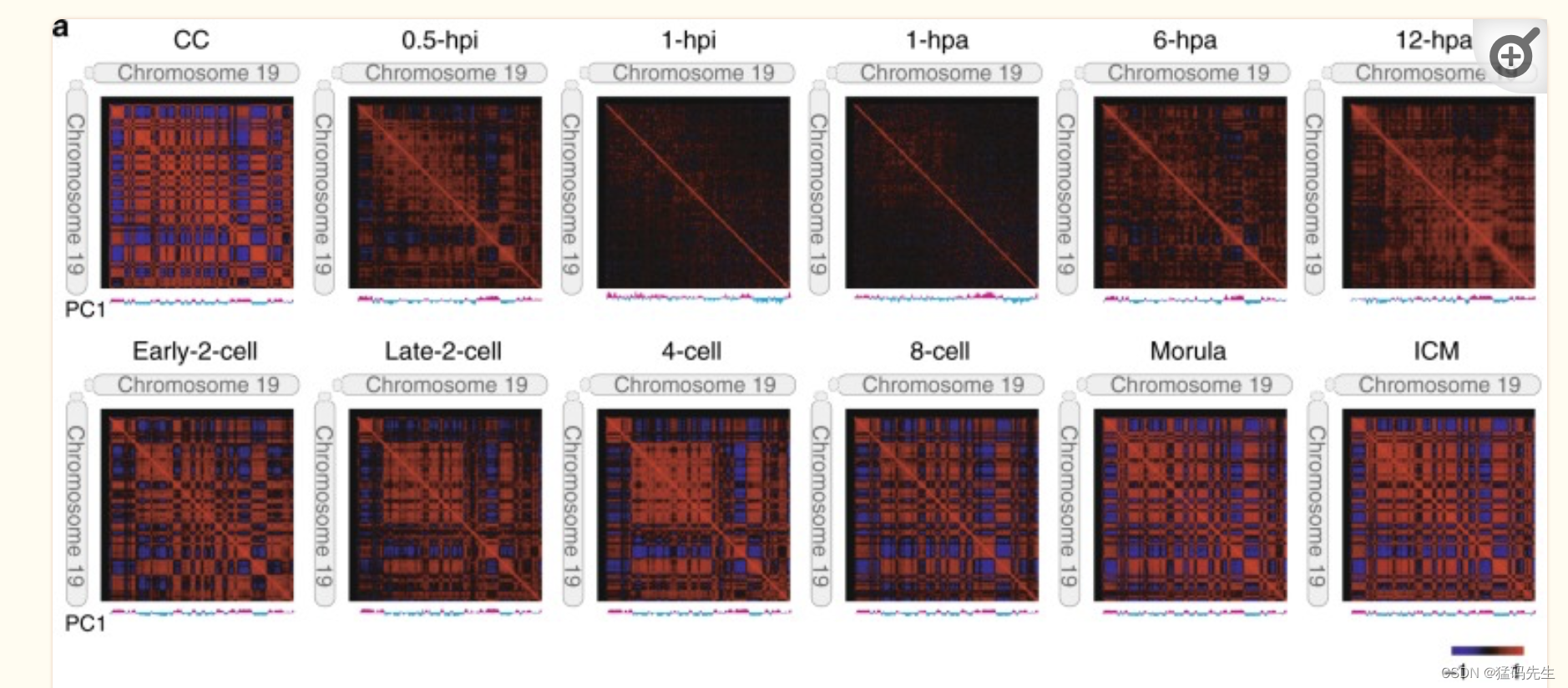
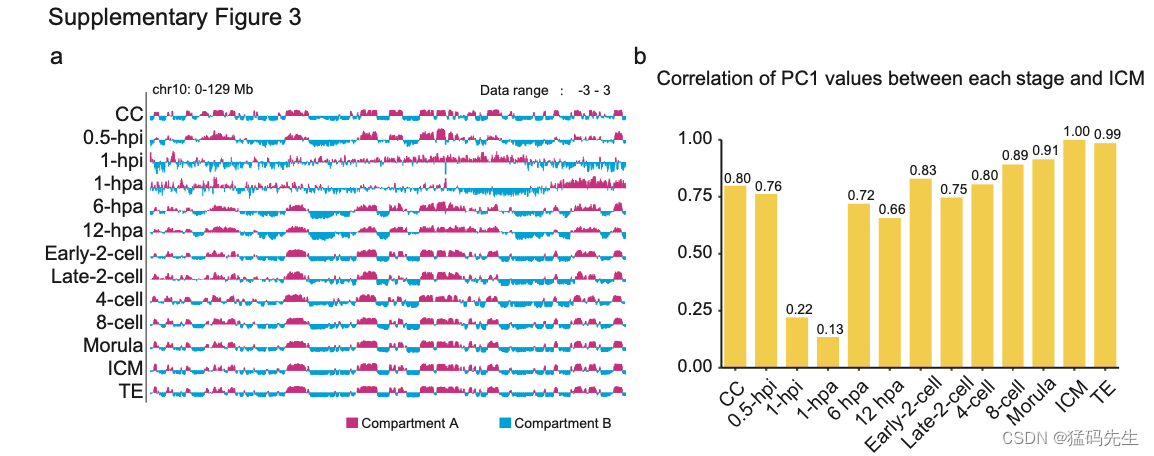
隔间是典型的高阶染色质结构,有助于确定细胞类型特异性基因表达模式。我们观察到除1-hpi和1-hpa阶段外,在所有阶段的相关热图中观察到染色质相互作用的清晰格子模式(图3a)。这些格子图案代表染色质隔间A和B。为了了解SCNT胚胎发育期间染色质分区的重新编程,我们首先确定了所有阶段的分区(见方法)。与中期样染色质状态一致,在1-hpi和1-hpa胚胎中分离了隔间,与CC或SCNT ICM中的成熟隔间没有相关性(补充图3a,b)。这一结果表明,CCs中的成熟隔间在注射到无核卵母细胞后基本溶解,导致1-hpi和1-hpa胚胎的染色质分隔不良。尽管如此,其他阶段的隔间是一致的。
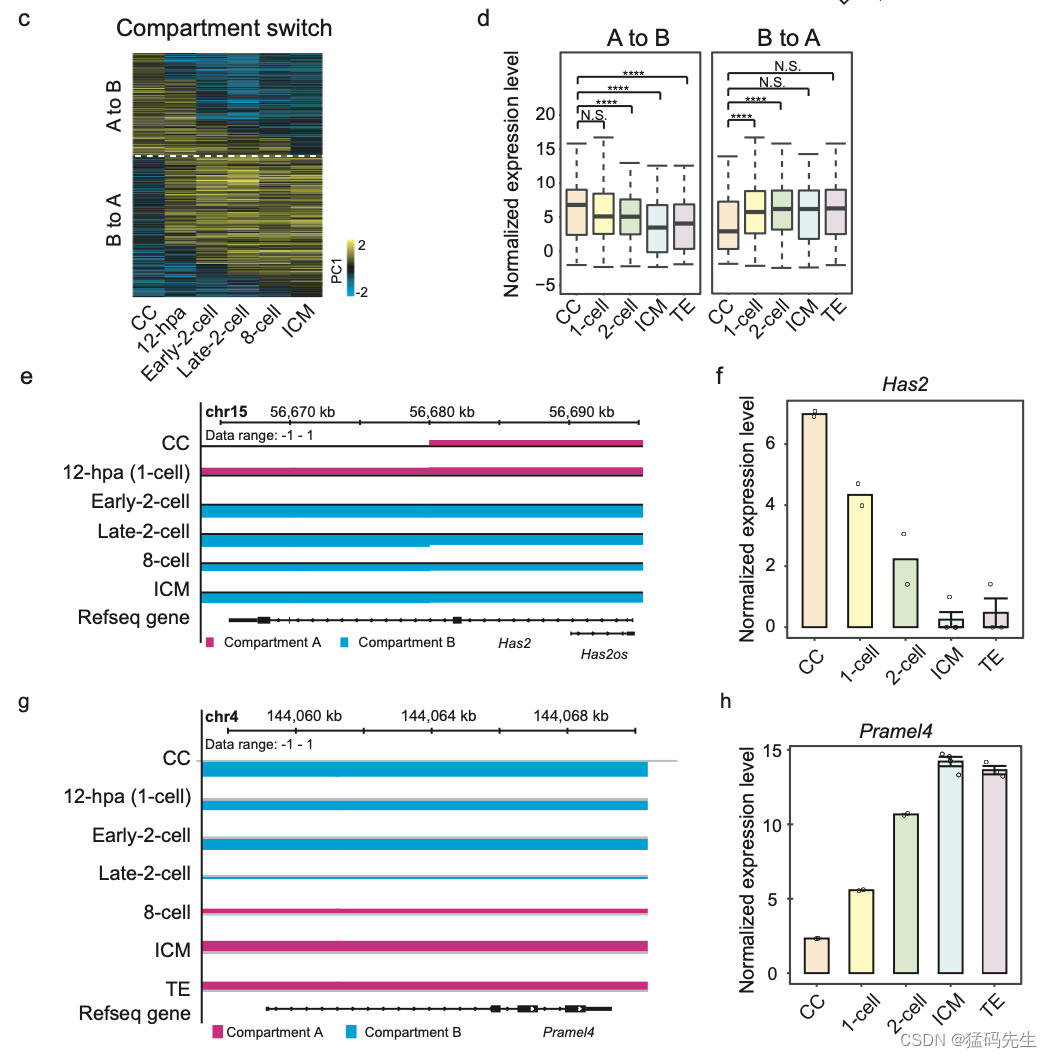
然后,我们分析了SCNT胚胎发育期间的分区转换,并收集了两组分区。一组由CC中的752个隔间A组成,这些隔间逐渐成为ICM中的B隔间。相比之下,CC中的另一组1046个隔间B逐渐成为ICM中的A隔间(补充图3c)。包括CC特异性基因Has2在内的A至B组分区中的基因表达水平随着分区间开关的进展而显著下降。B到A组的基因表达水平,包括胎盘相关基因Pramel4,表现出相反的趋势(补充图3d-h)。
5.在SCNT胚胎发育期间重新编程TAD
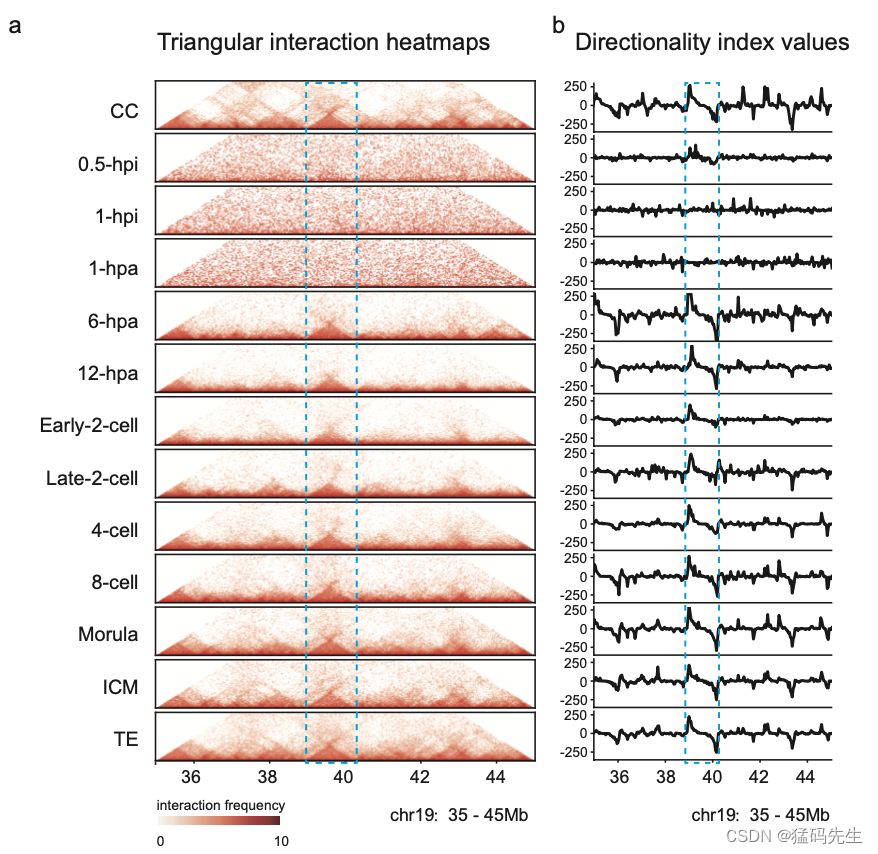
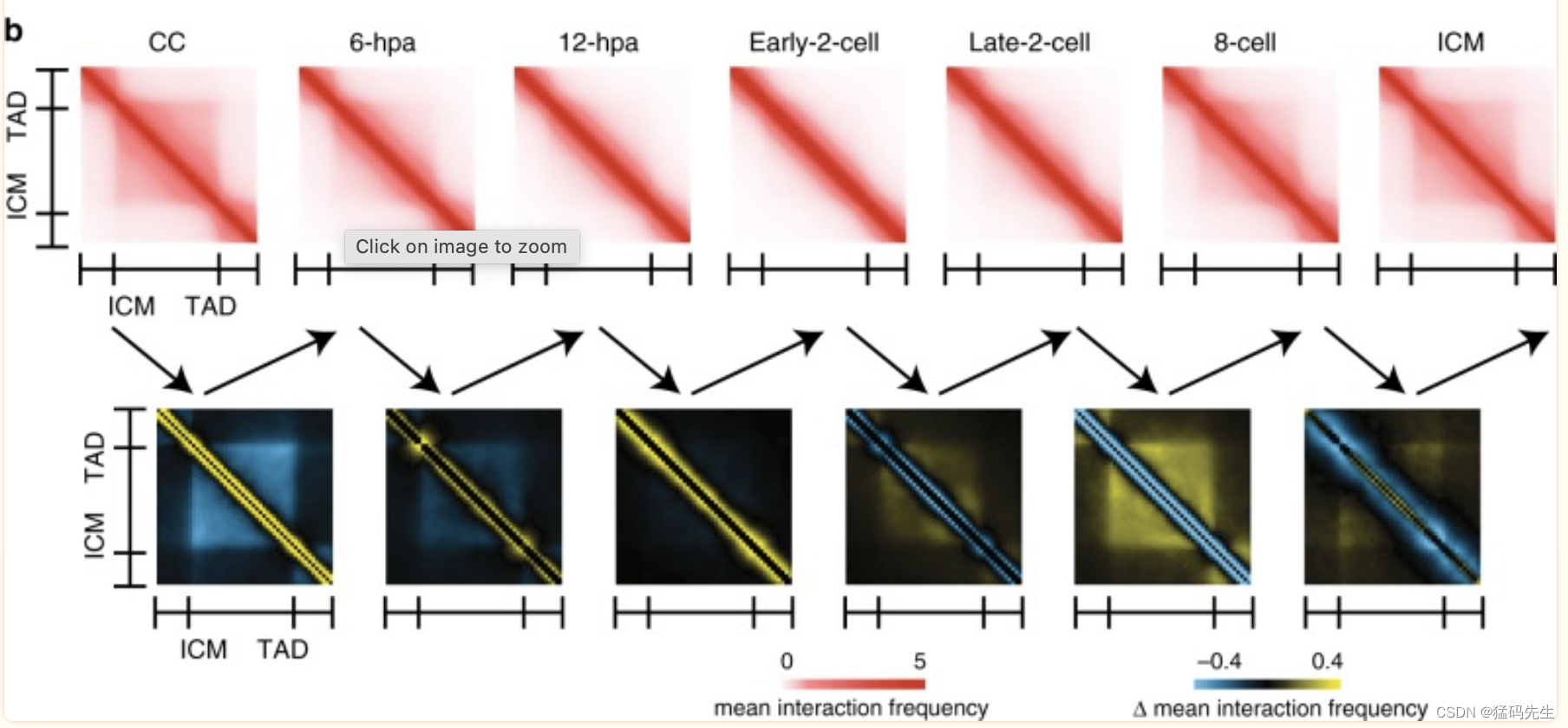
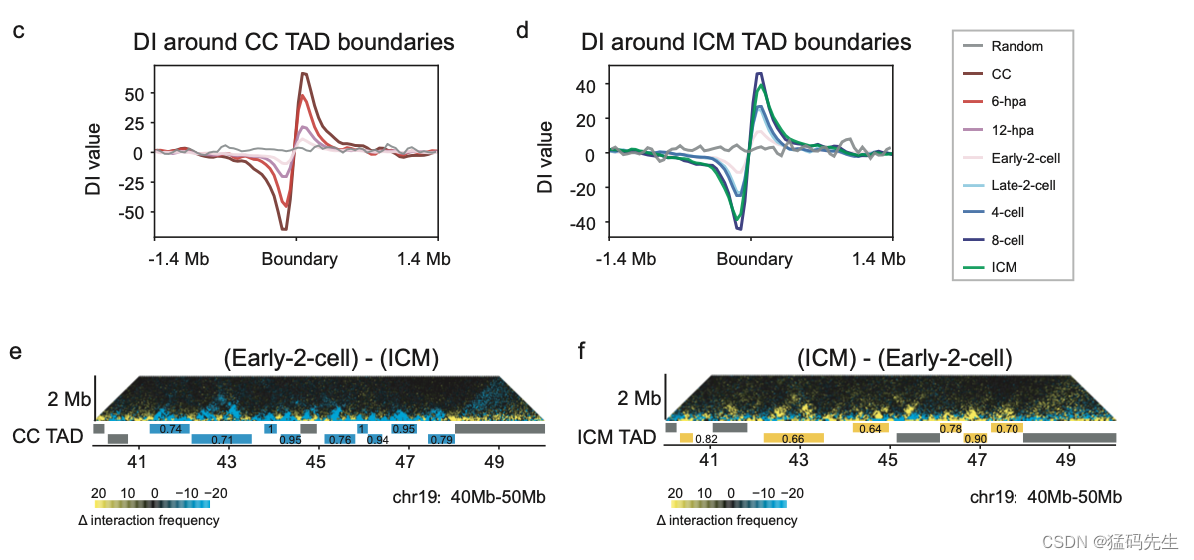
TAD是另一种典型的高阶染色质结构,具有“自结合”和“绝缘”特性。为了调查CC中的TAD在SCNT胚胎发育过程中是如何溶解和重新建立的,我们使用之前描述的绝缘评分确定了CC中的2516个TAD边界和ICM中的1941TAD边界。Hi-C相互作用热图显示,在注入去核卵母细胞后,远端(>2 Mb)染色质相互作用和CCs中的TAD都迅速溶解。染色质相互作用在1-hpi和1-hpa阶段是位点独立和均匀的,TAD也完全溶解(补充图4a-b)。这些结果描述了中期染色质相互作用的特征模式。TAD和稀疏远端相互作用都出现在6hpa阶段。高阶结构的出现是由于退出了中期样染色质状态,因为溶解的TAD可以在中期阶段完成后逐渐再生。6hpa阶段新出现的TAD比CC中的TAD弱,并继续减弱,直到早期-2细胞阶段,然后重建。一旦TAD建立,它们在后期阶段是一致的(图3b,补充图4a-f)。这些数据表明,在SCNT胚胎发育期间,TAD被去除并重新建立。
6.SCNT胚胎的异常染色质结构组织
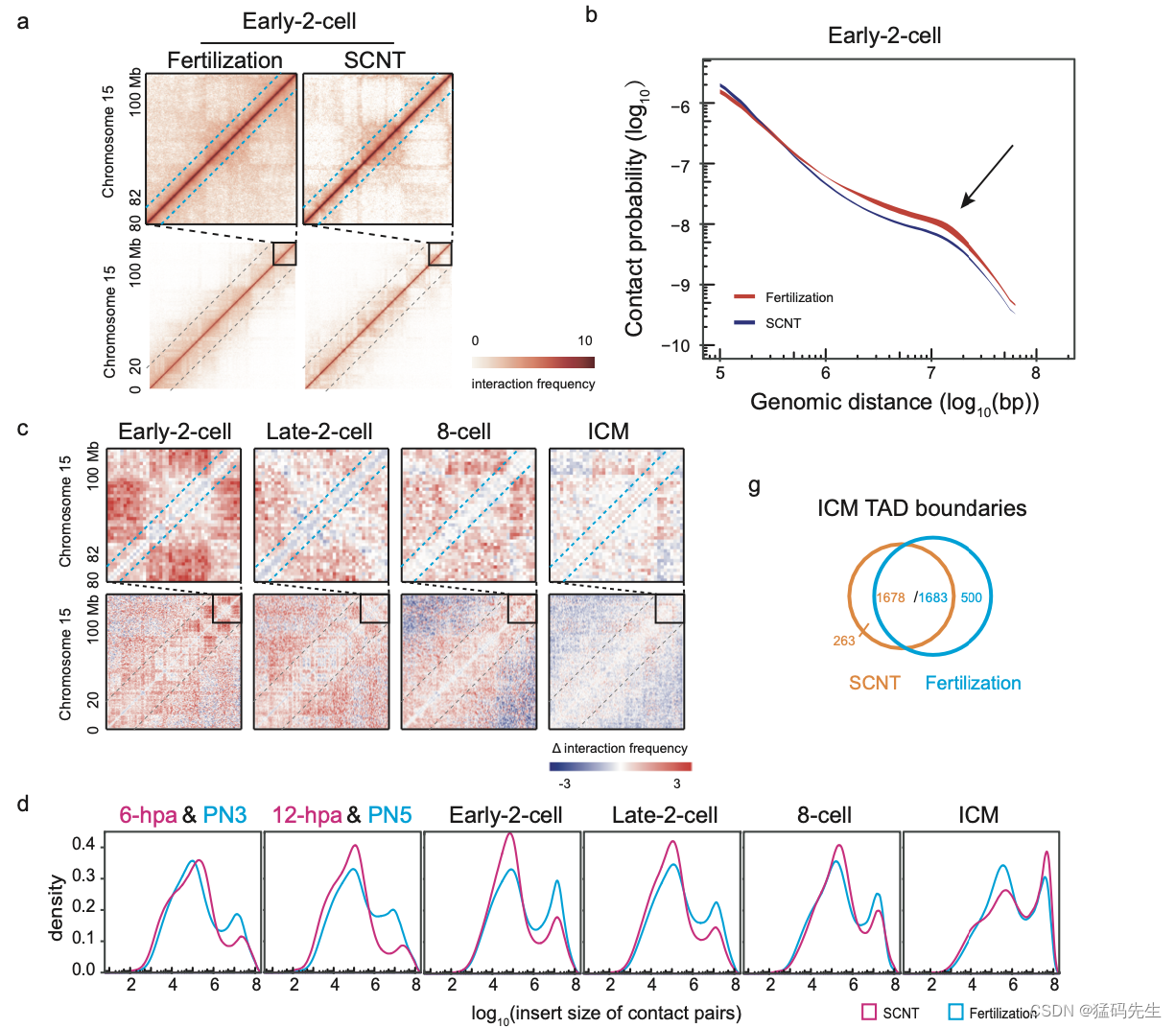
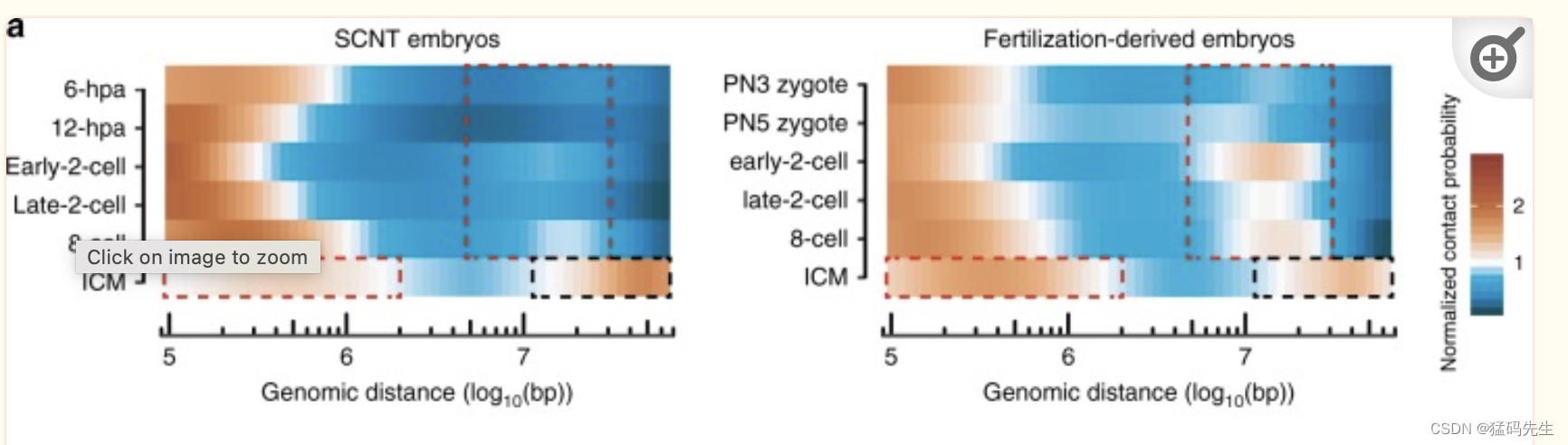
比较SCNT和受精衍生胚胎之间不同的染色质结构重新编程模式是有趣的,这应该有助于它们差异的胚胎发育潜力。我们首先比较了早期2细胞胚胎中SCNT和受精之间的相互作用热图。SCNT胚胎的域间区域分离比受精衍生胚胎更明显。此外,远端(>2 Mb)相互作用在SCNT胚胎中也较弱(补充图5a)。然后,我们比较了来自Hi-C数据集所有独立复制的SCNT和受精衍生的早期-2细胞胚胎的P(s)曲线。始终如一,在受精衍生胚胎中,远端接触概率更高。请注意,在10 Mb距离上有一个转折点,使受精衍生胚胎和SCNT胚胎之间的接触概率差异最大化(补充图5b)。为了排除距离效应,我们进一步归一化了参考曲线P(s)~s-1的相对接触概率。我们发现,在SCNT胚胎发育的早期阶段(6-hpa和12-hpa)远端(>2 Mb)相互作用的频率低于受精衍生胚胎(PN3和PN5)的频率。这种差异在早期-2细胞、晚期-2细胞和8细胞阶段变得明显。在ICM阶段,SCNT胚胎中的超长距离(>20 Mb)相互作用比受精衍生胚胎的相互作用更丰富,而近端相互作用则呈现出相反的趋势(图4a)。这种差分时空染色质包装也得到了相互作用读对数量的证实(补充图5c,d)。
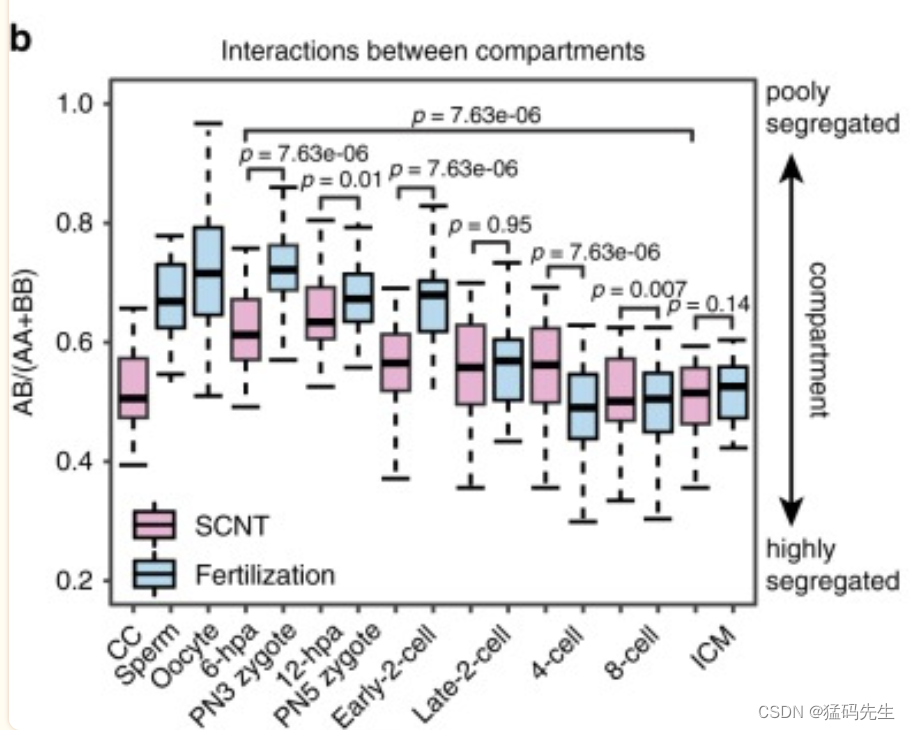
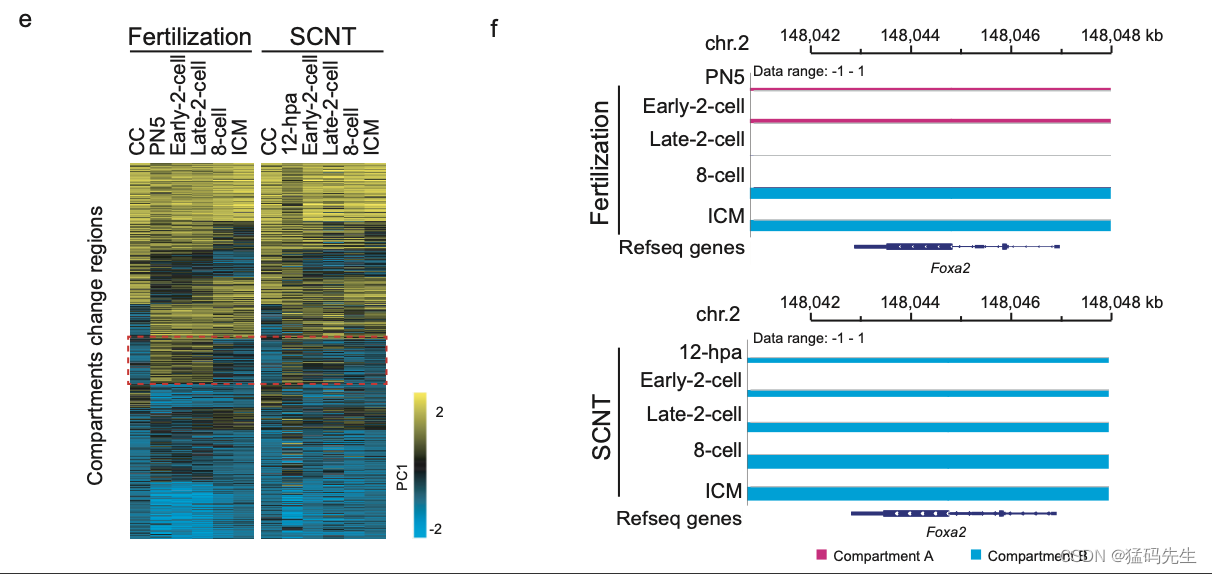
接下来,我们比较了SCNT和受精衍生胚胎之间的染色质室相互作用。虽然在发育过程中,A和B区段在SCNT和受精衍生胚胎中变得更加分离,但A和B区在受精衍生胚胎中的分离程度比在早期-2细胞阶段更差,在SCNT胚胎中(图4b),这可能是因为CC中发生的隔间相互作用比精子和卵母细胞少得多。然后,我们根据胚胎发育阶段的第一组分(PC1)值对区域进行聚类,并确定了一组在受精衍生胚胎发育的早期阶段(PN5,2细胞)在A组中活跃,但在SCNT胚胎发育早期阶段在B组中具有抑制性的区域(补充图5e)。之前的一项研究发现,在SCNT 2细胞畛胚胎中,包括Foxa2,有一组基因没有被激活。我们还发现,Foxa2的分区状态没有在SCNT胚胎的2细胞阶段重新编程到A分区(补充图5f)。因此,ZGA过程在SCNT胚胎发育期间可能有缺陷。
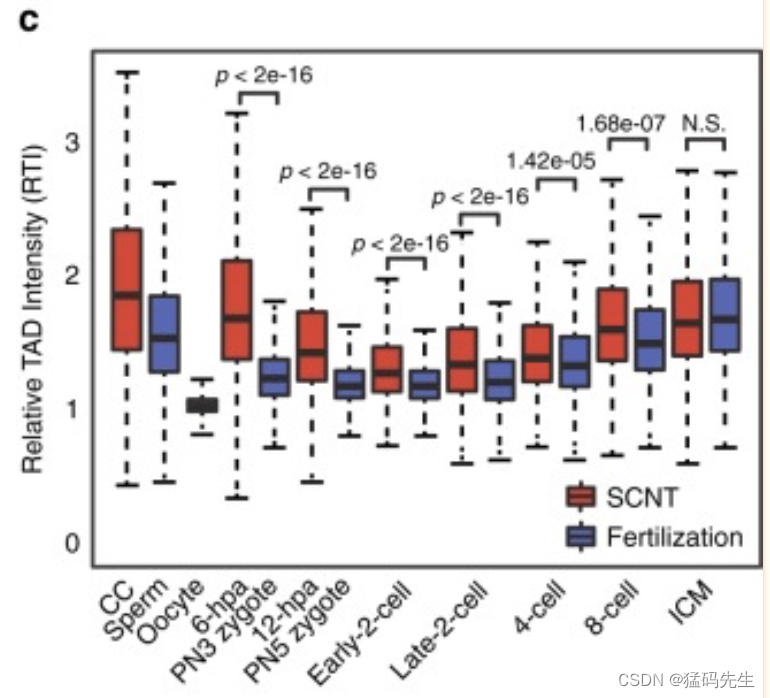
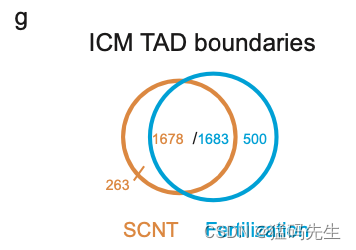
接下来,我们研究了受精衍生胚胎和SCNT胚胎之间TAD的差异。总体趋势是,在受精衍生和SCNT胚胎发育期间,TAD在ICM中变得成熟(图4c)。然而,在8细胞阶段之前,SCNT胚胎的TAD结构比受精衍生胚胎更强。在胚胎发育过程中,TAD强度差异逐渐减弱。TAD的差异也可能归因于CC中的TAD比精子和卵母细胞中的TAD更强。值得注意的是,SCNT和受精衍生的ICM之间仍然有许多不同的TAD(补充图5g)。总而言之,这些结果表明,SCNT和高阶染色质结构和受精衍生胚胎在早期阶段(如2细胞阶段)有很大差异,并在胚胎发育过程中变得越来越相似。
7.SCNT胚胎中ZGA基因的SE-P相互作用
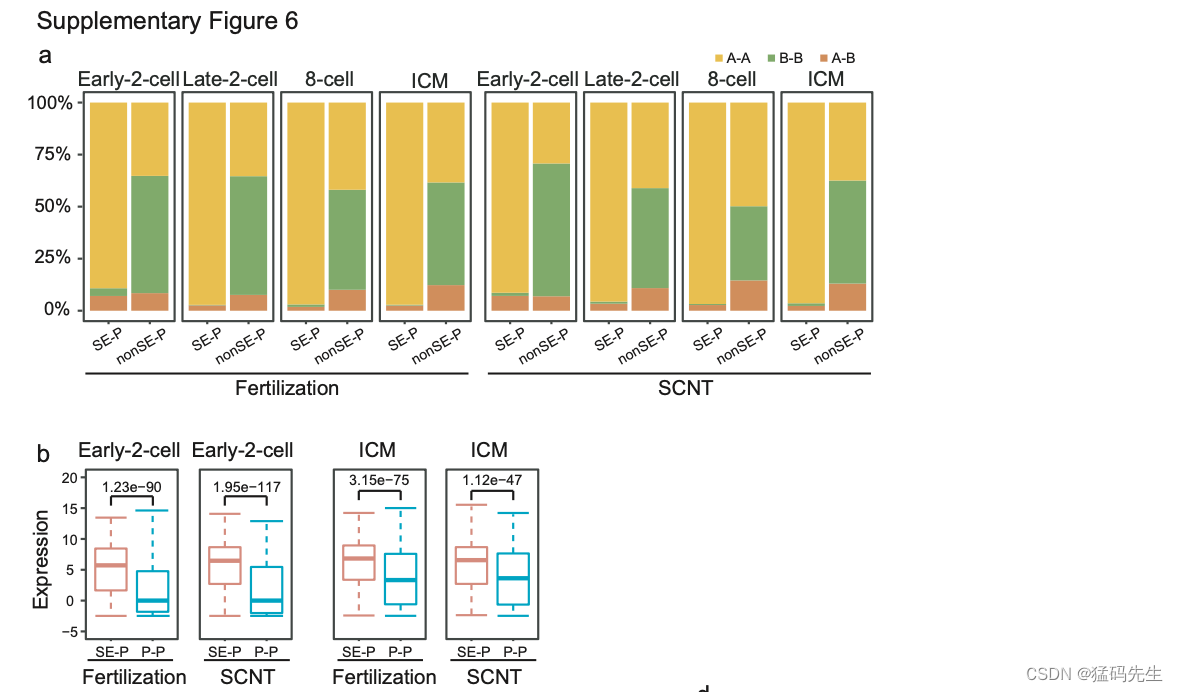
高阶染色质结构在基因调控中起着重要作用。基因可以通过位于同一TAD中的调节元素进行调节,例如典型的增强剂。此外,超级增强剂(SEs)是一个由典型增强剂聚集的大型结构域,与高密度的转录因子和共活化剂结合,并通过形成长距离相互作用来调节基因表达。因此,我们推测,这种差分染色质包装影响了超级增强剂和启动子之间的相互作用。为了验证这一假设,我们首先确定了之前描述的超级增强剂(详情请参阅方法)。接下来,我们使用Fit-HiC工具将超级增强剂和启动子之间的染色质相互作用归类为SE-P相互作用,将其余相互作用归类为非SE-P相互作用。在SCNT和受精衍生的胚胎发育期间,超过90%的SE-P相互作用发生在A室,而大约50%的非SE-P相互作用发生在B室(补充图6a)。此外,与SE-P相互作用相关的靶基因的表达水平明显高于与非SE-P相互作用相关的基因,如启动子-促进子(P-P)相互作用(补充图6b)。这些结果表明,本研究中确定的SE是可靠的。

进一步分析表明,受精衍生胚胎和SCNT胚胎之间的SE-P相互作用在发育过程中增加。(补充图6c)。这表明,SCNT胚胎的高阶染色质结构在发育过程中与受精衍生胚胎的结构更加相似。然后,我们确定了所有由SE-P回路调节并在受精衍生的ICM中激活的基因,但缺乏SE-P回路,并在SCNT ICM中被抑制。对这些基因的基因本体分析确定了基本生物过程的丰富性,如蛋白质运输、DNA模板转录、细胞对DNA损伤刺激的反应和细胞-细胞粘附(补充图6d)。这一结果意味着,SCNT胚胎发育期间差异染色质包装导致的异常SE-P相互作用广泛影响了生物过程,并可能导致SCNT胚胎发育率低。
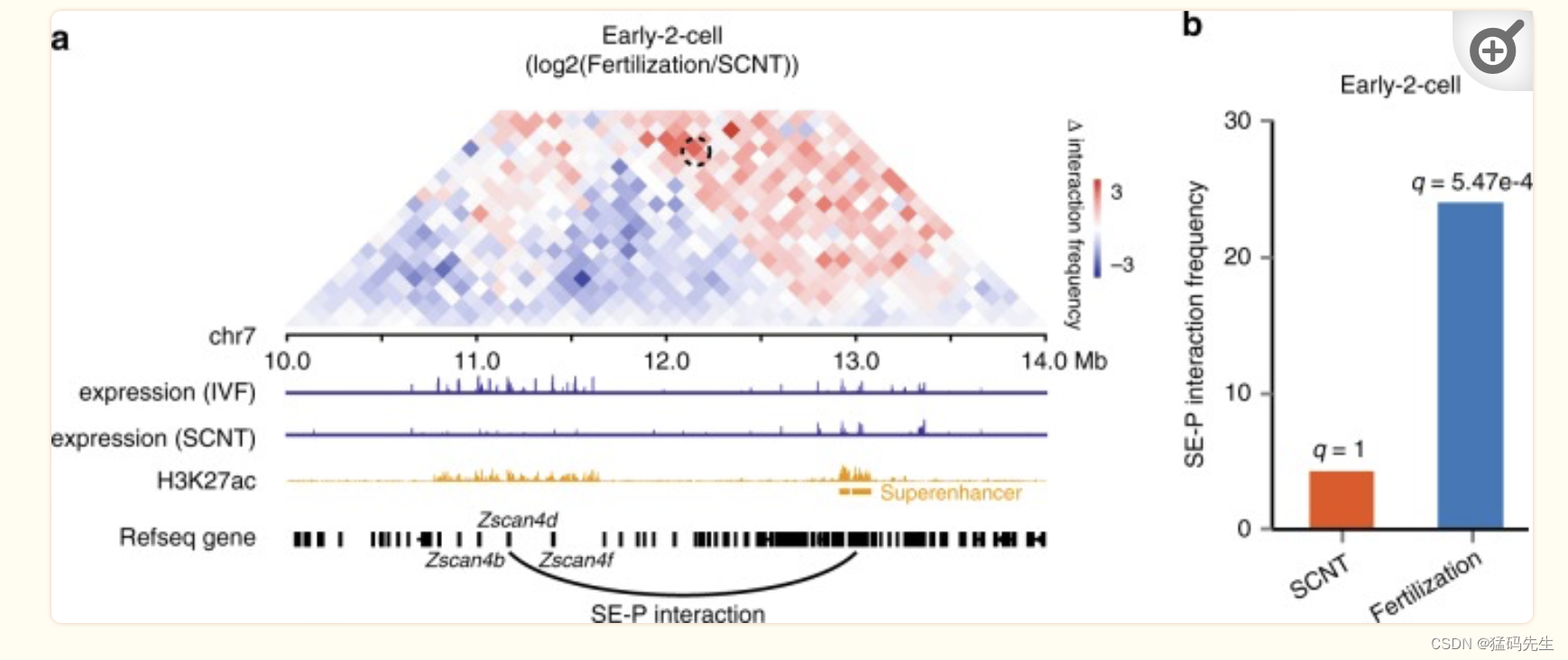
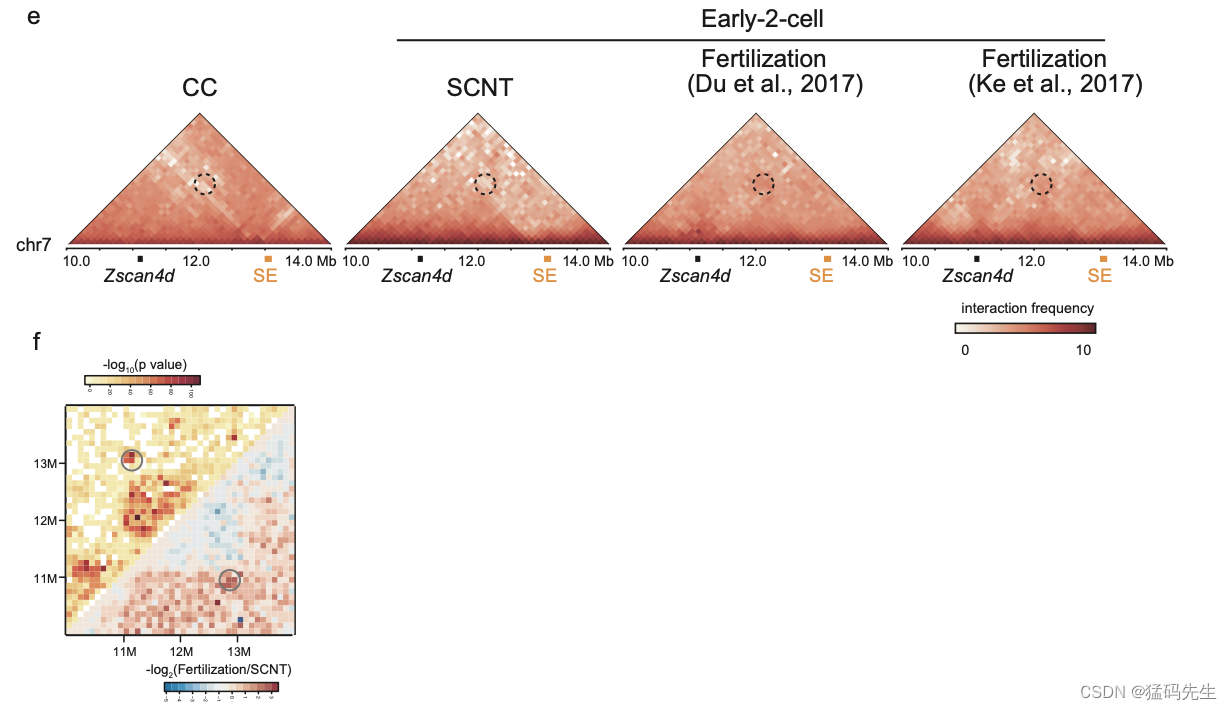
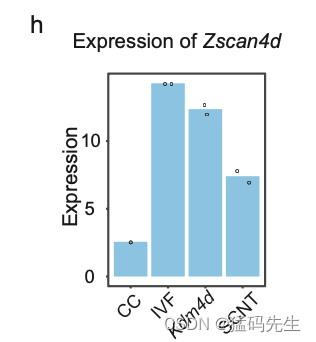
ZGA是早期胚胎发育过程中的一个关键事件。为了研究染色质组织和ZGA之间的关系,我们专注于早期-2细胞阶段的差异性SE-P相互作用,并发现Zscan4d(~2 Mb)位点的SE-P相互作用在受精衍生的2细胞胚胎中很明显,但在SCNT早期-2细胞胚胎中不存在(图5a,b;补充图6e)。我们进一步使用了FIND工具37分析这些位点周围的差异染色质相互作用,并验证这一差异(补充图6f)。Zscan4d基因在2细胞阶段特异性表达,对植入前发育至关重要,但它在SCNT 2细胞胚胎中被沉默了11。这一结果意味着,SCNT胚胎发育过程中异常的SE-P相互作用可能是异常基因激活和低胚胎发育率的原因。
8.H3K9me3是染色质结构重新编程的障碍
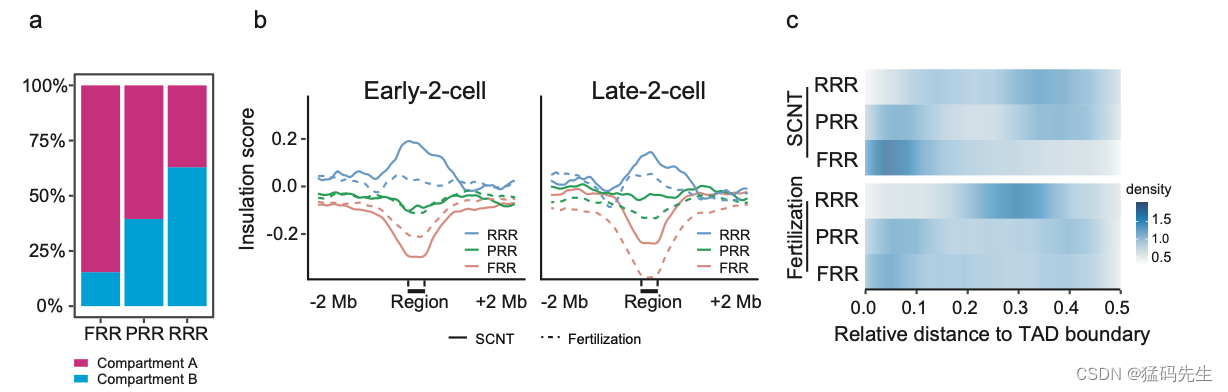
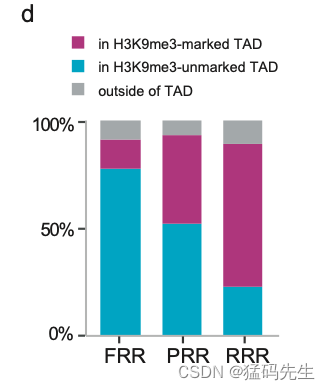
最后,我们试图探索SCNT胚胎发育期间染色质组织差异重新编程的潜在机制。之前的一项研究确定了SCNT胚胎中的三种区域:RRR(抗重新编程区域)、PRR(部分重新编程区域)和FRR(完全重新编程区域)。有趣的是,我们发现RRR在SCNT胚胎的B室中显著丰富(补充图7a)。此外,RRR的绝缘分数明显高于PRR和FRR(补充图7b)。在SCNT和受精衍生胚胎中,比PRR或FRR更远离边界位置的RRR(补充图7c)。这些结果表明,RRR更有可能居住在TAD中,而不是边界内。由于供体细胞基因组中的H3K9me3富集有助于RRRs的重新编程缺陷,TAD边界有可能限制抑制性染色质向活性域的传播,我们假设与H3K9me3相关的TAD结构在SCNT胚胎发育期间可能表现出高阶染色质结构重新编程缺陷。为了验证这一假设,我们首先在CC中确定了H3K9me3标记和H3K9me3未标记的TAD(见方法)。RRR在H3K9me3标记的TAD中比在FRR和PRR中更丰富(补充图7d)。

我们还发现,H3K9me3标记的CC TAD在SCNT胚胎中未能分解,尽管在受精衍生胚胎的相应区域没有观察到TAD。相比之下,H3K9me3无标记的CC TAD在SCNT胚胎中迅速溶解(图6a)。尽管这两种类型的TAD在CC中具有相似的相对TAD强度(RTI),但H3K9me3标记TAD的强度仍然明显高于SCNT后H3K9me3未标记TAD(图6b)。
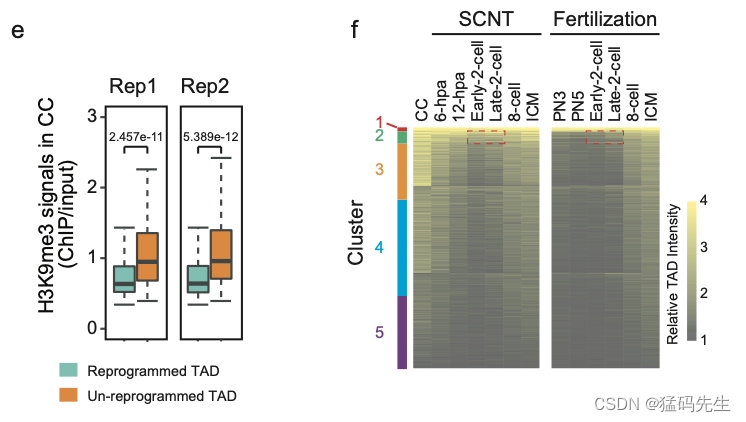
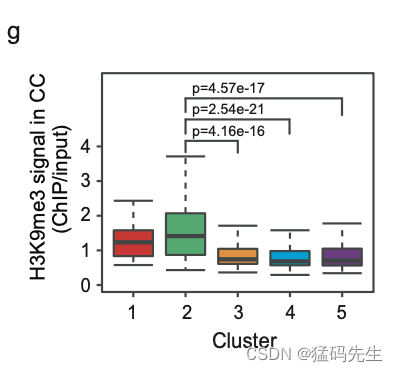
我们进一步比较了SCNT 12-hpa胚胎和体外受精(IVF)PN5合子之间TAD的RTI值,并定义了两种类型的TAD:重新编程的TAD和未重新编程的TAD(见方法)。CC基因组中的H3K9me3信号在未重新编程的TAD中明显高于重新编程的TAD(补充图7e)。最后,我们根据SCNT和受精衍生胚胎的发育阶段的RTI值,将TAD集中在受精衍生的ICM中,这些ICM处于“成熟”状态。TAD(C2)簇在SCNT 2细胞胚胎中具有较强的RTI值,但在体外受精2细胞胚胎中具有较低的RTI值(补充图7f)。因此,这些TAD在SCNT 2细胞胚胎中基本上没有重新编程。巧合的是,这些TAD(C2)在CC基因组中的H3K9me3信号明显高于在SCNT胚胎中重新编程的其他TAD簇(C3-C5)(补充图7g)。
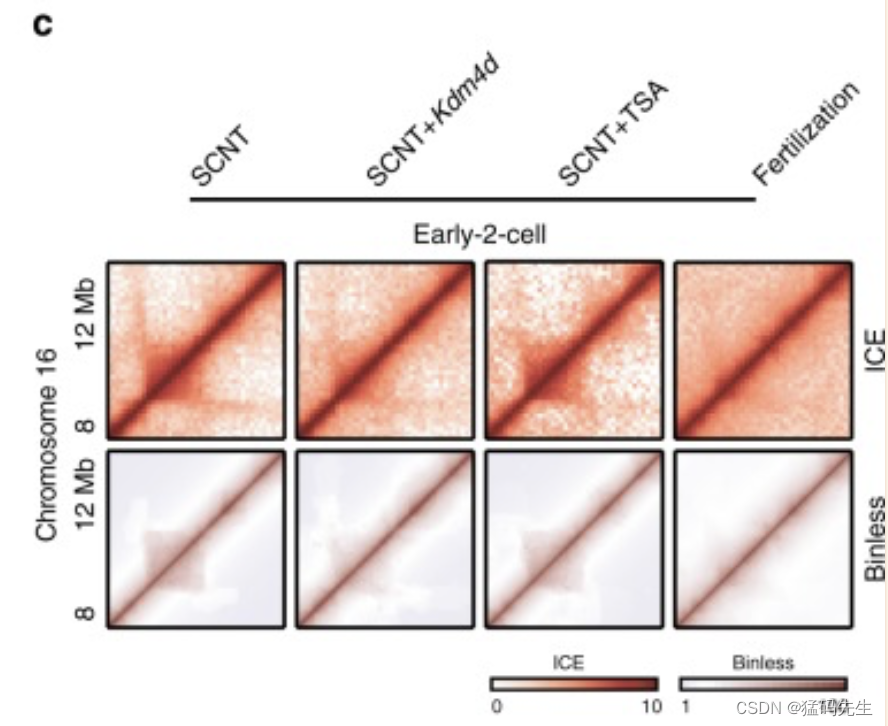
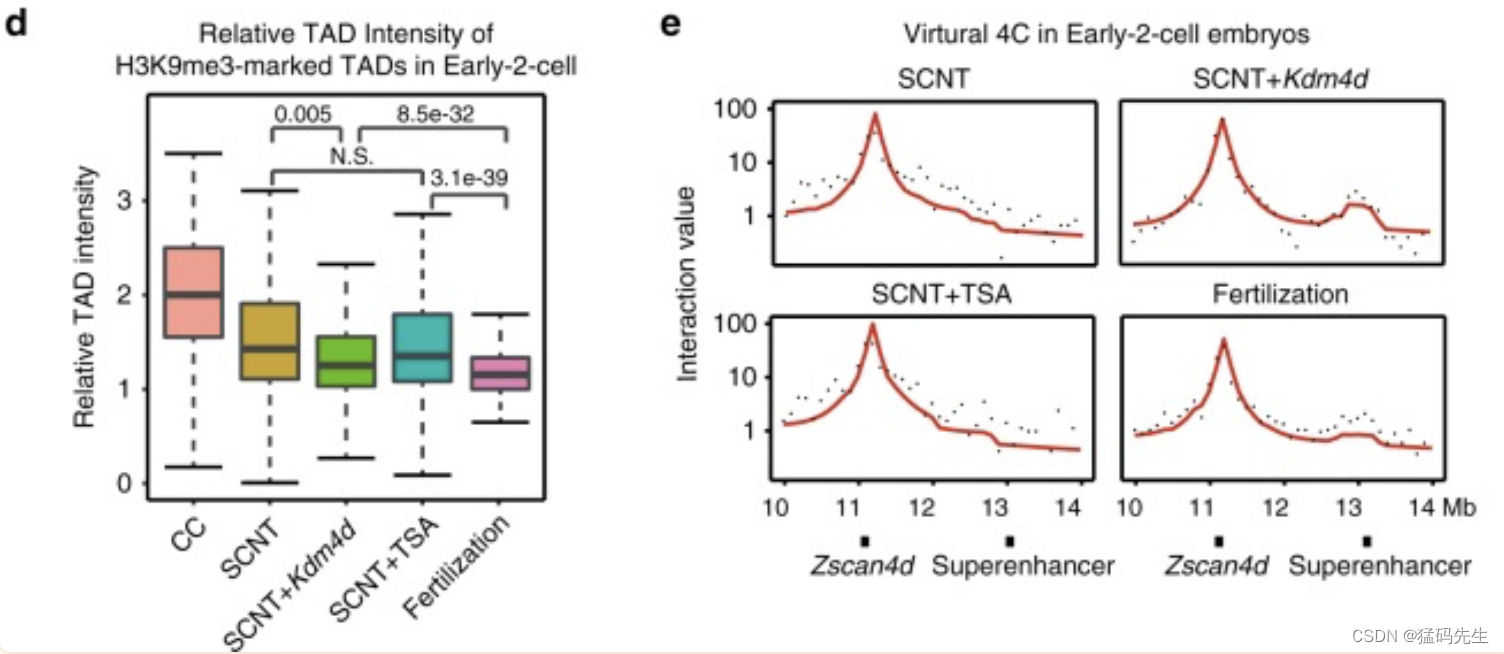
为了验证H3K9me3保留对TAD重新编程的影响,我们将Kdm4d mRNA、H3K9me3脱甲基酶注射到SCNT胚胎和组蛋白脱乙酰酶抑制剂Trichostatin A(TSA)中,治疗也作为对照进行。交互热图显示SCNT-Kdm4d组的TAD信号比SCNT或SCNT-TSA组弱(图6c)。框图显示,与SCNT和SCNT-TSA组相比,SCNT-Kdm4d组的H3K9me3标记TAD的RTI值显著下降,但这些值仍然高于受精衍生胚胎的RTI值(图6d)。另一方面,我们发现Zscan4d位点的SE-P相互作用在SCNT 2细胞胚胎中丢失,并在SCNT-Kdm4d胚胎中恢复(图6e;补充图7h)。这可能是由于在Kdm4d过表达后,它们之间相对未整合的染色质边界结构。这一结果意味着CC基因组中的H3K9me3富集是一个表观遗传屏障,阻碍了SCNT介导的染色质结构的重新编程,注射Kdm4d mRNA可以部分拯救SCNT早期-2细胞胚胎中未解构的H3K9me3标记的TAD。





















 1119
1119











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








