一、案例介绍
本项目旨在对鸢尾花数据集进行于混合高斯模型的聚类,并且深入分析与探索,以揭示数据的潜在模式与内在关联。通过对数据进行预处理,包括但不限于处理缺失值、异常值、标准化等操作,我们将确保数据的准确性和一致性。随后,将运用探索性数据分析(EDA)方法,结合数据可视化技术,呈现鸢尾花数据的多维特征,探索不同特征之间的关系和分布情况。
二、理论方法
这里就简单介绍,我们以运用为主:
聚类(Clustering)是按照某个特定标准(如距离)把一个数据集分割成不同的类或簇,使得同一个簇内的数据对象的相似性尽可能大,同时不在同一个簇中的数据对象的差异性也尽可能地大。也即聚类后同一类的数据尽可能聚集到一起,不同类数据尽量分离。
具体步骤:
1. 选择簇的数量(与K-Means类似)并随机初始化每个簇的高斯分布参数(均值和方差)。也可以先观察数据给出一个相对精确的均值和方差。
2. 给定每个簇的高斯分布,计算每个数据点属于每个簇的概率。一个点越靠近高斯分布的中心就越可能属于该簇。
3. 基于这些概率我们计算高斯分布参数使得数据点的概率最大化,可以使用数据点概率的加权来计算这些新的参数,权重就是数据点属于该簇的概率。
4. 重复迭代2和3直到在迭代中的变化不大
EM算法的基本流程:
1.隐变量模型: EM 算法通常用于解决包含隐含变量的概率模型参数估计问题,其中观测数据不完整或包含隐含未知信息。
2.期望步骤(E步): 在 E 步中,对于给定的模型参数估计值,计算隐含变量的后验概率分布,即给定观测数据的条件下,隐含变量的概率分布。
3.最大化步骤(M步): 在 M 步中,利用 E 步中计算得到的隐含变量的概率分布,更新模型参数,使得在当前参数估计下观测数据的对数似然函数达到最大化。
4.迭代更新: 交替进行 E 步和 M 步,直到模型参数收敛或达到预设的停止条件为止。
三、案例分析
library(mclust)
# 读取名为 "file_name.csv" 的 CSV 文件
data1 <- read.csv("data1.csv")
data1
# 显示读取的数据框
print(data1)
head(data1,5)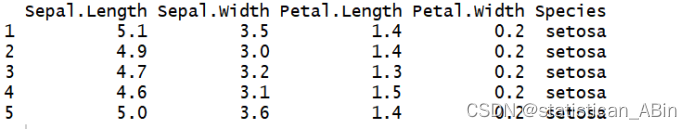
鸢尾花数据集是一个经典的机器学习数据集,其中包含了鸢尾花的四个特征。这些特征是:
萼片长度(Sepal Length): 指的是鸢尾花的萼片(sepal)的长度,即从基部到尖端的长度。
萼片宽度(Sepal Width): 表示鸢尾花的萼片的宽度,在宽度最宽的地方测量。
花瓣长度(Petal Length): 指的是鸢尾花的花瓣(petal)的长度,即从基部到尖端的长度。
花瓣宽度(Petal Width): 表示鸢尾花的花瓣的宽度,在宽度最宽的地方测量。
接下来检查数据集中的缺失值情况并且可视化
# 检查数据集中的缺失值情况
print(colSums(is.na(data1))) # 列出每列缺失值的数量
# 使用 VIM 包中的函数可视化缺失值情况
# 需要安装并加载 VIM 包
library(VIM)
# 创建缺失值的可视化图
aggr(data1, col=c('blue', 'red'), numbers=TRUE, sortVars=TRUE, labels=names(data), cex.axis=.7, gap=3)
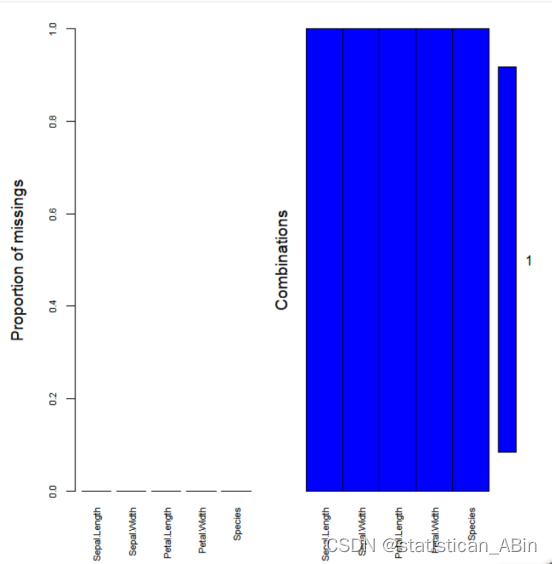
从上面可以看出,该数据由于比较“干净”,不存在缺失值,但是平时在使用其他数据集时是要必须检查的,这将会对模型最终预测结果产生影响。接下来对数据集进行可视化探索:
绘制散点图,使用散点图可以展示不同类别(鸢尾花的不同种类)在不同特征之间的分布
# 绘制散点图,使用散点图可以展示不同类别(鸢尾花的不同种类)在不同特征之间的分布。
ggplot(data1, aes(x = Sepal.Length, y = Sepal.Width, color = Species)) +
geom_point() +
labs(title = "Sepal Length vs Sepal Width by Species")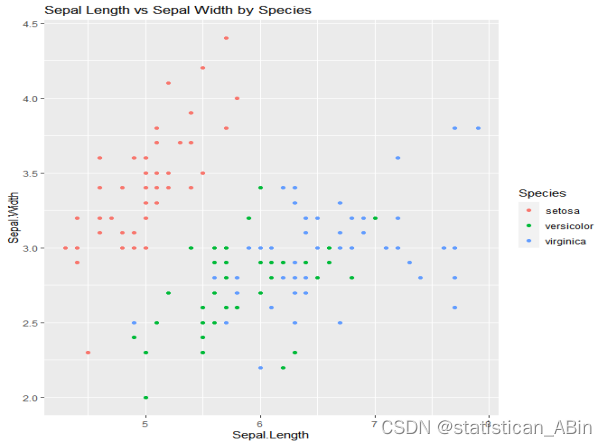
从上面可以看出,三种不同类型的鸢尾花由三种不同颜色表示。
接下来使用箱线图用来展示不同类别在单个特征上的分布情况。
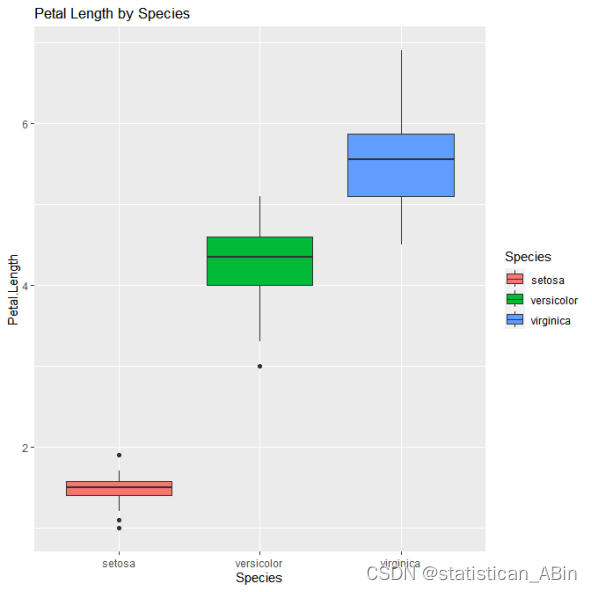
接下来绘制直方图和蜂群图来查看不同种类的花的其他类型:
#直方图可以显示特征的分布情况。
# 绘制直方图
ggplot(data1, aes(x = Petal.Width, fill = Species)) +
geom_histogram(binwidth = 0.1, position = "identity", alpha = 0.7) +
labs(title = "Petal Width Distribution by Species")
#蜂群图可以更直观地展示多个特征的分布。
# 绘制蜂群图\
library(ggbeeswarm)
ggplot(data1, aes(x = Species, y = Petal.Width, color = Species)) +
geom_beeswarm() +
labs(title = "Petal Width by Species")
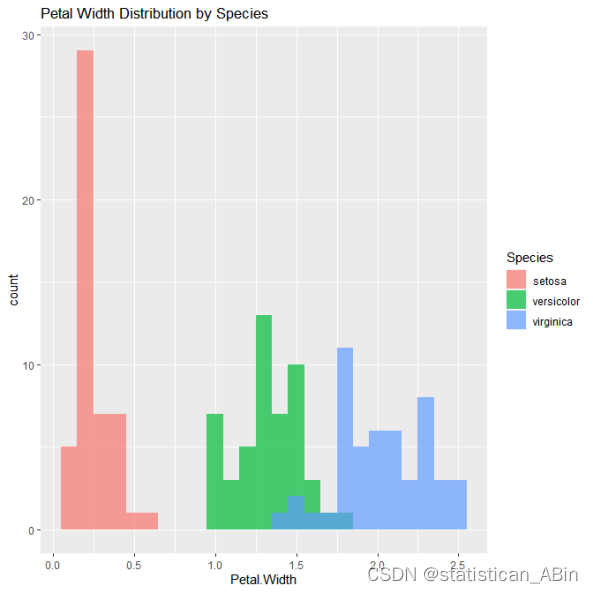
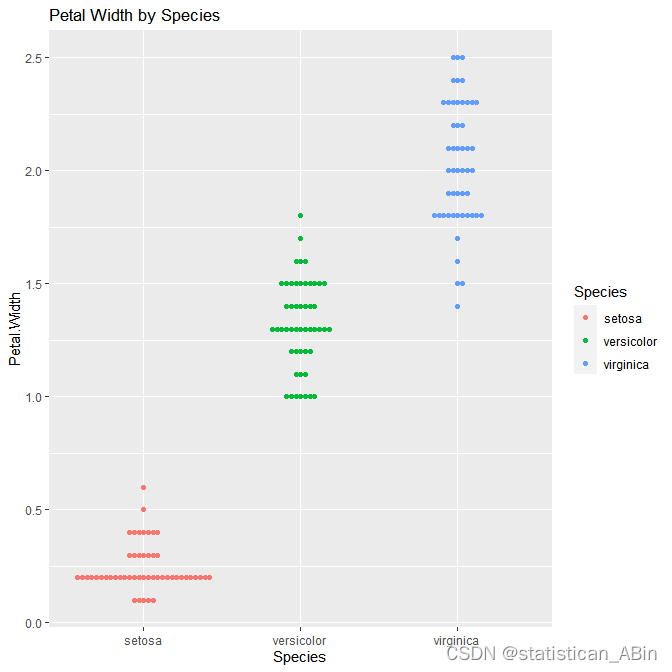
从这里可以看出,蜂群图和箱线图大体上是类似的,只是样式不同。接下来可以具体探索一些特征直接的关系,比如花萼与花瓣的长度和宽度之间的关系
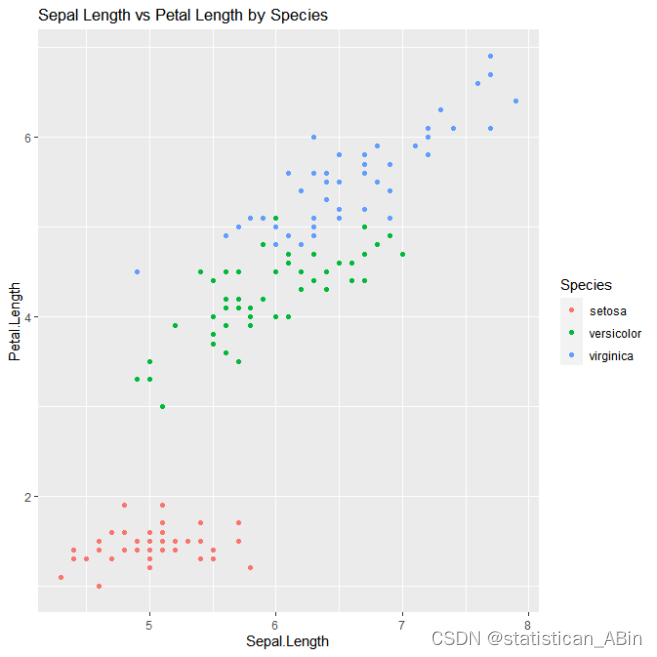
从上面图中可以看见,蓝色和绿色两类花的花萼与花瓣的长度和宽度总体上是呈现正相关的,而setosa则不是。接下来使用热力图展示特征之间的相关性。
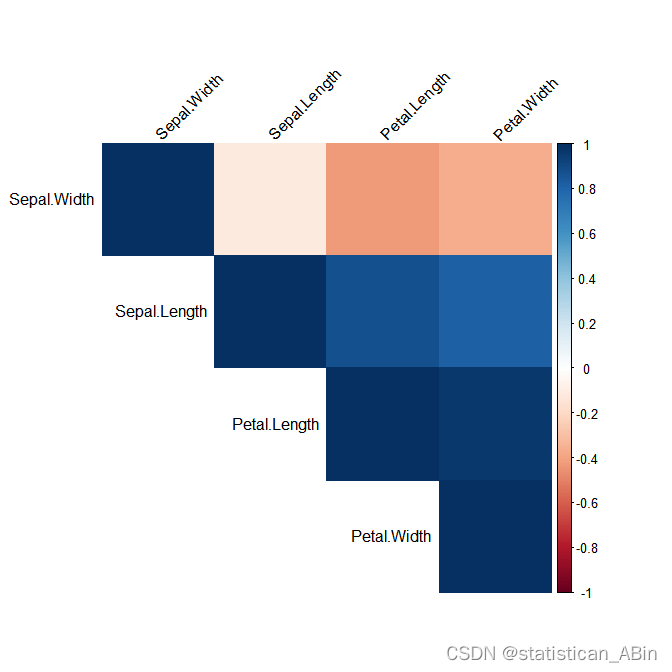
# 计算相关性矩阵
cor_matrix <- cor(iris[, 1:4])
# 使用 corrplot 包绘制相关性热力图
install.packages("corrplot")
library(corrplot)
# 绘制相关性热力图
corrplot(cor_matrix, method = "color", type = "upper", order = "hclust", tl.col = "black", tl.srt = 45)
从相关系数图可以看出,两个特征越相关则颜色表示越深。例如Sepal.Length与Petal.Length相对来说是比较相关的,Sepal.Width与Petal.Length则呈现负相关。接下来正式进入EM混合高斯模型的聚类。
首先划分数据集,划分训练集和测试机,划分比例为70%:30%
#创建训练数据集和测试数据集
set.seed(30)
index <- sample(1:2, size = nrow(data1), replace = TRUE, prob = c(0.7,0.3))
train <- data1[index == 1,]
test <- data1[index == 2,]
#EM算法进行聚类
EM <- Mclust(data = train[,-5], G=3)
summary(EM)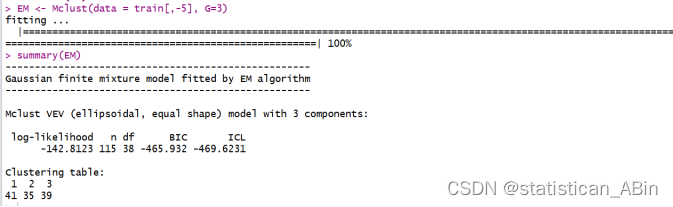
从上面可以看出,最终被分为了3类,其中这主要是上文的参数G决定的。接下来判断样本内的预测准确率:
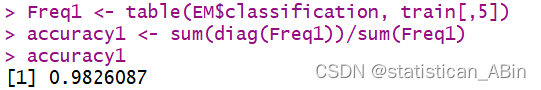
可以看出在训练集的分类准确率为0.9826

在测试集的分类准确率为0.9142,接下来可视化一下:
library(ggplot2)
# 创建数据框来存储准确率
accuracies_df <- data.frame(
Dataset = c("Training", "Testing"),
Accuracy = c(accuracy1, accuracy2)
)
# 创建柱状图
accuracy_plot <- ggplot(accuracies_df, aes(x = Dataset, y = Accuracy, fill = Dataset)) +
geom_bar(stat = "identity", position = "dodge", width = 0.5) +
labs(title = "Model Accuracy Comparison",
y = "Accuracy",
x = "Dataset") +
theme_minimal()
# 显示柱状图
print(accuracy_plot)四、总结
本研究利用 EM(Expectation Maximization)算法对鸢尾花数据集进行了聚类分析。在整个分析过程中,我们首先进行了数据预处理,包括缺失值处理、特征可视化等操作,以保证数据的质量和准确性。随后,我们运用 EM 算法,通过拟合混合高斯模型对鸢尾花数据集进行聚类。在训练集上,我们得到了较高的分类准确率(0.9826),表明模型在训练数据上有良好的拟合能力。而在测试集上,模型表现同样出色,得到了令人满意的分类准确率(0.9142),说明模型在未知数据上也具有较好的泛化能力























 533
533

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








