1写在前面
高考结束了,不知道各位考生考的怎么样,这种时候总是几家欢喜几家忧,但这也是实现阶级流动的最佳机会。🤔
回想自己高考过去10几年了,不能说学了医后悔吧,只能说后悔至极,苦不堪言啊,收入还少的可怜。🫠
真的劝各位学子,尽可能避免这类专业,如果有机会的话去看看外面的世界吧,还是和我们以为的差太多了。😘
最近在做多组的差异分析,分享一下我的code吧,因为是基于limma包,所以还是比较简单的,请放心食用。😋
2用到的包
rm(list = ls())
library(tidyverse)
library(limma)
library(GEOquery)
3示例数据
这里我们用之前从GEO数据库上down的一个dataset吧,在3个样本中对T细胞和B细胞分别进行了转录组分析。😘
GSE194314 <- getGEO('GSE194314', destdir=".",getGPL = F)
exprSet <- exprs(GSE194314[[1]])
4数据清洗
4.1 标准化处理
我们先进行数据的下载吧,boxplot不是很齐,标准化搞起来哦!🤨
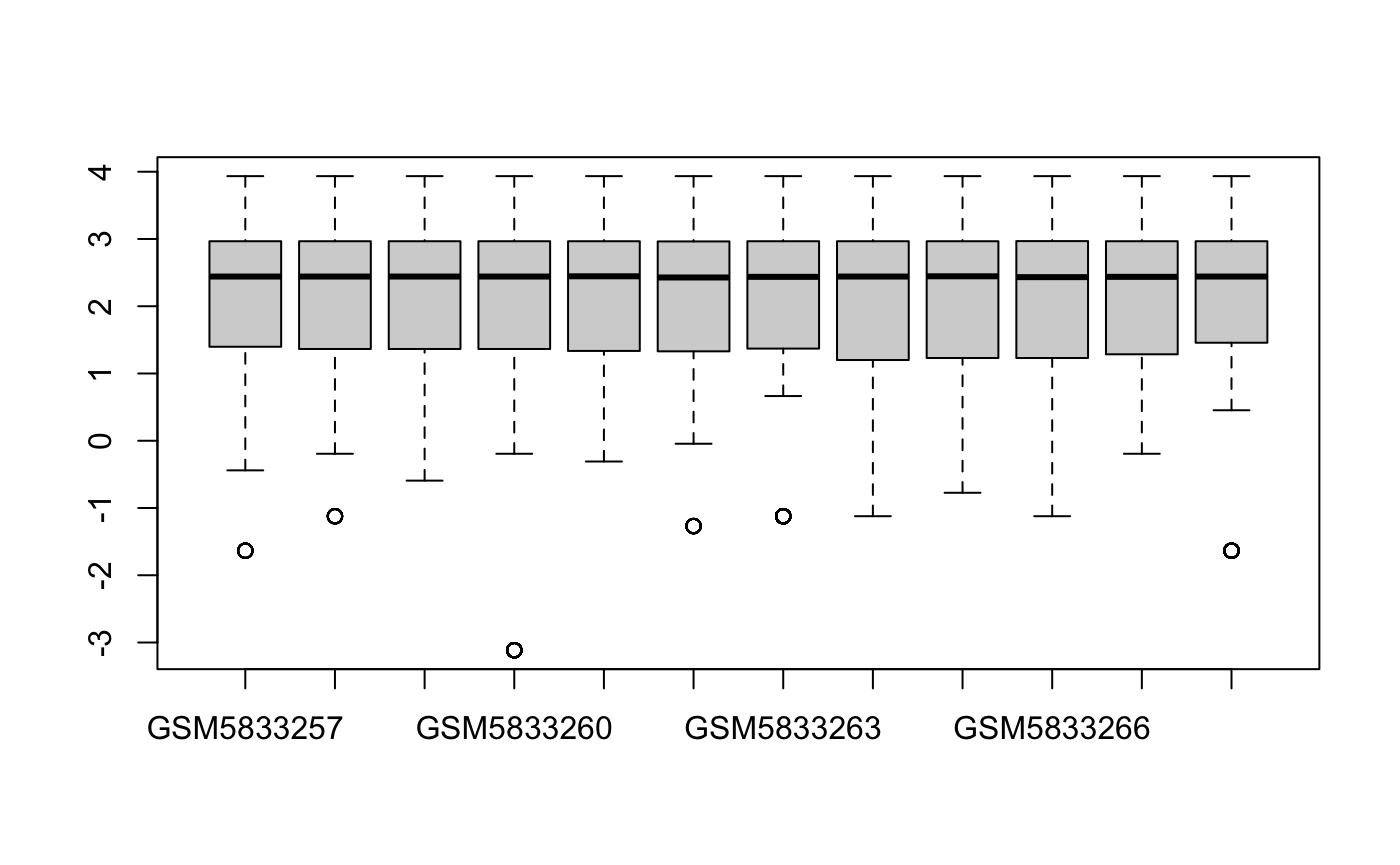
1️⃣ 标准化前
boxplot(log2(exprSet))
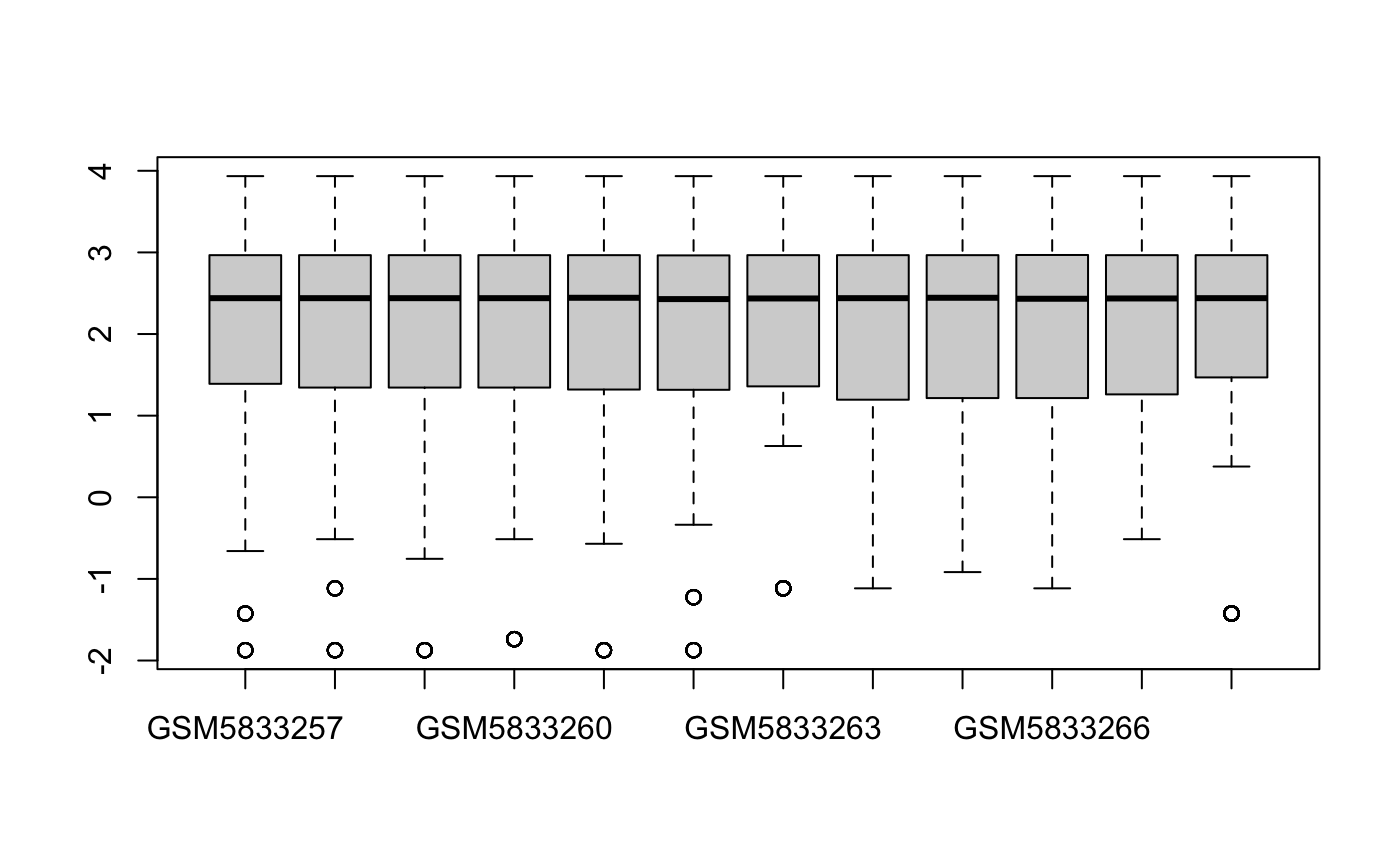
2️⃣ 标准化后
exprSet <- normalizeBetweenArrays(exprSet) %>%
log2(.)
boxplot(exprSet)
5获取分组数据
由于样本共有12个,我们认为的进行一下分组吧,分成3个组,每组4个样本。🥳
pdata <- data.frame(
sample = colnames(exprSet),
group = rep(c("A", "B", "C"), each = 4)
)
DT::datatable(pdata)
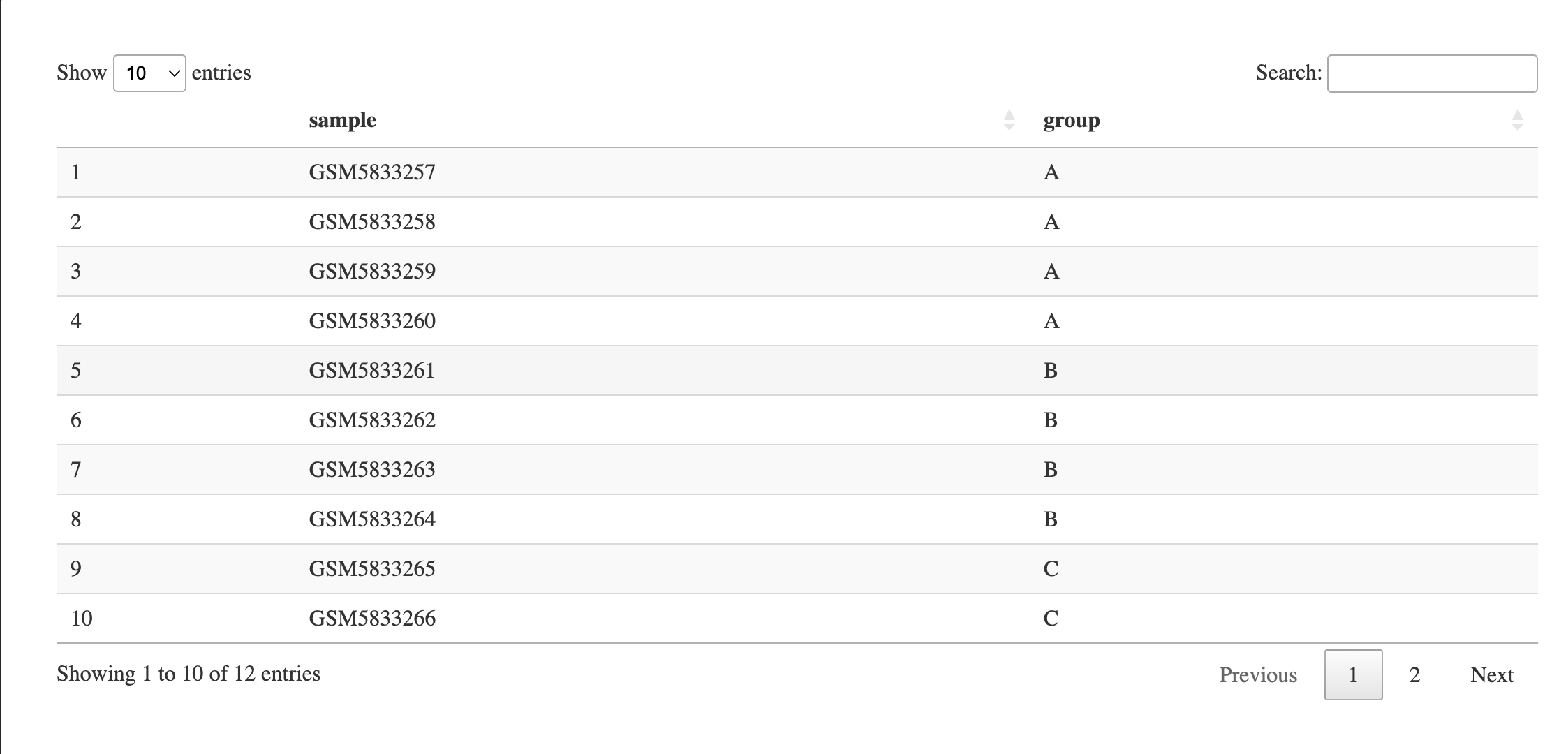
6整理分组数据
group <- factor(pdata$group,levels = unique(pdata$group))
7差异分析
7.1 整理design文件
#===========整理分组数据为分组矩阵
design <- model.matrix(~0 + group)
rownames(design) <- colnames(exprSet)
colnames(design) <- levels(group)
# 差异比较矩阵
cont.matrix <- makeContrasts("A vs B" = A-B,
"A vs C" = A-C,
"B vs C" = B-C,
levels = colnames(design)
)
cont.matrix

7.2 开始差异分析
#========== 定义阈值
logFCcutoff <- log2(0)
adjPvalueCutoff <- 0.3
#=========== 进行差异分析
fit <- lmFit(exprSet, design)
#=========== 针对给定的对比计算估计系数和标准误差
fit2 <- contrasts.fit(fit, cont.matrix)
fit2 <- eBayes(fit2)
plotSA(fit2)
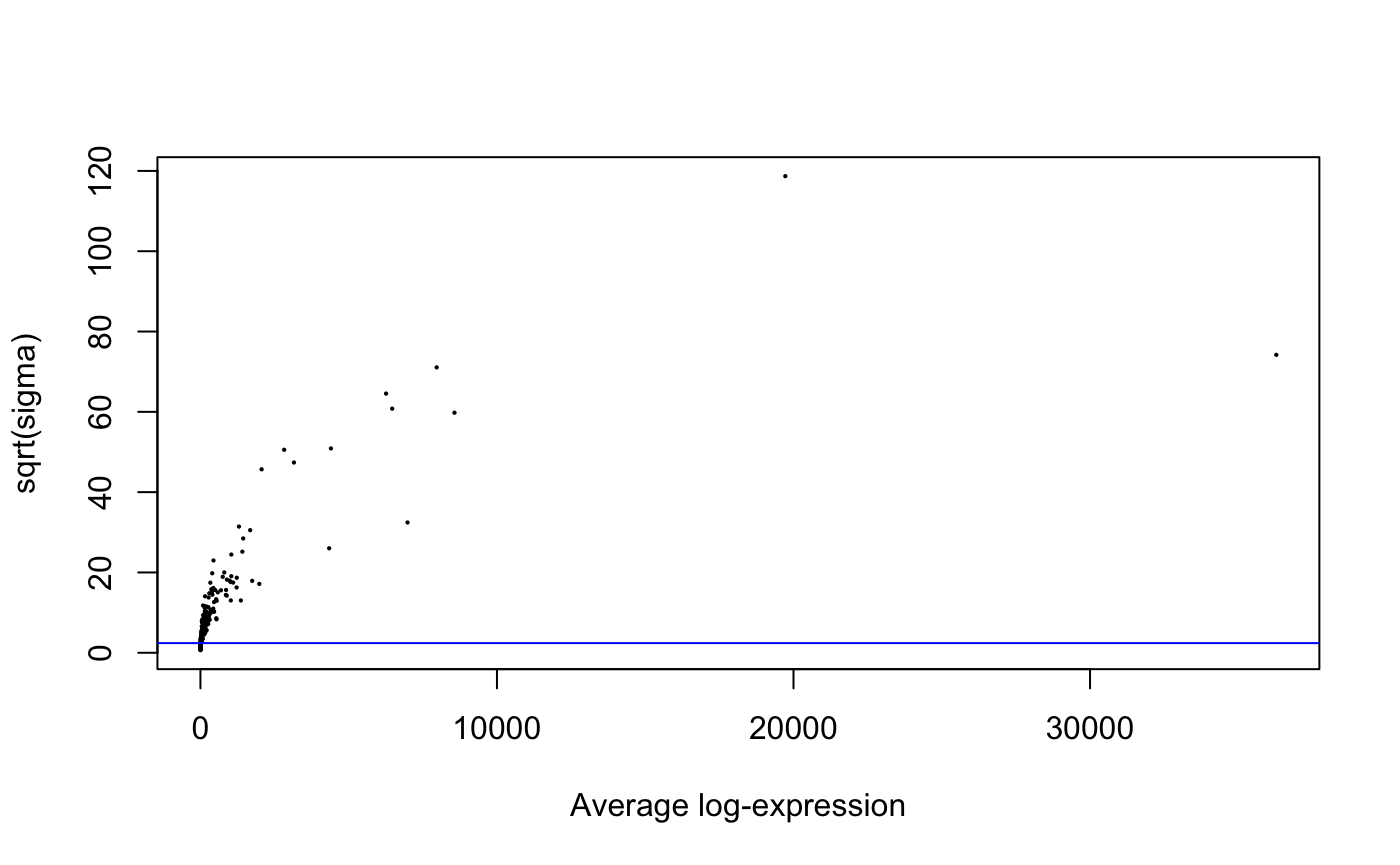
7.3 查看基因
summary(decideTests(fit2, p.value = adjPvalueCutoff, lfc = logFCcutoff))

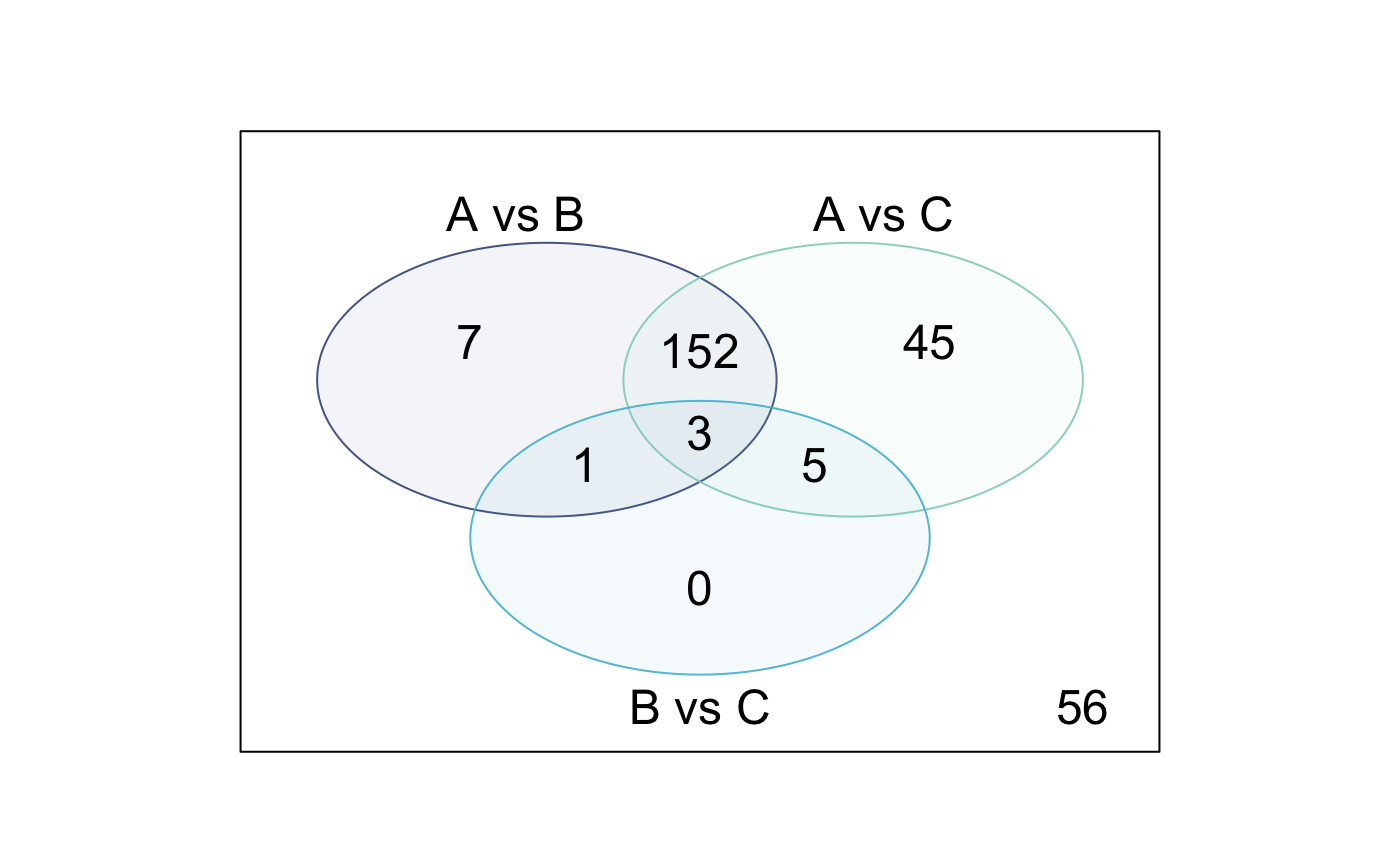
7.4 Venn图可视化差异基因
dt <- decideTests(fit2,p.value = adjPvalueCutoff, lfc = logFCcutoff)
de.common <- which(dt[,1]!=0 & dt[,2]!=0 & dt[,3]!=0)
length(de.common)
vennDiagram(dt[,1:3], circle.col=c("#3C5488E5", "#91D1C2E5", "#4DBBD5E5"))
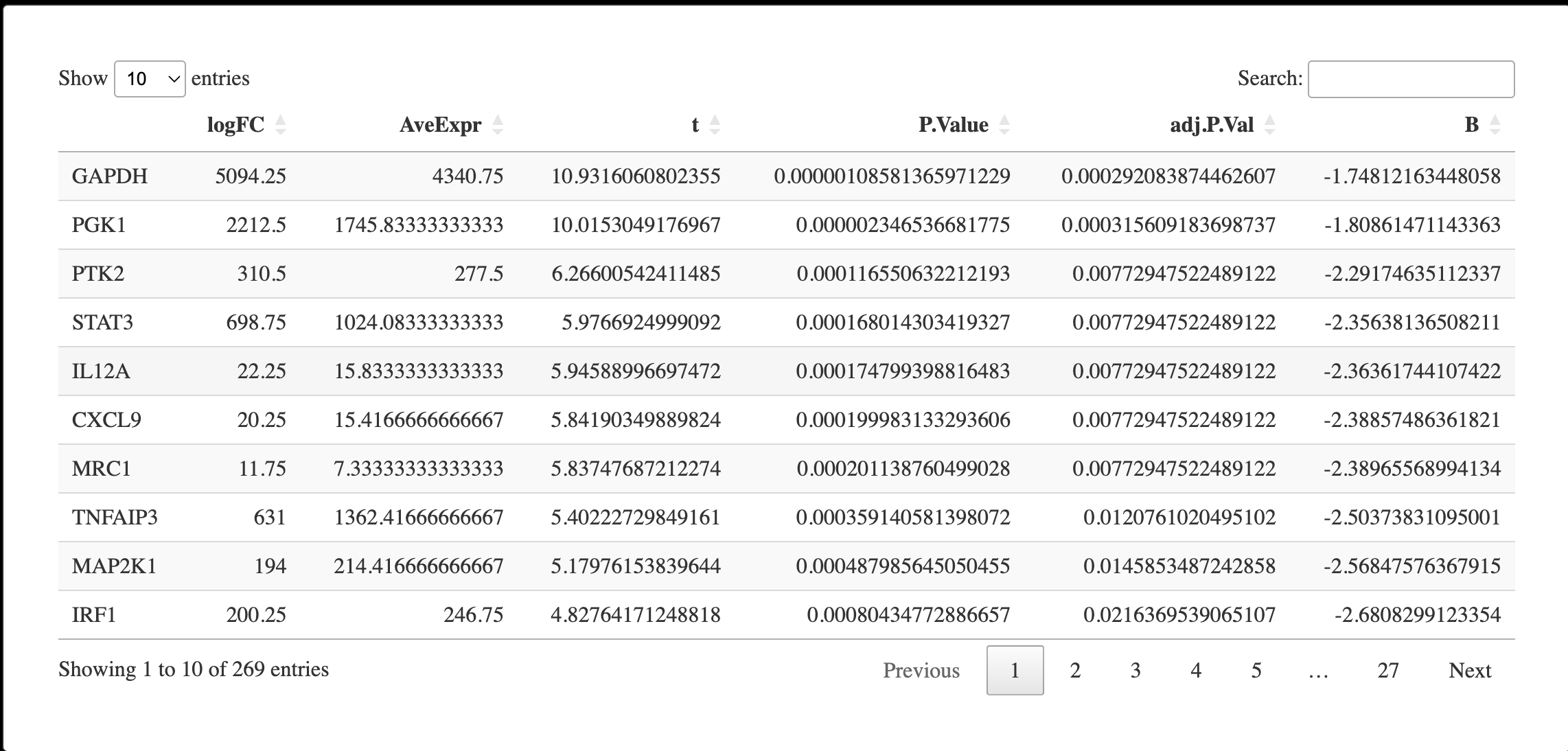
7.5 输出结果
colnames(fit2)

A_VS_B <- topTreat(fit2, coef=1, n=Inf)
A_VS_C <- topTreat(fit2, coef=2, n=Inf)
B_VS_C <- topTreat(fit2, coef=3, n=Inf)
DT::datatable(A_VS_B)

点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰
📍 🤣 chatPDF | 别再自己读文献了!让chatGPT来帮你读吧!~
📍 🤩 WGCNA | 值得你深入学习的生信分析方法!~
📍 🤩 ComplexHeatmap | 颜狗写的高颜值热图代码!
📍 🤥 ComplexHeatmap | 你的热图注释还挤在一起看不清吗!?
📍 🤨 Google | 谷歌翻译崩了我们怎么办!?(附完美解决方案)
📍 🤩 scRNA-seq | 吐血整理的单细胞入门教程
📍 🤣 NetworkD3 | 让我们一起画个动态的桑基图吧~
📍 🤩 RColorBrewer | 再多的配色也能轻松搞定!~
📍 🧐 rms | 批量完成你的线性回归
📍 🤩 CMplot | 完美复刻Nature上的曼哈顿图
📍 🤠 Network | 高颜值动态网络可视化工具
📍 🤗 boxjitter | 完美复刻Nature上的高颜值统计图
📍 🤫 linkET | 完美解决ggcor安装失败方案(附教程)
📍 ......
本文由 mdnice 多平台发布





















 2912
2912











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








