目录
医学影像学
医学影像是指为了医疗或医学研究,对人体或人体某部份,以非侵入方式取得内部组织影像的技术与处理过程。
目前的主要处理方式有:X光成像(X-ray),电脑断层扫描(CT),核磁共振成像(MRI), 超声成像(ultrasound),正子扫描(PET),脑电图(EEG),脑磁图(MEG),眼球追踪(eye-tracking),穿颅磁波刺激(TMS)等现代成像技术,用来检查人体无法用非手术手段检查的部位的过程。
医学影像学的崛起使得对于病人的诊断有了更加直观的证据。除此之外,对于医学领域的相关研究,也有着巨大的影响。
dicom文件相关
1.1 什么是dicom图像
DICOM(Digital Imaging and Communications in Medicine)即医学数字成像和通信,是医学图像和相关信息的国际标准。其是目前应用最为广泛的医学影像格式,常见的CT,核磁共振,心血管成像等大多采用的是docim格式的存储。
1.2 dicom图像中有什么
doicm主要存储两方面信息:
(1)关于患者的PHI(protected health imformation)信息
简单来说,就是患者的相关信息(这些信息是收到保护的)。例如,姓名,性别,年龄,继往病历等
(2)直观的图像信息
一部分信息是扫描过后患者图像的某一层(切片,稍后会讲到),医生可以通过专门的dicom阅读器去打开,从而察看患者病情。另一部分是相关的设备信息,例如生产的dicom图像是X光机扫描出的X光图像的某一层,那么dicom就会存储关于此X光机的相关设备信息。
更具体地划分,这些信息可以被分为以下四类:
(1)Patient
例如患者的姓名,性别,体重,ID等等,反映的主要是患者个人的相关信息。
(2)Study
也就是关于患者接受某项检查所记录的信息,例如检查号,检查日期,检查时患者的年龄,检查的部位等等。
(3)Series
存储关于检查的简单信息,例如图像的层厚,层间距,图像方位等等。
(4)Image
存储关于检查结果(例如X光影像的某一层)的详细信息,例如影像拍摄日期,图像的分辨率,像素间距等等。
注意
dicom文件
≠
\neq
= 患者影像
一方面,在实际检查过程中,例如CT检查,是利用X光线等放射性光线与探测器一同围绕人体做多层的断面扫描。也就是说,我们得到的结果是一层一层的断面图像,将这些断面图像在z轴叠加起来,才是一个完整的医学影像。而每一个dicom存储其中一层图像(但又不仅仅是存储图像),换句话说,一位患者有多个dicom文件。

如上图,一个完整的医学影像是由许多个断面图像在z轴上堆叠而成的三维图像。而一个dicom文件只存储其中的一切图像及相关信息。
另一方面,dicom文件不仅仅存储的是患者影响的切片,还存储着相关的信息(在上面已经提到)。
综上,dicom是一个存储患者所拍摄医学影像的某一层断面图和相关信息的文件。
1.3 dicom结构及组成
dicom文件内部的组成结构如下图:
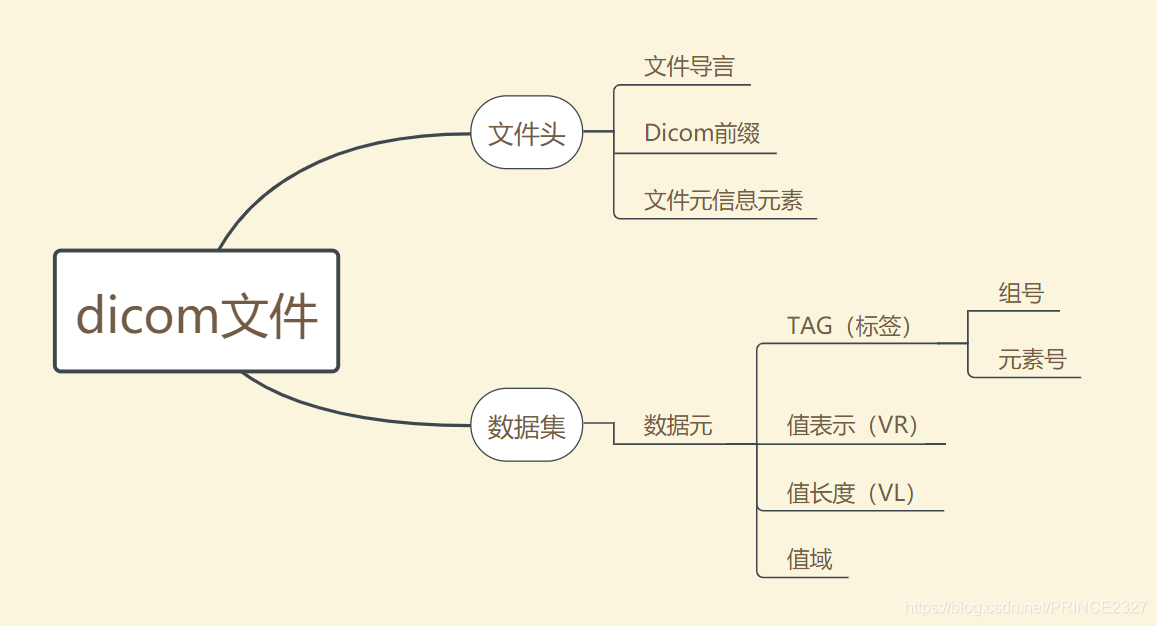
可以看出,一个完整的dicom文件由两部分组成:文件头和数据集。
1.3.1 文件头
Dicom文件头包含的是关于数据集的相关信息,主要有以下几部分:
⋅ · ⋅ 文件导言
⋅ · ⋅ Dicom前缀,也是计算机读取过程中判断一个文件是否是dicom文件的依据。
⋅ · ⋅ 文件信息元素
1.3.2 数据集
数据集即存储相关数据的地方,数据集的基本单位是数据元。数据元由四个部分组成:
(1) Tag
也即标签部分,用于识别数据的具体类型。包括组号(Group)和元素号(Element)两部分。在这里,Group对应的编码是指所存储信息的大类,也就是指明信息是patient,study,series,image四大类中哪一个大类。在确定了Group以后iu,再去检索Element从而确定具体的信息。例如(0008,0050)指的就是病患的检查号信息。
更具体的Tag表见附录。
(2) VR(值表示)
存储Tag指向信息的数据类型。
(3)VL(数据长度)
保存Tag指向信息的数据长度。
(4)值域
保存Tag指向信息的具体值。
一般来说,在处理时,dicom显得有些麻烦。所以会把dicom文件进行转换,从而处理转换后的文件。这里采用的转换方法是将dicom文件转换为raw文件和mhd文件。
raw文件相关
Raw文件,意为“未处理的文件”,其保存的是纯像素信息。常常是一个病人的所有dicom文件中的图像提取出来放在一个raw文件里。也就是说,一个病人对应一个raw文件,其中存储的是该病人的图像信息。(可以理解为将该病人不同的dicom切片图像都叠到一起,形成了一个三维图像)。
mhd文件相关
上面已经提到,dicom文件除了包含切片图像外,还包含其他的一些信息。那么在文件格式转换后,图像信息被raw文件提取,非图像信息则存储在mhd头文件中。简单来说,mhd头文件是存储关于一个病人的所有dicom文件中的非图像信息。
由上述关系可以知道:raw文件与mhd文件是一一对应的,且它们的数量小于等于(实际中一定是小于)dicom文件数量。
相应代码
2.1 dicom可视化
import matplotlib.patches as patches
import matplotlib.pyplot as plt
import pydicom
from pydicom.data import get_testdata_files
import os
import xlrd
import xlwt
print(__doc__)
path = r'C:\Users\86152.000\Desktop\python\M'
files = os.listdir(path)
for filename in files:
dataset = pydicom.dcmread(filename)
# Normal mode:
print()
print("Filename.........:", filename)
print("Storage type.....:", dataset.SOPClassUID)
print()
pat_name = dataset.PatientName
display_name = pat_name.family_name + ", " + pat_name.given_name
print("Patient's name...:", display_name)
print("Patient id.......:", dataset.PatientID)
print("Modality.........:", dataset.Modality)
print("Study Date.......:", dataset.StudyDate)
if 'PixelData' in dataset:
rows = int(dataset.Rows)
cols = int(dataset.Columns)
print("Image size.......: {rows:d} x {cols:d}, {size:d} bytes".format(
rows=rows, cols=cols, size=len(dataset.PixelData)))
if 'PixelSpacing' in dataset:
print("Pixel spacing....:", dataset.PixelSpacing)
# use .get() if not sure the item exists, and want a default value if missing
print("Slice location...:", dataset.get('SliceLocation', "(missing)"))
def readexcel():
workbook=xlrd.open_workbook(r'C:\Users\86152.000\Desktop\python\annotated_coordinates.xlsx')
print(workbook.sheet_names())
sheet1=workbook.sheet_by_name('Sheet1')
nrows=sheet1.nrows
ncols=sheet1.ncols
print(nrows,ncols)
r=2
c=4
while True:
cellA=sheet1.cell(r,c).value
cellB=sheet1.cell(r,c).value
print(cellA,cellB)
rect=patches.Rectangle((20,30),10,20,linewidth=1,edgecolor='red',facecolor='none')
ax=plt.subplot(1,1,1)
ax.add_patch(rect)
plt.imshow(dataset.pixel_array, cmap=plt.cm.bone)
plt.show()
r=r+1
if r==nrows:
c=c+1
r=2
if c==ncols-2:
print('完成')
break
readexcel()
输出如下:


这里的红框可以去掉,在代码里注释即可。(本想标注dicom数据的,结果导师说应该是raw数据)
2.2 dicom读取并转换为raw及mhd文件
import cv2
import os
import pydicom
import numpy
import SimpleITK
# 路径和列表声明
# 与python文件同一个目录下的文件夹,存储dicom文件,该文件路径最好不要含有中文
PathDicom = r"/home/Wangling/TuoBaoqiang/python/Test1"
# 与python文件同一个目录下的文件夹,用来存储mhd文件和raw文件,该文件路径最好不要含有中文
SaveRawDicom = r"/home/Wangling/TuoBaoqiang/python/Data_Img/test1"
lstFilesDCM= []
lstFilesDCM_0 = []
temp = []
listdicom = []
# dicom文件夹下的第一层目录 ——> group一维列表
root_1 = os.listdir(PathDicom)
group = []
for i in range(0,len(root_1)):
group.append(PathDicom + '/' + root_1[i]) #group列表
# dicom文件夹下的第二层目录 ———> 000x一维列表
root_2 = []
for j in range(0,len(group)):
root_2.append(os.listdir(group[j])) # group下dicom文件夹,root_2为二维列表,例[['0001', '0002'], ['0003', '0104']]
nameformhd = sum(root_2,[]) # 将root_2降为一维列表,以备程序末尾mhd文件的命名
# 每个dicom文件的完整目录 ———> /group/000x
for m in range(0,len(group)):
for n in range(0,len(root_2[0])): # 假设每个group中dicom文件夹的数量是相等的,即10,如果有改变,要修改!
eachdicom = group[m] + '/' + root_2[m][n]
lstFilesDCM_0.append(eachdicom)
# print('The dicom filelist -> %s'%lstFilesDCM_0) # 打印dicom文件列表
# 将完整列表lstFilesDCM_0下的dicom文件地址读取到listdicom中
for k in range(0,len(lstFilesDCM_0)):
for dirName, subdirList, fileList in os.walk(lstFilesDCM_0[k]):
for filename in fileList:
if "img" in filename.lower(): # 判断文件是否为dicom文件
temp.append(os.path.join(dirName, filename))
listdicom.append(temp) # 加入到列表中
temp = []
# print('listdicom=%s'%listdicom) #打印dicom全部文件载入的列表
# print('The number of listdicom is %d'%len(listdicom)) #打印预备处理dicom文件的个数
for filenames in listdicom:
# print('the current processing list is %s'%filenames) #打印当前正在处理的dicom列表
lstFilesDCM = filenames
# 第一步:将最后一张图片作为参考图片,并认为所有图片具有相同维度
RefDs = pydicom.read_file(lstFilesDCM[-1],force = True) # 读取最后一张dicom图片
RefDs_0 = pydicom.read_file(lstFilesDCM[0],force = True) # 读取第一张dicom图片,第三步备用
# 第二步:得到dicom图片所组成3D图片的维度
ConstPixelDims = (int(RefDs.Rows), int(RefDs.Columns), len(lstFilesDCM)) # ConstPixelDims是一个元组
# 第三步:得到x方向和y方向的Spacing并得到z方向的层厚
RefDs.SliceThickness = abs(RefDs_0.ImagePositionPatient[2]-RefDs.ImagePositionPatient[2])/(len(lstFilesDCM)-1)
# 打印slicethickness的值,判断数据是否正确
print(float(RefDs_0.SliceThickness),RefDs.SliceThickness,RefDs_0.ImagePositionPatient[2],RefDs.ImagePositionPatient[2],len(lstFilesDCM))
print(RefDs_0.ImagePositionPatient)
print(RefDs.ImagePositionPatient)
ConstPixelSpacing = (float(RefDs.PixelSpacing[0]), float(RefDs.PixelSpacing[1]), float(RefDs.SliceThickness))
# 第四步:得到图像的原点
Origin = RefDs.ImagePositionPatient
# 根据维度创建一个numpy的三维数组,并将元素类型设为:pixel_array.dtype
ArrayDicom = numpy.zeros(ConstPixelDims, dtype=RefDs.pixel_array.dtype) # array is a numpy array
# 第五步:遍历所有的dicom文件,读取图像数据,存放在numpy数组中
i = 0
for filenameDCM in lstFilesDCM:
ds = pydicom.read_file(filenameDCM)
ArrayDicom[:, :, lstFilesDCM.index(filenameDCM)] = ds.pixel_array
cv2.imwrite("out_" + str(i) + ".png", ArrayDicom[:, :, lstFilesDCM.index(filenameDCM)])
i += 1
# 第六步:对numpy数组进行转置,即把坐标轴(x,y,z)变换为(z,y,x),这样是dicom存储文件的格式,即第一个维度为z轴便于图片堆叠
ArrayDicom = numpy.transpose(ArrayDicom, (2, 0, 1))
# 第七步:将现在的numpy数组通过SimpleITK转化为mhd和raw文件
sitk_img = SimpleITK.GetImageFromArray(ArrayDicom, isVector=False)
sitk_img.SetSpacing(ConstPixelSpacing)
sitk_img.SetOrigin(Origin)
SimpleITK.WriteImage(sitk_img,os.path.join(SaveRawDicom,nameformhd[listdicom.index(filenames)]+ ".mhd"))
输出如下:

2.3 raw文件可视化
import os
import SimpleITK as sitk
import matplotlib.pyplot as plt
if __name__=="__main__":
#识别中文路径名
path=r"C:\Users\86152.000\Desktop\python\SaveRaw\0002.mhd"
pwd = os.getcwd()
os.chdir(os.path.dirname(path))
#读取切片图像
image = sitk.ReadImage(os.path.basename(path))
image = sitk.GetArrayFromImage(image)
#显示图像
for i in range(1,image.shape[0],1):
plt.figure()
plt.imshow(image[i,:,:], cmap='gray')
plt.pause(1)
plt.close()
print(i)
输出如下:
2.4 遍历指定文件
import os
import SimpleITK as sitk
import matplotlib.pyplot as plt
def show_files(path, all_files):
# 首先遍历当前目录所有文件及文件夹
file_list = os.listdir(path)
# 准备循环判断每个元素是否是文件夹还是文件,是文件的话,把名称传入list,是文件夹的话,递归
for file in file_list:
# 利用os.path.join()方法取得路径全名,并存入cur_path变量,否则每次只能遍历一层目录
cur_path = os.path.join(path, file)
# 判断是否是文件夹
if os.path.isdir(cur_path):
show_files(cur_path, all_files)
else:
# 判断是否满足后缀
if suffix in cur_path:
all_files.append(cur_path)
return all_files
# 对指定的文件进行的操作
import os
def show_files(path, all_files):
# 首先遍历当前目录所有文件及文件夹
file_list = os.listdir(path)
# 准备循环判断每个元素是否是文件夹还是文件,是文件的话,把名称传入list,是文件夹的话,递归
for file in file_list:
# 利用os.path.join()方法取得路径全名,并存入cur_path变量,否则每次只能遍历一层目录
cur_path = os.path.join(path, file)
# 判断是否是文件夹
if os.path.isdir(cur_path):
show_files(cur_path, all_files)
else:
# 判断是否满足后缀
if suffix in cur_path:
all_files.append(cur_path)
return all_files
# 对指定的文件进行的操作
def handle_file(file_path):
open(file_path)
print(file_path)
def find_file():
# 传入空的list接收文件名
all_files = show_files(read_dir_path, [])
# 循环打印show_files函数返回的文件名列表
for file in all_files:
handle_file(file)
print(file)
if __name__ == "__main__":
# 要读取的文件夹
read_dir_path = r'C:\Users\86152.000\Desktop'
# 要匹配的文件类型
suffix = '.mhd'
find_file()
输出如下:

附录(常见Tag说明)
Patient Tag
| Group(组号) | Element(元素号) | Tag Description | 中文解释 | VR |
|---|---|---|---|---|
| 0010 | 0010 | Patient’s Name | 患者姓名 | PN |
| 0010 | 0020 | Patient ID | 患者ID | LO |
| 0010 | 0030 | Patient’s Birth Date | 患者出生日期 | DA |
| 0010 | 0032 | Patient’s Birth Time | 患者出生时间 | TM |
| 0010 | 0040 | Patient’s Sex | 患者性别 | CS |
| 0010 | 1030 | Patient’s Weight | 患者体重 | DS |
| 0010 | 21C0 | Pregnancy Status | 怀孕状态 | US |
Study Tag
| Group | Elemen | Tag Description | 中文解释 | VR |
|---|---|---|---|---|
| 0008 | 0050 | Accession Number:A RIS generated number that identifies the order for the Study. | 检查号:RIS的生成序号,用以标识做检查的次序. | SH |
| 0020 | 0010 | Study ID | 检查ID. | SH |
| 0020 | 000D | Study Instance UID: Unique identifier for the Study. | 检查实例号:唯一标记不同检查的号码. | UI |
| 0008 | 0020 | Study Date: Date the Study started. | 检查日期:检查开始的日期. | DA |
| 0008 | 0030 | Study Time:Time the Study started. | 检查时间:检查开始的时间. | TM |
| 0008 | 0061 | Modalities in Study | 一个检查中含有的不同检查类型. | CS |
| 0008 | 0015 | Body Part Examined | 检查的部位. | CS |
| 0008 | 1030 | Study Description | 检查的描述 | . LO |
| 0010 | 1010 | Patient’s Age | 做检查时刻的患者年龄,而不是此刻患者的真实年龄. | AS |
Series Tag
| Group | Element | Tag Description | 中文解释 | VR |
|---|---|---|---|---|
| 0020 | 0011 | Series Number:A number that identifies this Series. | 序列号:识别不同检查的号. | IS |
| 0020 | 000E | Series Instance UID:Unique identifier for the Series. | 序列实例号:唯一标记不同序列的号码. | UI |
| 0008 | 0060 | Modality 检查模态(MRI/CT/CR/DR) | CS | |
| 0008 | 103E | Series Description | 检查描述和说明 | LO |
| 0008 | 0021 | Series Date | 检查日期 | DA |
| 0008 | 0031 | Series Time | 检查时间 | TM |
| 0020 | 0032 | Image Position (Patient):The x, y and z coordinates of the upper left hand corner of the image, in mm. | 图像位置:图像的左上角在空间坐标系中的x,y,z坐标,单位是毫米.如果在检查中,则指该序列中第一张影像左上角的坐标. | DS |
| 0020 | 0037 | Image Orientation (Patient):The direction cosines of the first row and the first column with respect to the patient. | 图像方位 | DS |
| 0018 | 0050 | Slice Thickness:Nominal slice thickness, in mm. | 层厚. | DS |
| 0018 | 0088 | Spacing Between Slices | 层与层之间的间距,单位为mm | DS |
| 0020 | 1041 | Slice Location:Relative position of exposure expressed in mm. | 实际的相对位置,单位为mm. | DS |
| 008 | 0023 | MR Acquisition | 收购者 | CS |
| 0018 | 0015 | Body Part Examined | 身体部位 | . CS |
Image Tag
| Group | Element | Tag Description | 中文解释 | VR |
|---|---|---|---|---|
| 0008 | 0008 | Image Type:Image identification characteristics. | 图像类型 | CS |
| 0008 | 0018 | SOP Instance UID | SOP实例 | UID. |
| 0008 | 0023 | Content Date:The date the image pixel data creation started. | 影像拍摄的日期. | DA |
| 0008 | 0033 | Content Time | 影像拍摄的时间. | TM |
| 0020 | 0013 | Image/Instance Number:A number that identifies this image. | 图像码:辨识图像的号码. | IS |
| 0028 | 0002 | Samples Per Pixel:Number of samples (planes) in this image. | 图像上的采样率. | US |
| 0028 | 0004 | Photometric Interpretation:Specifies the intended interpretation of the pixel data. | 光度计的解释,对于CT图像,用两个枚举值MONOCHROME1,MONOCHROME2.用来判断图像是否是彩色的,MONOCHROME1/2是灰度图, RGB则是真彩色图,还有其他. | CS |
| 0028 | 0010 | Rows: Number of rows in the image. | 图像的总行数,行分辨率. | US |
| 0028 | 0011 | Columns: Number of columns in the image. | 图像的总列数,列分辨率. | US |
| 0028 | 0030 | Pixel Spacing:Physical distance in the patient between the center of each pixel. | 像素间距.像素中心之间的物理间距. | DS |
| 0028 | 0100 | Bits Allocated:Number of bits allocated for each pixel sample. Each sample shall have the same number of bits allocated. | 分配的位数:存储每一个像素值时分配的位数,每一个样本应该拥有相同的这个值. | US |
| 0028 | 0101 | Bits Stored:Number of bits stored for each pixel sample. Each sample shall have the same number of bits stored | 存储的位数:有12到16列举值.存储每一个像素用的位数.每一个样本应该有相同值. | US |























 1939
1939











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








