一、题目要求
对Swiss-Model和Modeller建模结果进行VMD可视化比较
二、操作过程记录及结果
RMSD比对
模型间比对
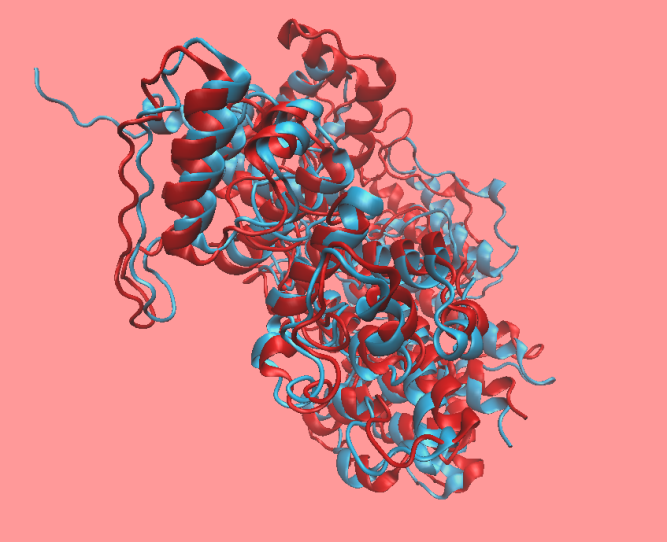
选取多模板建模出来的两个结果,使用VMD的RMSD Tool,对1-630全部氨基酸,计算出RMS deviations:8.02,VMD可视化看得出来两个结果如同重影一般,非常相近。
图表 1 1-630RMSD比对
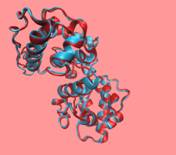
接下来,依次对12-87的EF-hand、109-382以及383-627的两个Actin-binding区域,计算RMSdeviations,下图从左到右依次是EF-hand、Actin-binding 1和Actin-binding 2。
| 氨基酸位置 | 12-87 | 109-382 | 383-627 |
| RMS deviations | 20.10 | 1.32 | 1.95 |
可以看出来,EF-hand的建模间的差异较大。
模型模板间比较
将使用1rt8.1模板,用Swiss-model建立的模型,红色是1rt8模板,黄色是模型,可以看出来两个几乎一模一样,由此可以知道Swiss-model同源建模,对于有已知结构的部分,是直接使用模板的。
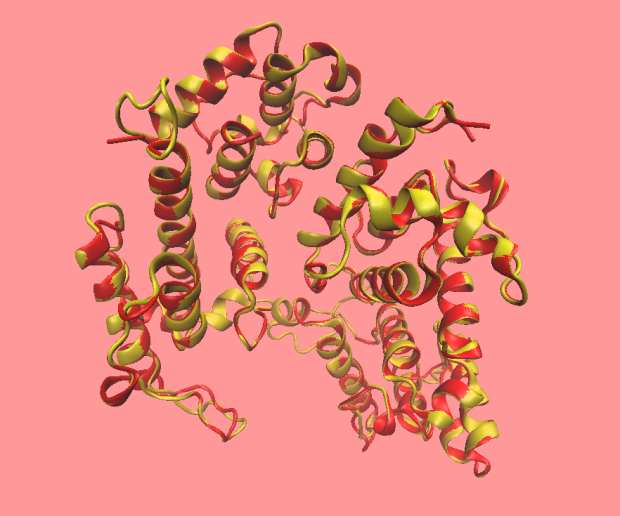
图表 2 Swiss-model模板模型比较
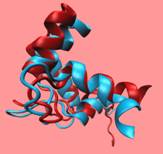
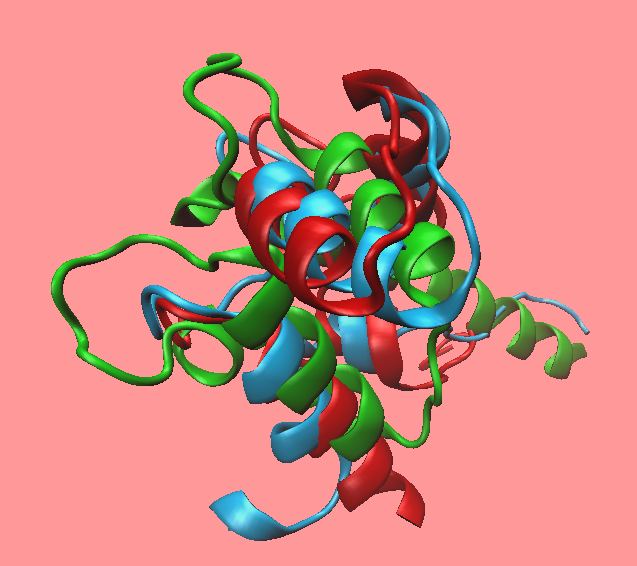
由于EF-hand区域不同模型间差异较大,下面对2f2o模板(template)与模型(model)进行比对,绿色为2f2o模板。下表为三个结构两两计算RMSD的值。
| RMS deviations | Model1 | Model2 | 2f2o |
| Model1 | 0 | 4.54 | 8.86 |
| Model2 | 4.54 | 0 | 11.68 |
| 2f2o | 8.86 | 11.68 | 0 |
可以得出结论, EF-hand区域建模效果很不好,模型与模板间并没有完全匹配到一起去。究其原因,可能是之前modeller建模时,直接补上EF-hand那段氨基酸。
图表 3EF-hand区域
分析与讨论
Loop区域由于本身结构可变化,所以建模时候,会有差异,并且之前EF-hand部分之前补上,那段区域效果也不是很好。
























 2621
2621

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








