上周微信小伙伴问了一个图,想要用ggplot做拟时热图,其实我在看文献的时候,已经想要复现了,所以这里写一篇帖子,将一众问题一网打尽。**这是一篇超长贴,我们的内容还是一如既往的不仅实现了修饰,更多的是让你学到新的内容:
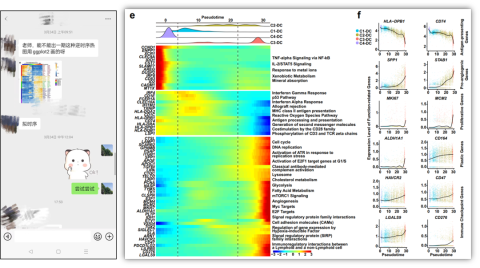
(reference:Single-cell sequencing of ascites fluid illustrates heterogeneity and therapy-induced evolution during gastric cancer peritoneal metastasis | Nature Communications)
在这个帖子中,我们主要完成了四件事: 一、之前就有小伙伴问,拟时热图的颜色怎么修改,它的实质是一个热图,当然是按照热图的颜色修改呀,很简单:
setwd('D:/KS项目/公众号文章/ggplot修饰monocle热图')
library(monocle)
library(monocle)
library(tidyverse)
library(ggridges)
library(RColorBrewer)
library(scales)
library(dplyr)
library(tidytree)
library(viridis)
library(scales)
load("D:/KS项目/公众号文章/ggplot修饰monocle热图/cds.RData")#拟时cds
#查看随着pseudotime变化的基因
# cds_DGT_pseudotimegenes <- differentialGeneTest(mouse_monocle,fullModelFormulaStr = "~sm.ns(Pseudotime)")
#这里完全是为了展示较少的基因所以控制了阈值,实际需要展示什么基因,自己选择
cds_DGT_pseudotimegenes_sig <- subset(ds_DGT_pseudotimegenes, qval < 0.01)
mouse_data <- readRDS("D:/KS项目/公众号文章/mouse_data.rds")
# marker <- FindAllMarkers(mouse_data, only.pos = T,logfc.threshold = 0.5)
# marker <- marker[which(marker$p_val_adj<0.05),]
top15 <- marker %>% group_by(cluster) %>% top_n(n = 15, wt = avg_log2FC)
top15_ordergene <- cds_DGT_pseudotimegenes_sig[top15$gene, ]
Time_genes <- top15_ordergene %>% pull(gene_short_name) %>% as.character()
Time_genes <- unique(Time_genes)
p <- plot_pseudotime_heatmap(mouse_monocle[Time_genes,],
num_cluster = 4,
show_rownames = T,
return_heatmap = T,
hmcols = colorRampPalette(c("navy","white","firebrick3"))(100))
p <- plot_pseudotime_heatmap(mouse_monocle[Time_genes,],
num_cluster = 4,
show_rownames = T,
return_heatmap = T,
hmcols =colorRampPalette(rev(brewer.pal(9, "PRGn")))(100))
p <- plot_pseudotime_heatmap(mouse_monocle[Time_genes,],
num_cluster = 4,
show_rownames = T,
return_heatmap = F,
hmcols = viridis(256))
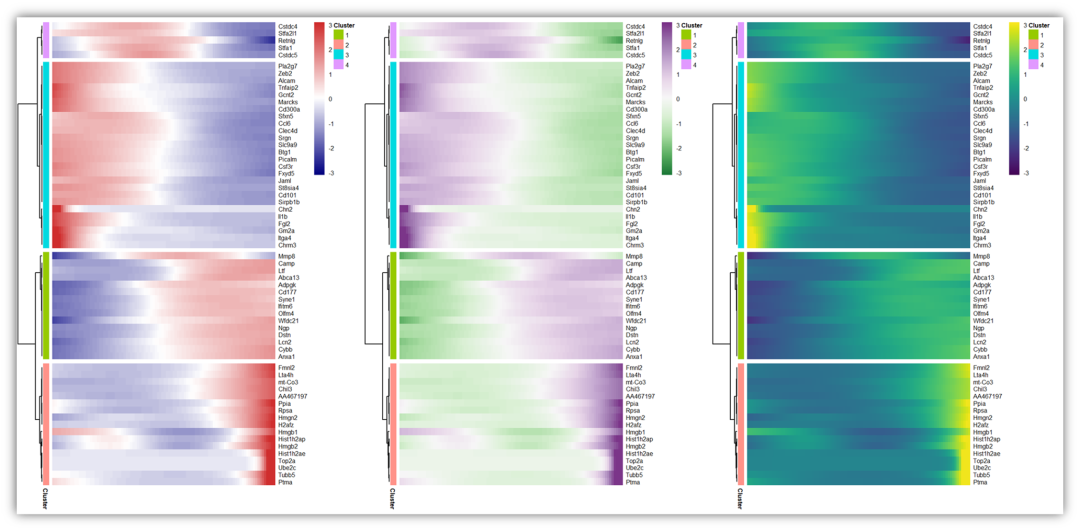
二、提取热图数据,用pheatmap修饰。
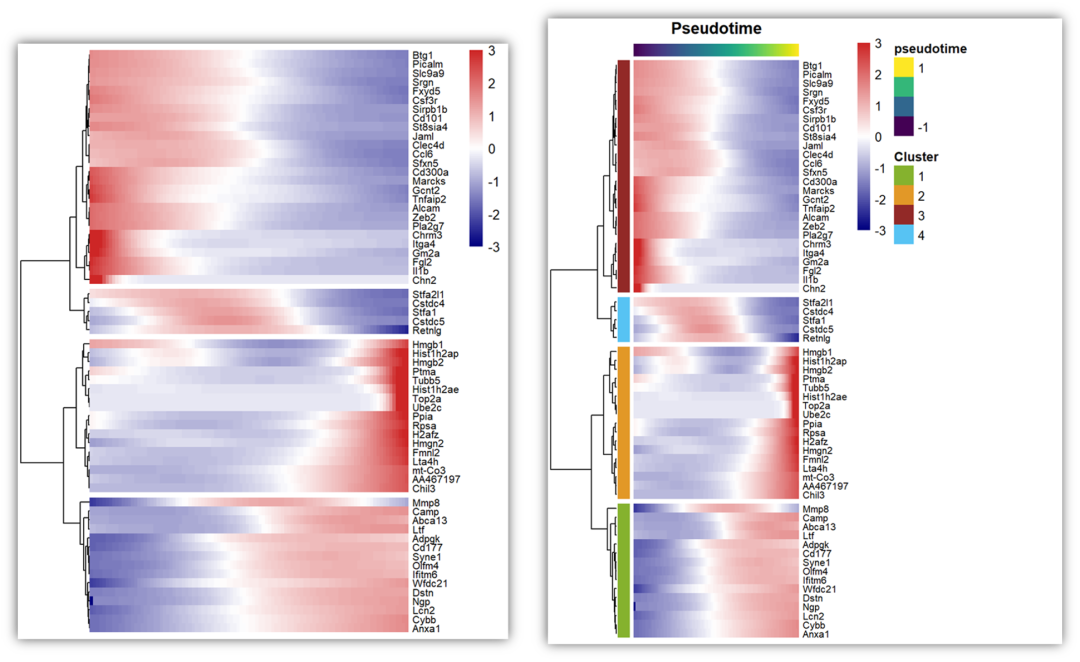
三、ggplot做拟时热图并进行修饰,结合一个山脊图,可以看出细胞在拟时的分布情况。

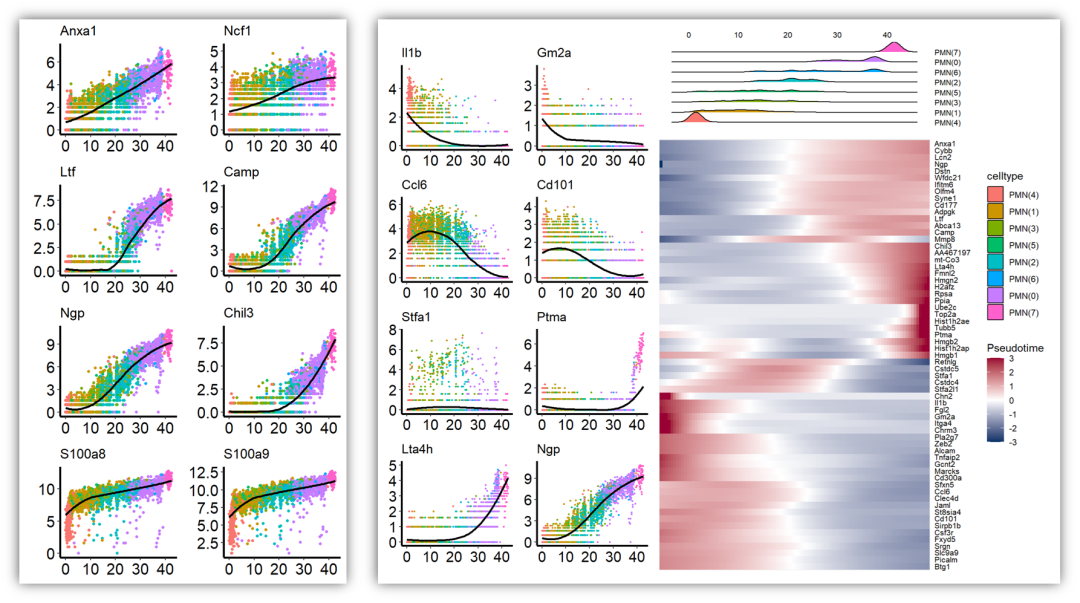
四、复现一下拟时基因的散点趋势图,这个数据处理类似于单细胞拟时分析:基因及通路随拟时表达变化趋势。这里和热图组合在一起,还是很好的。
至于文献中的富集分析,其实也很简单,在中间分析过程中,我们可以提取每个聚类cluster的基因去做富集分析,然后添加在图上。这图的修饰不得是CNS级别的。有需要的学习起来吧。
这里需要提一句,对于拼图不必请求R,如果觉得默认的拼图效果不是特别好的时候,AI可以更好地修饰。更多精彩内容请至我的公众号KS科研分享与服务





















 1978
1978

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








