一、前置环境
1.1 R 语言
下载对应系统的R软件
R: The R Project for Statistical Computing (r-project.org)
以win11为演示
https://mirrors.tuna.tsinghua.edu.cn/CRAN/bin/windows/
1.2 R Studio
安装R Studio
https://download1.rstudio.org/electron/windows/RStudio-2022.12.0-353.exe
安装后启动并关联已安装的R语言
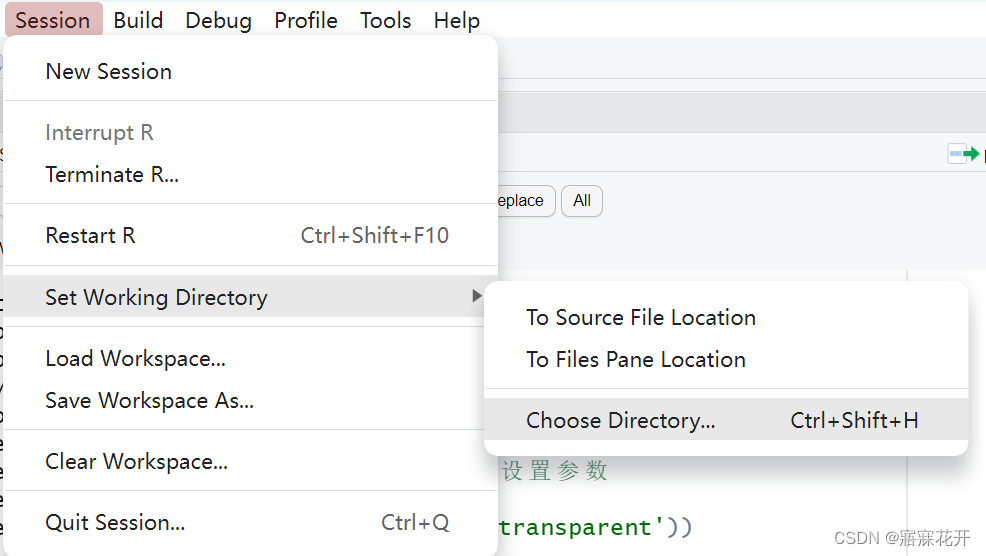
设置工作目录Session ----> Set Working Directory ---> Choose Directory
注意最好是全英文路径,避免奇奇怪怪的Bug
1.3 安装ggplot2 R包
##安装包
install.packages("ggplot2")
##加载包
library(ggplot2)
二、DE基因火山图绘制
2.1 准备文件
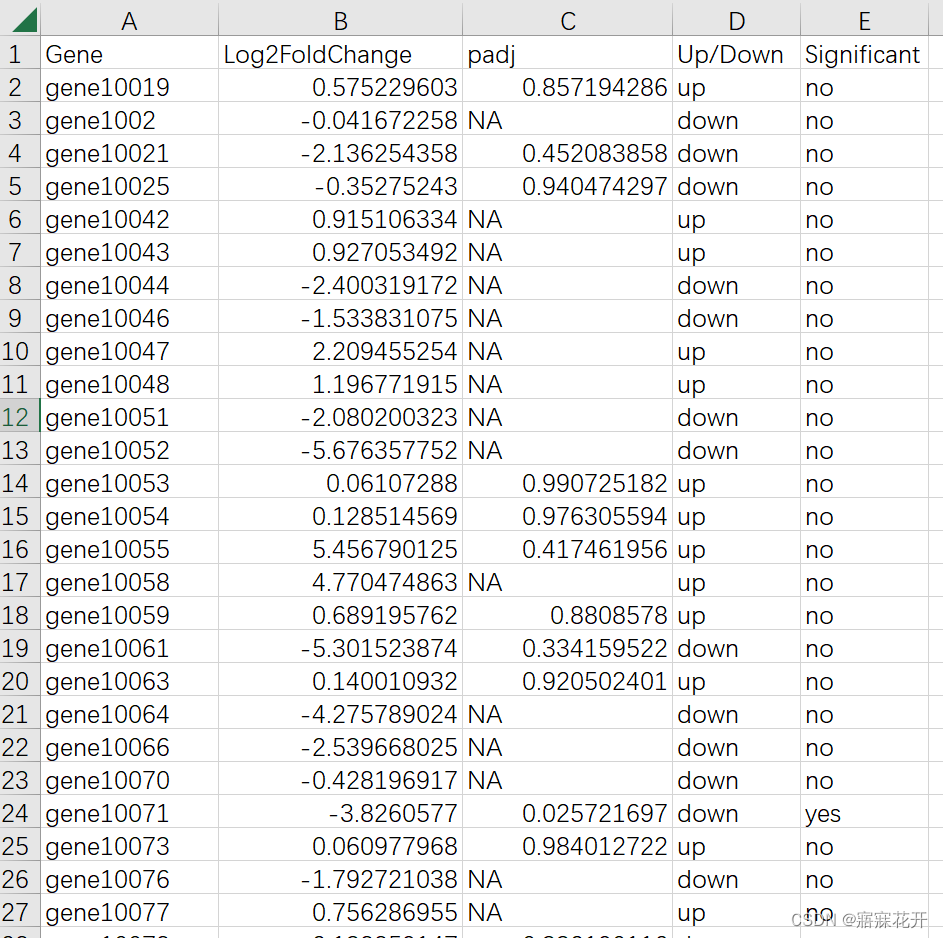
主要需要三列信息:Gene Log2FoldChange padj
可自行命名,其中
Log2FoldChange列的数值为Log2(FoldChange)
padj 是校正P值或者 可以用原始P value
2.2 读取文件
##文件输入
data <- read.table("V_GV_GV.anno.xls_ggplot2",header = T)
##去除NA行
data_na <-na.omit(data)
##或将NA替换为其他值,如1,但出来的图会在底部多出许多异常点,不建议此操作
data[is.na(data)] <- "1"
2.3 标记上下调基因
##设置阈值,其中padj< 0.05; Log2FoldChange) > 1;需相应更改
data_na$changed <- factor(ifelse(data_na$padj < 0.05 & abs(data_na$Log2FoldChange) > 1,ifelse(data_na$Log2FoldChange > 1,'Up','Down'),'Stable'))

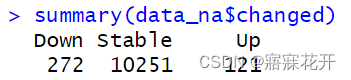
##查看DE基因数
summary(data_na$changed)
2.4 绘图并保存
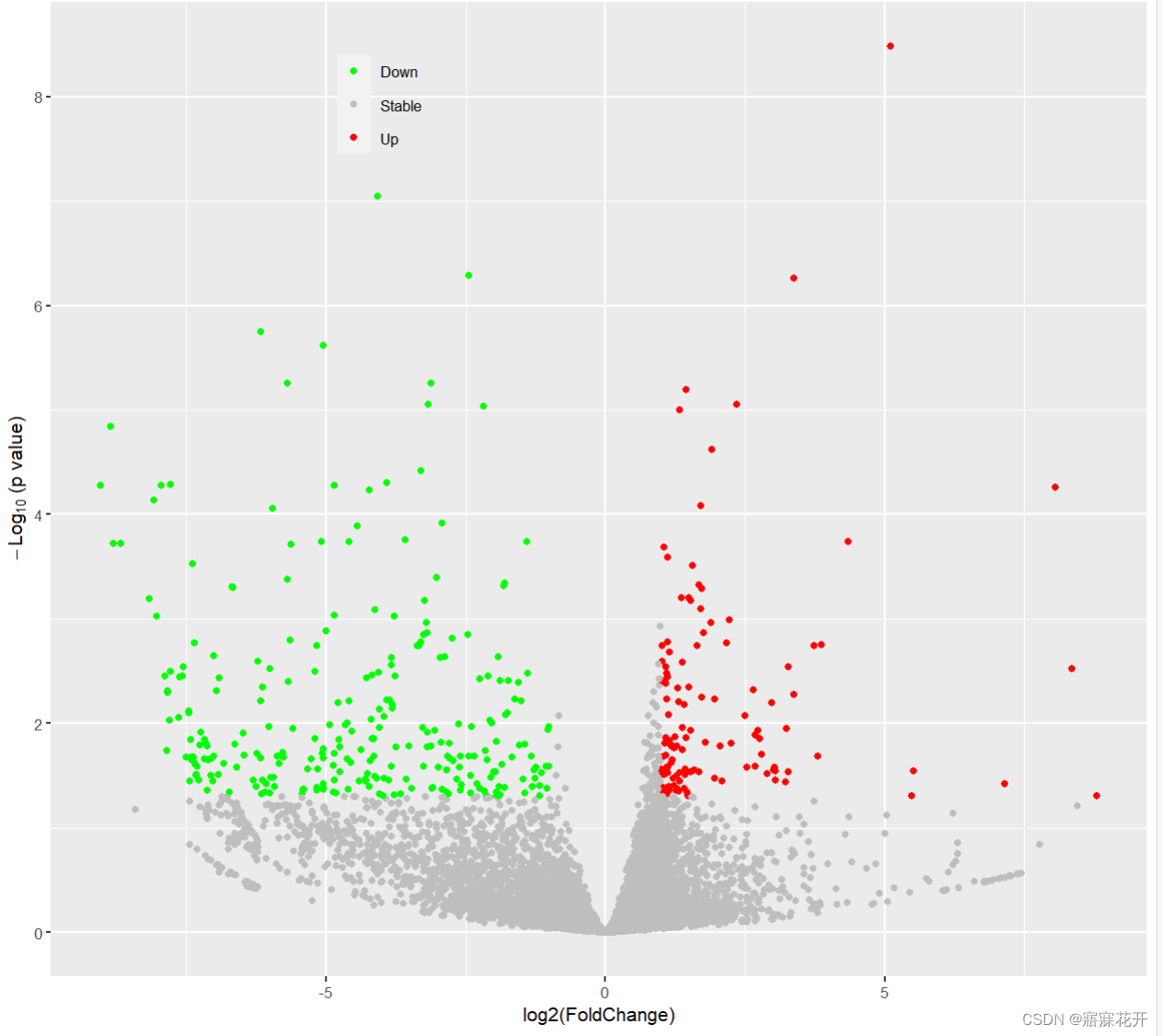
##绘图
ggplot(data_na,aes(Log2FoldChange,-log10(padj),
color=factor(changed),
)) +
geom_point() +
labs(x=expression("log2"*"(FoldChange)"), ##x轴Title
y=expression(-Log[10]*" (p value)")) + ##y轴Title
theme_grey(base_size = 10)+
scale_color_manual(values = c('green','grey','red'))+ ##对应分类点的颜色
scale_size_manual(values = c(1,1,1))+ ##对应分类点的Size
theme(legend.title = element_blank(),
legend.position = c(0.3,0.9),
legend.background = element_rect(fill='transparent'))
##保存
ggsave("V_GV_GV_mRNA.png",width=10,height=10,dpi=300)
参考文章:
R语言 ggplot2火山图 - 知乎 (zhihu.com)
(22条消息) R语言如何删除数据集中包含缺失数据NA的行?_ofoliao的博客-CSDN博客_r语言删除缺失值所在一行
ggplot2之二:图像的保存 - 知乎 (zhihu.com)
声明:
代码源于参考文章,进行相应更改,如有侵权,请联系删除,感谢各位的教程!






















 4426
4426











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








