文章目录
Toward Clinically Assisted Colorectal Polyp Recognition via Structured Cross-Modal Representation Consistency
摘要
背景
结直肠息肉的分类是一项重要的临床检查。为了提高分类精度,大多数计算机辅助诊断算法都采用窄带成像(NBI)来识别结肠息肉。然而,在实际临床场景中,NBI通常缺乏利用,因为当通过使用白光(WL)图像检测到息肉时,该特定图像的采集需要手动切换光模式。为了避免上述情况,提出了一种新的方法,通过进行结构化的跨模态表示一致性,直接实现白光结肠镜图像的精确分类
本文方法
在实践中,一对多模态图像,即NBI和WL被送到共享Transformer中以提取分层特征表示。然后,采用一种新设计的空间注意力模块(SAM)来计算特定模态图像的类标记和patch标记之间的相似性。
通过在不同级别对齐成对的NBI和WL图像的类标记和空间注意力图,Transformer实现了保持上述两种模态的全局和局部表示一致性的能力
代码地址
本文方法

我们提出的通过单个Transformer架构的跨模态共享结肠息肉识别框架以及提出的CGA和SAM的概述。注意,在推理阶段,所有由虚线连接的部分都可以删除
Shared Transformer Block
将双模态分别送入到transformer主干中


Cross-Modal Global Alignment
计算两个配对图像的两个模态特定类标记(class token)之间的余弦差。然后,对它们的余弦相似性施加损失函数会减少两种模态的图像对之间的平均余弦距离。这样,来自配对图像的特征表示可以更紧密地匹配,并且模型可以学习从NBI图像捕获硬特征(例如,在NBI图像中清晰但在WL图像中不清楚的纹理)。

Spatial Attention Module
尽管模态特定的类标记是全局对齐的,但我们也提出了空间注意力模块(SAM)来追求两种模态之间的多级结构化语义一致性。首先,我们通过SAM获得全局引导的亲和力,即每个图像的全局表示和局部区域之间的响应图。随后,我们通过限制两种模态的响应图之间的距离来调整两种模态之间的局部语义。
输出patchtoken特征{Fw,Fn}∈RN×d以及模态特定类标记{cw,cn}∈Rd作为输入,利用{Fw、Fn}通过线性投影来生成空间注意力(SpA)操作的key,而SpA的query是基于{cw,cn}获得的,描述如下


总损失为下面:

实验结果
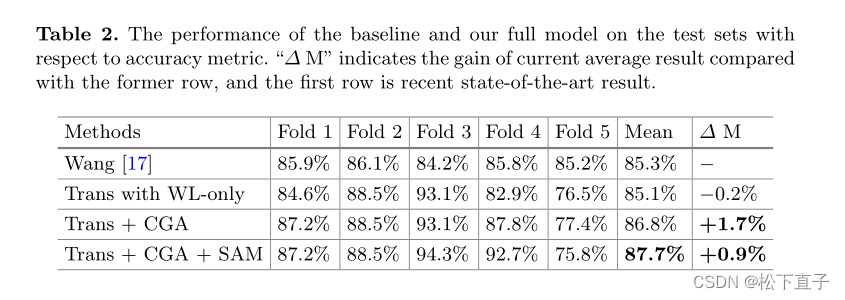









 本文提出了一种新的计算机辅助诊断方法,通过共享Transformer块和跨模态全局对齐,结合空间注意力模块,实现白光结肠镜图像的精确分类,无需依赖窄带成像。这种方法能促进不同模态图像间表示的一致性,提高息肉识别的准确性。
本文提出了一种新的计算机辅助诊断方法,通过共享Transformer块和跨模态全局对齐,结合空间注意力模块,实现白光结肠镜图像的精确分类,无需依赖窄带成像。这种方法能促进不同模态图像间表示的一致性,提高息肉识别的准确性。

















 2935
2935

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










