一、概念
BUSCO 是从单拷贝基因的角度去对基因组的完整性做评估的。
BUSCO 收集了一个叫做 OrthoDB 的数据库,这个数据库收录了很多物种已经发表的蛋白序列,对这些蛋白序列进行从头的计算聚类,之后生成一个一个的cluster,称为基因家族或者是orthologous groups。每个cluster或者是orthologous groups里面认为他们全部是同源基因,从中去挑选单拷贝直系同源基因来去形成BUSCO数据库。

https://busco.ezlab.org/
挑选的过程,首先是基于整个 OrthoDB 数据库里所有的数据在各个分类层级去挑选,在90%物种里面呈现单拷贝的这种group,就当做orthologous groups。它会在不同的分类水平去做单拷贝直系同源基因数据库的构建。比如说真菌层面,植物,动物。再下面又会有哺乳动物的层面,植物又会分单子叶植物的,双子叶植物等等层面的一些数据集。
BUSCO去做基因组评估的时候,可以做基因组水平的评估,也可以做转录本水平的评估。
基因组层面评估:先去基因组上预测这些基因,再去做评估,如果说给他是一个注释好的基因集,这个时候提供的是一个蛋白集合,它就直接是去进行同源搜索,再去评估,看BUSCO数据库里面的这些单拷贝基因有或者无;有的话,是完整的还是不完整的;完整的话,是单拷贝的还是多拷贝的。
转录本层面评估:如果说有一套完整的转录本的集合也可以去评估。首先,tblastn也是同源搜索,同源搜索之后,去对转录本进行读码框预测,预测完的结果还是去进行hmmsearch的搜索(hmmsearch是做蛋白质结构域搜索的),最终拿到BUSCO评估结果。
二、BUSCO软件安装
查看软件帮助

三、数据库下载
BUSCO 评估是基于数据库里面已经生成好的单拷贝直系同源基因来做评估,所以这里如果想要本地加载数据库的话,可以先下载数据库
使用 busco --list-datasets 命令查看BUSCO里面已有的数据库,需要联网才可以查看,如果服务器不可以联网的话,是查看不了的。
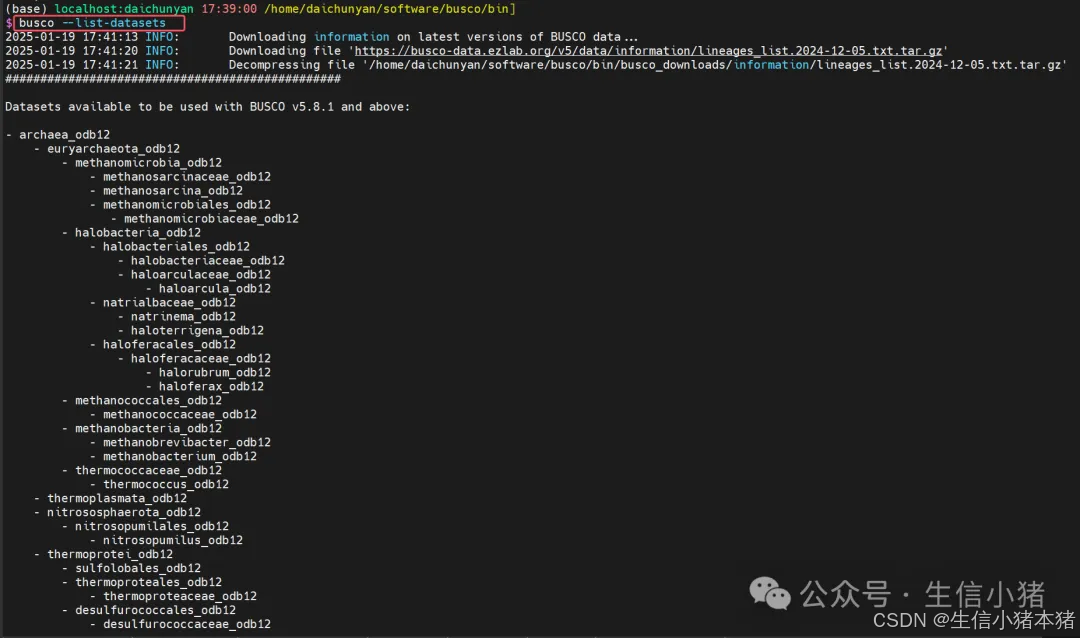
BUSCO 数据库包含了多个层级的数据库,涵盖了不同的物种分类。比如,细菌类有多个细分数据库,接着是古菌和真核生物的数据库。在真核生物中,又细分为真菌、动物、植物等子类。此外,还有病毒相关的数据库。可以根据自己的物种所属的分类选择合适的数据库进行分析。
去这里下载自己物种所属的数据库
下载地址:https://busco-data.ezlab.org/v5/data/lineages/
比如这里我的物种是拟南芥,属于芸薹属,所以我这里下载https://busco-data.ezlab.org/v5/data/lineages/brassicales_odb10.2024-01-08.tar.gz数据库

放到服务器上解压

记住brassicales_odb10文件夹路径,下面运行软时会用到。我这里是/home/daichunyan/test2/test19/buscoDB/brassicales_odb10
四、运行busco
busco 做评估的时候有两种方式,一种是联网运行,另外一种是本地准备好数据库,本地运行。
1、本地运行命令
busco -i genome.fa \
-o brassicales_odb10 \
-m geno \
-l /home/daichunyan/test2/test19/buscoDB/brassicales_odb10 \
-c 40 \
--offline1) -i genome.fa:-i 指定输入文件,这里 genome.fa 是待评估的基因组序列文件,格式为 FASTA。
2) -o brassicales_odb10:-o 指定输出文件夹的前缀,所有分析结果将保存在以 brassicales_odb10 命名的文件夹中。
3) -m geno:指定分析的模式。在此命令中,geno 表示执行 基因组模式(Genome mode)分析,适用于基因组装配数据(DNA)。其他模式包括 tran(转录组模式)和 prot(蛋白质模式)。
4) -l /home/daichunyan/test2/test19/buscoDB/brassicales_odb10:-l 指定使用的BUSCO数据库。在这个命令中使用的是 brassicales_odb10 数据库。这个数据库包含了针对 十字花科植物(Brassicaceae)的一组标志性基因集。
注:因为我这里组装的是拟南芥的基因组,所以这里选择了brassicales_odb10,如果自己分析的是别的物种,这块要进行相应的修改。这块要使用busco --list-datasets 命令去查一下自己物种应该分类于哪个数据库。
5) -c 40:指定使用的线程数。
6)-offline:不联网,用本地数据库去运行

虽然结果有busco_downloads文件夹,但是这个文件夹为空。
2、联网运行命令
busco -i genome.fa \
-o brassicales_odb10 \
-m geno \
-l brassicales_odb10 \
-c 10联网运行比本地运行命令只是少了一个 --offline 参数。还有 -l 参数不需要指定本地下载好的数据库,只需要提供数据库的名字,软件就会自动下载,使用这个数据库了。
联网先把要用的数据库下载下来,直接去运行BUSCO评估。因为BUSCO数据库是国外的网站,国内下载会比较慢。所以建议可以先把数据库下载下来,直接使用本地运行命令,运行速度会好点。

结果有busco_downloads文件夹,这个文件夹里有下载的数据库

五、查看结果

看一下本地运行命令和联网运行命令得到的结果是一致的

查看short_summary.specific.brassicales_odb10.brassicales_odb10.txt文件。
C: 99.2%:表示在总 BUSCO 基因组中的完整 BUSCO 基因数所占比例是 99.2%。该值表示输入的基因组序列中有 99.2% 的基因是完整的。
S: 97.9%:其中 97.9% 的 BUSCO 基因是单拷贝基因(single-copy),没有重复,基因组序列中仅存在一次。
D: 1.3%:其中 1.3% 的 BUSCO 基因是重复基因(duplicated),在基因组序列中有多个拷贝。
F: 0.1%:表示有 0.1% 的 BUSCO 基因是碎片化的(fragmented),部分基因不完整。
M: 0.7%:表示有 0.7% 的 BUSCO 基因缺失(missing),基因组序列中未找到这些基因。
n: 4596:表示使用的 BUSCO 基因组数据库中的 BUSCO 基因总数是 4596 个。
4560 Complete BUSCOs (C):在基因组中检测到 4560 个完整的 BUSCO 基因。
4500 Complete and single-copy BUSCOs (S):在完整 BUSCO 基因中,4500 个是单拷贝的基因。
60 Complete and duplicated BUSCOs (D):在完整 BUSCO 基因中,有 60 个是重复的基因。
3 Fragmented BUSCOs (F):在基因组中检测到 3 个 BUSCO 基因是碎片化的,说明这些基因并不完整。
33 Missing BUSCOs (M):有 33 个 BUSCO 基因在基因组中未检测到。
该基因组有 99.2% 的基因完整(包含单拷贝和重复的基因),其中绝大部分(97.9%)为单拷贝基因。
仅有 0.7% 的基因缺失和 0.1% 的基因碎片化,表明基因组的完整性非常高。
说明该基因组的组装质量较好,适合进一步分析和注释。
结果解读部分摘抄自:https://mp.weixin.qq.com/s/GPxFoqQSVQ3iTrzdFju2Yw
六、评估结果可视化
这里可以使用BUSCO软件包自带的generate_plot.py去画图
mkdir BUSCO_summaries
cp ./*/short_summary.*.txt BUSCO_summaries/
python ~/software/busco/scripts/generate_plot.py -wd BUSCO_summaries/-wd BUSCO_summaries 去指定输出文件所在目录名称,它是用R去绘图。

绘制出的busco_figure.png图是下面这样的,即对short_summary.specific.brassicales_odb10.brassicales_odb10.txt文件上的内容进行一个展示

会把数据库里面的这些单拷贝同源基因,基于评估结果分了几类:
首先是有和无,红色的这部分就是Missing(M),在数据库是里面有的,但是在基因组上没有预测出来的这部分单拷贝直系同源基因。Missing(M)越多,表示基因组的组装情况不太好。
在数据库里面有,在基因组上也预测到的这些基因分成两类,一类是片段是完整的,一类是片段是不完整。不完整的片段(fragmented(F)越多,基因组组装的情况可能也越不好。
完整的这部分又会分成完整的单拷贝 (Complete(C) and single-copy(S))和完整的多拷贝 (Complete(C) and duplicated(D))。因为数据库收集的时候找的就是在不同的物种间都是单拷贝的基因,所以说在评估的物种里面应该也是单拷贝的状态才对。但它现在出现了多拷贝,大概率是基因组组装结果里面存在重复导致的。所以如果自己组装的基因组里面duplication比例很高,并且你这个物种没有很近期的全基组复制,大概率是自己基因组组装结果存在着冗余。























 1335
1335

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








